猪圆环病毒2型重组病毒分离株的鉴定
2014-06-17李海超韩凌霞
李海超,韩凌霞*
(1.东北农业大学动物医学院,黑龙江哈尔滨150030;2.中国农业科学院哈尔滨兽医研究所实验动物与比较医学团队 兽医生物技术国家重点实验室,黑龙江哈尔滨150001)
猪圆环病毒2型(Porcine circovirus virus type 2,PCV-2)是广泛分布于猪体内的一种病原体,与多种病原混合感染导致发病,统称为猪圆环病毒相关疾病(PCVAD)[1]。其主要症状之一的断奶仔猪多系统衰竭综合征(PMWS)对全球的养猪业造成了严重的经济损失[2],临床症状为消瘦、生长迟缓和淋巴结肿大[3]。其他临床症状还包括猪皮炎和肾病综合症[4]、猪呼吸综合征[5]和繁殖障碍[6]等。此外,PCV-2还与其他的疾病有关联,例如渗出性皮炎[7]和仔猪先天性震颤[8]等。
PCV-2属于圆环病毒科圆环病毒属,是无囊膜、单链环形DNA 病毒,基因组大小约为1.7kb 左右[3]。由11个开放阅读框(ORF)组成,其中ORF1编码复制相关的蛋白Rep和Rep′[9],ORF2编码衣壳蛋白Cap[10],ORF3编码的蛋白可能参与诱导病毒的凋亡[11]。
通常根据ORF2基因进行进化树分析,将PCV-2 分 为PCV-2a、PCV-2b 和PCV-2c3个基因型,PCV-2a有5个亚型2A、2B、2C、2D 和2E,PCV-2b有3个亚型1A、1B和1C[12-13]。PCV-2a和PCV-2b与猪圆环病毒疾病密切相关,2005 年以前流行的PCV-2毒株主要是PCV-2a,2005 年以后PCV-2b则成为优势流行毒株,并引起了更为严重的猪圆环病毒疾病[14]。PCV-2c基因型,仅存在于丹麦并且与猪圆环病毒疾病无关。尽管不同PCV-2 分离株的致病性存在差异,但是通过对GenBank上公布的PCV-2全基因组序列分析显示核酸序列同源性在93%以上[12]。虽然如此,点突变和重组仍可能是PCV-2进化的重要因素,导致了病毒基因组的遗传变异。Olvera等在2007年通过使用生物学软件对公布的PCV-2全基因组序列进行重组分析提出了PCV-2存在重组现象。也有一些田间试验支持PCV-2重组理论[15-16]。此外,也有猪群混合感染不同基因型PCV-2 的报道,是重组事件发生的前提[17]。我国关于PCV-2田间分离株亚型之间发生自然重组的报道还很少。本研究对来自临床疑似PMWS症状的猪病料进行病毒分离鉴定及全基因组克隆与测序,目的在于分析PCV-2毒株之间的遗传关系,为今后深入研究该病的分子生物学特性及开发有效疫苗提供科学依据。
1 材料与方法
1.1 材料
1.1.1 病料 2013年采自黑龙江省某猪场临床疑似PMWS患病猪的脾脏、肺脏、心脏和淋巴结等样品共6份。病料匀浆后分装在EP 管内,置-80℃保存备用。
1.1.2 试剂 LA-TaqDNA 聚合酶、pMD18-T 载体和DNA Marker DL 2 000均为宝生物(大连)工程有限公司产品;DNAzol Reagent为Invitrogen公司产品;胶回收试剂盒和质粒提取试剂盒为Axygen公司产品;其他试剂均为国产分析纯。
1.2 方法
1.2.1 病毒总DNA 的提取 按照DNAzol Reagent提取试剂盒的说明书操作,首先取750μL DNAzol Reagent与250μL 组织匀浆液混匀,室温静止5min。4 ℃、12 000r/min离心10min。取上清加到一个新的EP管中,加500μL 无水乙醇室温静置5min,4 ℃、12 000r/min 离心10 min,弃上清。加1 mL 75 0 mL/L 乙醇,4 ℃、12 000r/min离心5min弃掉液体。用20μL无DNA 酶和RNA酶的焦碳酸二乙酯(DEPC)处理水溶解含有病毒核酸的总DNA 到中,置-20 ℃储存备用。
1.2.2 引物设计 参考GenBank上公布的PCV-2的序列(AF055394,属PCV-2b基因型),使用OLIGO6和PRIMER 5.0软件设计引物,P1和P2扩增PCV-2ORF2部分基因,用于检测样品中的PCV-2,并且可区分PCV-1 和PCV-2;P3 和P4用来扩增PCV-2全基因(表1)。
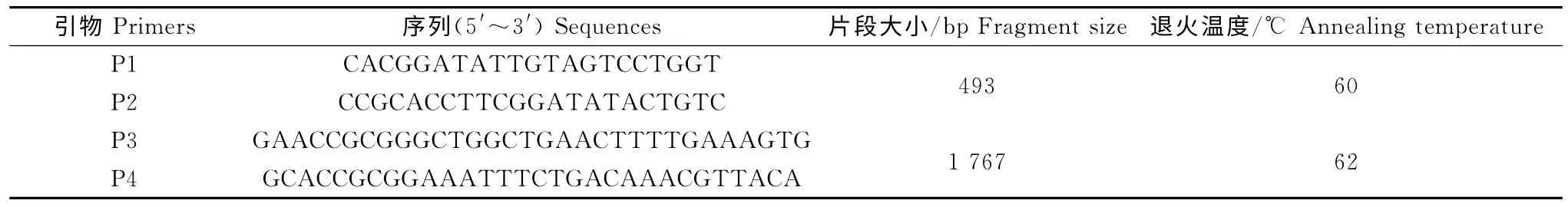
表1 引物信息Table 1 Information of primers
1.2.3 PCR检测 以提取的总DNA 为模板,利用引物P1和P2进行PCR 扩增。PCR反应体系为25 μL,反应条件为:95 ℃5min;94 ℃30s,60 ℃30 s,72 ℃30s,35个循环;72℃延伸10min。退火温度按表1进行。扩增产物用10g/L 的琼脂糖进行电泳检测。
1.2.4 PCV-2全基因组的扩增与克隆 使用引物P3和P4从PCV-2核酸鉴定是阳性的病料中扩增PCV-2全基因组,PCR 扩增体系为50μL:LA-TaqDNA 聚合酶0.5μL,dNTP 8μL,10×LA buffer 5 μL,上、下游引物(1 0pmol/μL)各2μL 的,DNA模板2μL,加去离子水至50μL。PCR 程序为:95℃5min;94℃30s,62℃30s,72℃70s,35个循环;72 ℃延伸10min。在10g/L 琼脂糖凝胶上电泳检测PCR 产物。用胶回收试剂盒(Axygen公司)按照说明书回收目的产物。按照TaKaRa公司的pMD18-T 载体说明书,将目的片段与pMD18-T 载体按照适当浓度的比例于16 ℃连接过夜。转化大肠埃希菌DH 5α感受态细胞,经IPTG/X-Gal诱导后挑取白斑,通过菌液PCR鉴定,将正确的阳性质粒送至华大基因科技股份有限公司进行序列测定。
1.2.5 PCV-2 分离株序列及毒力的分析 利用MEGA5.03软件和DNA Star软件,从提交到Gen-Bank中的PCV-2 全基因序列中选取PCV-2a2A、PCV-2a2B、PCV-2a2C、PCV-2a2D、PCV-2a2E、PCV-2b1A、PCV-2b1B和PCV-2b1C八个亚型的参考毒株(每个亚型选取2 株,登录号见图1),利用DNA Star软件中的MegAlign方法与1.2.4中的6株PCV-2全基因组进行遗传进化分析,生成系统进化树,并确定基因亚型。同时根据宋勤叶等[18]的报道,根据ORF2 76位和131位点的氨基酸残基初步分析PCV-2分离株的毒力。
1.2.6 PCV-2 分离株的重组分析 使用RDP4(recombination detection program,RDP)软件中的RDP、GENECONV、BootScan、Maxchi、Chimaera、Siscan和3Seq算法对6株PCV-2分离株序列和参考序列进行重组预测。用重组RDP4 软件检测PCV-2分离株是否存在重组位点,并确定可能的亲本序列。重组检测方法的常规设置为:window 大小=30,P=0.01 以及Bonferroni多重比较校正。最后用SIMPLOT 3.5.1软件对RDP 软件预测的结果进行验证。
2 结 果
2.1 病料PCR检测结果
本研究中采集的6 份病料均可以扩出493bp大小的目的条带(图1)。
2.2 PCV-2全基因组的扩增及测序
从PCV-2 核酸阳性的病料组织DNA 中经PCR 扩增出特异性的目的条带,其大小与预期值相符合(图2)。经胶回收后将目的片段与pMD-18T载体相连,对筛选出的阳性重组质粒进行测序。测序结果显示这6株分离株的基因组大小均为1 767 bp。Blast比对结果显示这6株分离株与NCBI上提交的PCV-2全基因组的核苷酸同源性为94%~100%,而与PCV-1 全基因组核苷酸的同源性在75.6%~76.6%之间,表明分离到的6株分离株为PCV-2。
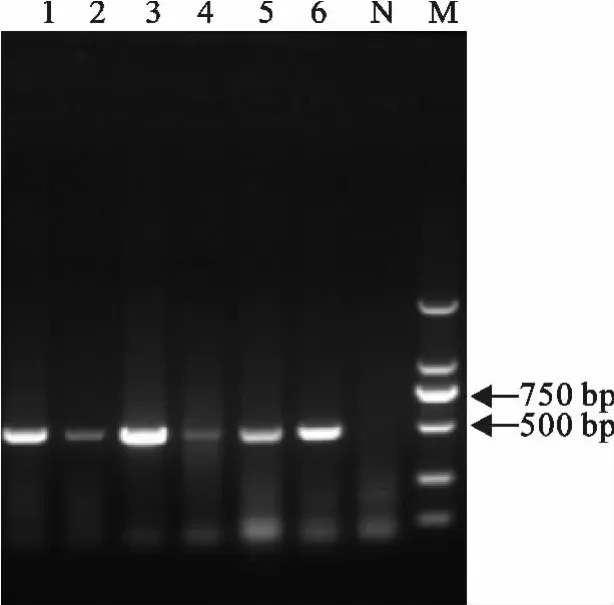
图1 病料PCR 扩增结果Fig.1 PCR amplification results of samples
2.3 分离株与参考株的核酸序列的同源性比较和系统进化分析
MegAlign同源性比较结果表明,分离株与参考株的核酸序列同源性为94.5%~99.9%。6个分离株之间核酸同源性为94.6% ~99.9%。用MEGA5.03软件中的N~J法系统进化树分析结果显示,6株分离毒中LNF3、HEBF5 和HEN65 为PCV-2b1C,LN51为PCV-2b1B、HEN60 株 为PCV-2b1A,HEBF7 属 于PCV-2a,但不属于任何PCV-2a亚型(图3)。
2.4 分离毒株的ORF2序列特征及毒力分析
通过MEGA5.03软件分析,6株分离株的ORF1在氨基酸水平上高度保守。3 株PCV-2b1C亚型和1株PCV-2a型的ORF2长702bp,编码的Cap蛋白为233 个氨基酸,另外2 个亚型毒株的ORF2长705bp,这个突变导致了1个氨基酸的延长,这与参考序列相符合。用DNA Star软件对ORF2推导氨基酸同源性分析,分离株与参考株之间的氨基酸同源性为88.1%~99.6%,5 株PCV-2b型分离株之间的氨基酸的同源性为93.2%~99.6%;与PCV-2b 和PCV-2a亚型参考序列的氨基酸的同源性分别为93.2%~99.1%和87.7%~93.2%。而HEBF7的氨基酸与其他5株分离株的同源性为88.1%~90.6%,与PCV-2b和PCV-2a亚型的氨基酸同源性分别为90.1%~91.0%和92.8%~94.9%。LNF3、HEBF7、HEN65、LN51和HEN60的ORF2的76位点为I,131位点为T,根据PCV-2强、弱毒株氨基酸位点差异[17],初步判定LNF3、HEBF7、HEN65、LN51 和HEN60 均为强毒。但是HEBF7在76位点为I,而131位点为P,根据上述的强、弱毒的判断标准不能进行判断。
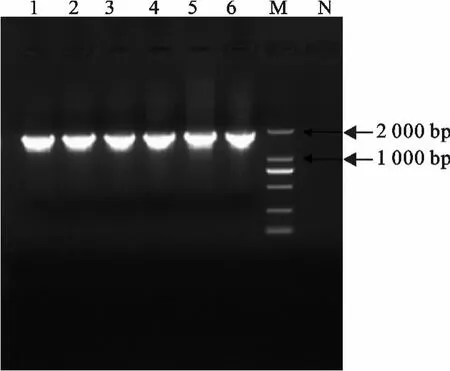
图2 PCV-2分离株全基因组扩增结果Fig.2 Amplification results of PCV-2isolate genome
2.5 分离株的重组分析
用RDP4.2.4 软件中的RDP、GENECONV、BootScan、Maxchi、Chimaera、Siscan和3Seq算法对本次分离到的6株PCV-2分离株和参考序列进行重组预测,算法/P 值分别为RDP/9.646×10-5、GENECONV/7.832×10-3、BootScan/3.484×10-5、Maxchi/9.196×10-6、Chimaera/8.816×10-5、Siscan/5.405×10-4和3Seq/1.534×10-7仅仅检测到LN51发生重组。与主要亲本PCV-2b1A(GenBank登录号DQ2207301株)和次要亲本PCV-2a2A(GenBank登录号AF117753)的比例概率超过99%,暗示可能是PCV-2a和PCV-2b基因型发生遗传交换,预测其重组起始和终止位点分别为1 765nt和508nt(图4)。
以DQ220730-PCV-2b1A 和AF117753-PCV-2a2A 为亲本,用SIMPLOT 3.5.1软件对LN51进行验证,结果显示与RDP 软件分析的结果类似(图4)。LN51在rep 基因区域与PCV-2a2A 在核酸水平上高度同源,与PCV-2b1A 在cap基因区域的核酸高度同源。Bootscan分析(图5)表明交叉区域位于rep 基因区域内,与RDP4.2.4软件分析结果相似。
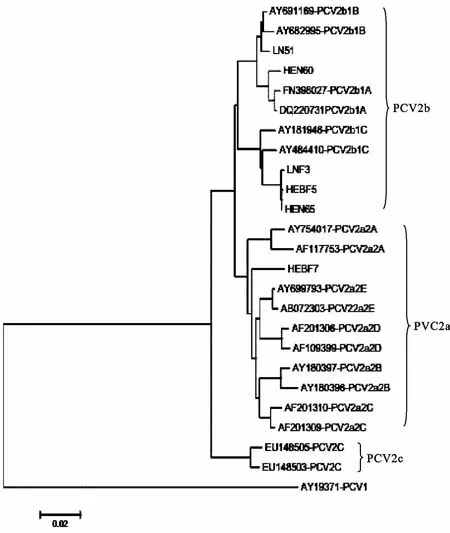
图3 6株分离株与部分参考序列全基因组聚类分析Fig.3 The genome sequence cluster analysis of 6isolates and reference strains
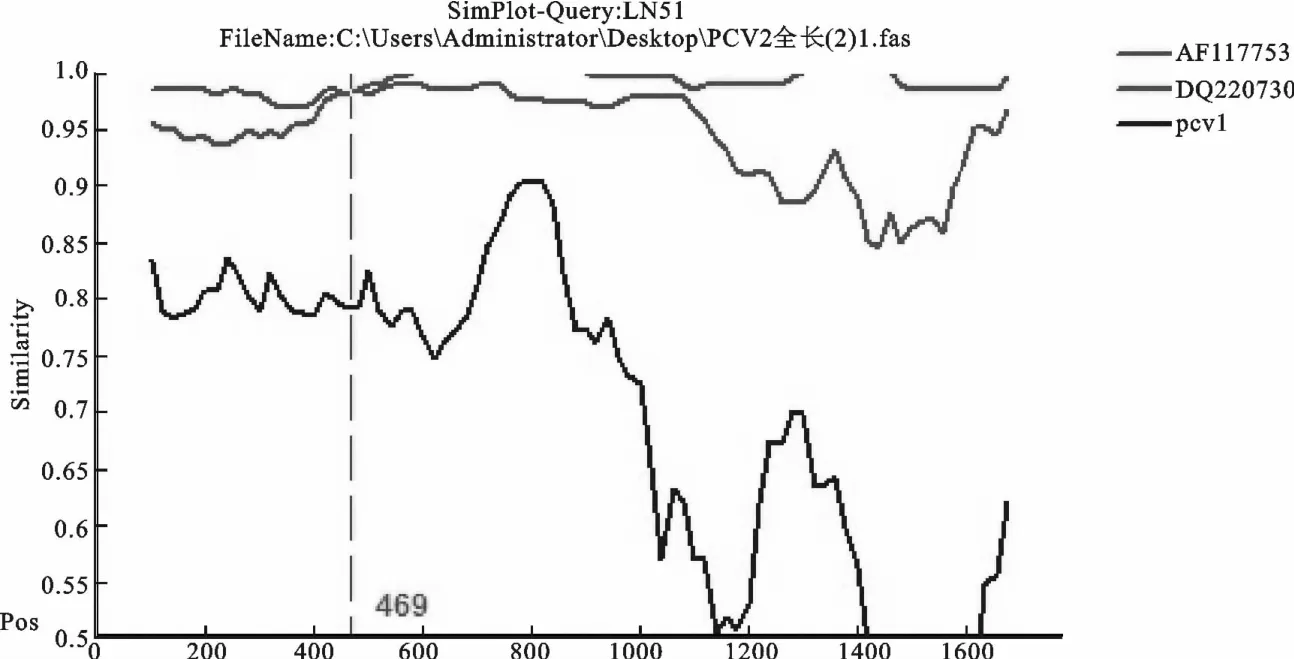
图4 重组序列的同源性分析Fig.4 Similarity analysis of recombinant sequences
3 讨 论
PCV-2主要损伤免疫器官造成免疫抑制,是导致其他疾病混合感染的主要因素,因此对PCV-2的基因型、毒力和病毒重组等的鉴定能对PCV-2的致病机理和疫苗研制提供重要信息。有资料表明,PCV-2的突变率比任何DNA 病毒的突变率都高,与多数RNA 病毒的突变率相似,是导致不同基因型出现的原因之一[19]。目前公认PCV-2 有PCV-2a、PCV-2b和PCV-2c3个基因型,其中PCV-2a和PCV-2b分别有5 个(2A-2E)和3 个(1A-1C)亚型[20]。PCV-2a和PCV-2b是猪圆环相关疾病的主要病原体,也与本研究从现地病料中得到的PCV-2基因型相符(5株PCV-2b,1 株PCV-2a)。HEBF7与PCV-2a和PCV-2b亚型ORF2的氨基酸同源性均在90%以上,依据PCV-2强、弱毒株氨基酸位点差异原则,在ORF2的76位和131同时具有强毒和弱毒的特征,可能进化上正处于PCV-2a向PCV-2b的过渡。
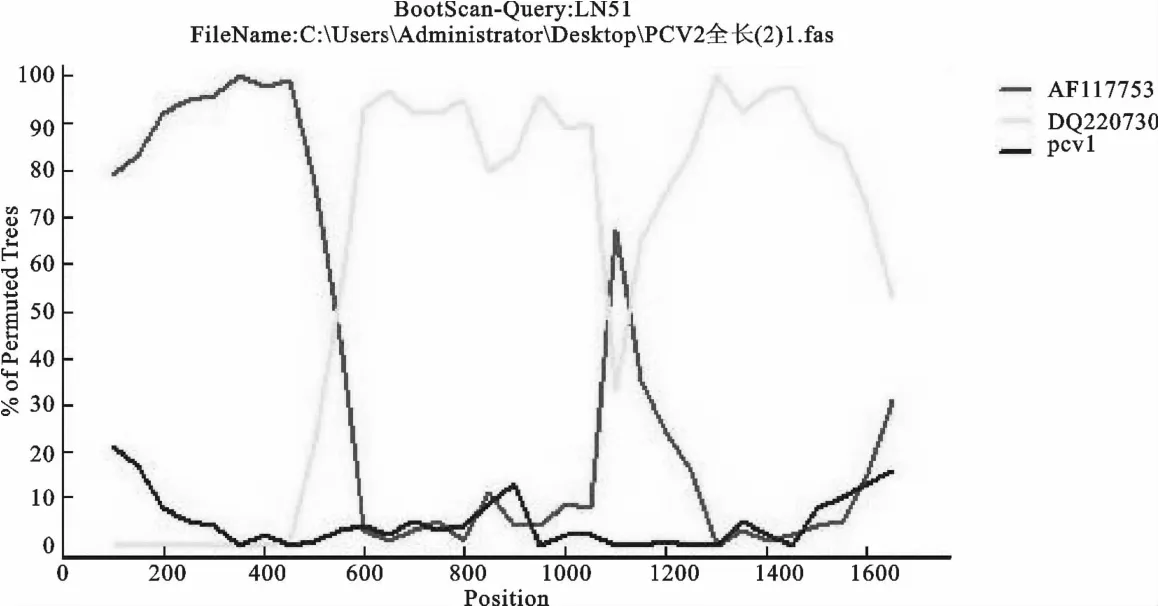
图5 重组序列的Bootscan分析Fig.5 Analysis of recombinant sequences by Bootscan
重组对于病毒的进化非常重要,是PCV-2遗传多样性产生的原因之一[12]。本研究分析结果表明LN51分离株可能是PCV-2b和PCV-2a基因型之间出现的重组,且2b1A 和2a2A 基因型毒株分别为主要亲本和次要亲本,与之前的研究相一致。事实上,根据Olvera A 等[12]的研究,这两个基因亚型是最常见的PCV-2重组亲本。
Ramos N 等[21-23]根据重组理论对GenBank 收录的PCV-2序列进行重组分析,认为rep 基因区域是最佳重组区域—最佳重组起始位点在rep基因的5′端42nt~89nt,最佳结束位点位于rep 基因的389nt和429nt(对应于rep′的转录剪切位点),也有自然条件下的重组证据。然而,也有很多试验提出在ORF2区域各基因型之间和内部也能出现重组现象[24]。本研究预测LN51 株是来自ORF1 基因1 765nt~508nt之间的重组,意味着PCV-2重组的区域可能更广泛。另外,不同基因型PCV-2 在ORF1和ORF2 之间重组能显著提高病毒复制能力,改变抗原表位[25]。
研究PCV-2 毒株间重组、重组发生的高频区域,以及重组对于病毒抗原性和致病性的潜在影响,对于阐明PCV-2相关疾病的分子致病机理、生物学特性以及开发疫苗都有重要的指导意义。
[1]Opriessnig t,Meng X J,Halbur P G.Porcine circovirus type 2 associated disease:update on current terminology,clinical manifestations,pathogenesis,diagnosis,and intervention strategies[J].Vet Diagn Invest,2007,19(6):591-615.
[2]Rose N,Opriessnig T,Grasland B,et al.Epidemiology and transmission of porcine circovirus type 2(PCV-2)[J].Virus Res,2012,164(1-2):78-89.
[3]Ge X,Wang F,Guo X,et al,Porcine circovirus type 2and its associated diseases in China[J].Virus Res,2012,164(1-2):100-106.
[4]Rosell C,Segalés J,Ramos Vara J,et al.Identification of porcine circovirus in tissues of pigs with porcine dermatitis and nephropathy syndrome[J].Vet Rec,2000,146(3):40-43.
[5]汤 洁,虞飞龙.猪繁殖与呼吸综合征与猪圆环病毒病混合感染的诊断与防治方法探析[J].科学时代,2013,16(2):82-85.
[6]West K H,Bystrom J M,Wojnarowicz C,et al.Myocarditis and abortion associated with intrauterine infection of sows with porcine circovirus 2[J].J Vet Diagn Invest,1999,11(6):530-532.
[7]Kim J,Chae C.Concurrent presence of porcine circovirus type 2 and porcine parvovirus in retrospective cases of exudative epidermitis in pigs[J].Vet J,2004,167(1):104-106.
[8]Stevenson G W,Kiupel M,Mittal S K,et al.Tissue distribution and genetic typing of porcine circovirus in pigs with naturally occurring congenital tremors[J].Vet Diagn Invest,2001,13(1):57-62.
[9]Mankertz A,Hillenbrand B.Replication of porcine circovirus type 1requires two proteins encoded by the viral rep gene[J].Virology,2001,279(2):429-438.
[10]Nawagitgul P,Morozov I,Bolin S R,et al.Open reading frame 2of porcine circovirus type 2encodes a major capsid protein[J].Gen Virol,2000,81(Pt 9):2281-2287.
[11]Liu J,Chen I,Kwang J.Characterization of a previously unidentified viral protein in porcine circovirus type 2-infected cells and its role in virus induced apoptosis.[J].Virology,2005,79(13):8262-8274.
[12]Olvera A,Cortey M,Segalés J.Molecular evolution of porcine circovirus type 2genomes:phylogeny and clonality[J].Virology,2007,357(2):175-185.
[13]Segalés J,Olvera A,Grau-Roma L,et al.PCV-2genotype definition and nomenclature[J].Vet Rec,2008,162(26):867-868.
[14]Wiederkehr D D,Sydler T,Buergi E,et al.A new emerging genotype subgroup within PCV-2b dominates the PMWS epizooty in Switzerland[J].Vet Microbiol,2009,136(1-2):27-35.
[15]Cadar D,Cságola A,Lorincz M,et al.Detection of natural in-ter-and intra-genotype recombination events revealed by cap gene analysis and decreasing prevalence of PCV-2in wild boars[J].Infect Genet Evol,2012,12:420-427.
[16]Ca L,Ni J,Xia Y,et al.Identification of an emerging recombinant cluster in porcine circovirus type 2[J].Virus Res,2012,165:95-102.
[17]Zhai S L,Chen S N,Wei Z Z.et al.Co-existence of multiple strains of porcine circovirus type 2in the same pig from China[J].Virol J,2011,13(8):517.
[18]宋勤叶,马增军,李潭清.猪圆环病毒2型河北分离株的基因型鉴定及其毒力分析[J].中国兽医杂志,2008,44(9):6-9.
[19]Duffy S,Shackelton L A,Holme E C.Rates of evolutionary change in viruses:patterns and determinants[J].Nat Rev Genet,2008,9(4):267-276.
[20]Segals J,Olvera A.PCV-2genotype definition and nomenclature[J].Vet Rec,2008,162(26):867-868.
[21]Ramos N,Mirazo S,Castro G,et al.Molecular analysis of porcine circovirus Type 2strains from Uruguay:evidence for natural occurringrecombination[J].Infect Genet Evol,2013,19:23-31.
[22]Kim H K,Luo Y,Moon H J,et al.Phylogenetic and recombination analysis of genomic sequences of PCV-2isolated in Korea[J].Virus Genes,2009,39(3):352-358.
[23]Ma C M,Hon C C,Lam T Y,et al.Evidence for recombination in natural populations of porcine circovirus type 2in Hong Kong and mainland China[J].J Gen Virol,2007,88(t6):1733-1737.
[24]Cai L,Ni J,Xia Y,et al.Identification of an emerging recombinant cluster in porcine circovirus type 2[J].Virus Res,2012,165(1):95-102.
[25]Guo L J,Lu Y H,Huang L P,et al.First construction of infectious clone for newly emerging mutation porcine circovirus type 2 (PCV-2)followed by comparison with PCV-2aand PCV-2bgenotypes in biological characteristics in vitro[J].Virology,2011,419:57-63.
