利用非培养技术初步研究古井贡酒窖泥细菌群落结构
2014-05-17张会敏李天婵孙美青周庆伍李安军何宏魁张治洲
张会敏,李天婵,孙美青,周庆伍,李安军,何宏魁,张治洲,,*
(1.哈尔滨工业大学(威海)海洋科学与技术学院,山东威海264209;2.安徽古井贡酒股份有限公司,安徽亳州236826)
中国许多局部地区的土壤、空气及水环境污染已经比较严重。许多酿造企业(白酒、酱油等)依赖特定的天然微生物群落,而环境恶化非常有可能逐步改变微生物群落的结构,继而影响企业产品质量的稳定性。另外,基于天然微生物群落发酵的酿造企业新建生产基地在目前的技术水平下是不能远迁的,其原因就是它不能离开特定的水土所孕育的微生物群落结构,就近扩大生产基地并尽快投产一般也需要成熟的微生物群落技术来相助。目前国内较大河流之数目已经减半,短时间内非常容易发生大面积或局部地区的气候失调。可以肯定的是,很多企业将陆续遇到不同程度的酿造过程发生异常的问题,继而可能带来产品质量的波动或成本提高。为了应对上述挑战,研究人员需要研发能够快速定量检测微生物群落结构的生物技术,以便对发酵过程从分子水平上进行实时跟踪测定,继而研制群落结构调制技术。为此,需要首先对发酵相关微生物群落结构进行分析鉴定。
研究人员已对若干传统白酒酿造的窖泥微生物群落结构进行了非常有效的分子鉴定和初步的群落调制[1-13]。比如熊小毛等[7-8]采用 DGGE 技术,对白云边酒酿酒工艺过程中的大曲、堆积发酵产物、酒醅和窖泥,以及酱香型和浓香型酒相应工艺中的原核微生物和真菌进行了分子水平分析,发现白云边酒整个工艺过程中的原核微生物种类和酱香型酒相近,但窖底泥的原核微生物种类比酱香型多,而真菌数量比较接近;本研究通过构建16S rDNA文库和DNA测序对古井贡酒池壁窖泥和池底窖泥中所含的细菌微生物群落结构组成进行了初步解析。
1 材料与方法
1.1 材料与仪器
古井窖泥样品2012年11月18日采于古井贡酒股份有限公司西厂和四分厂的八个样品S10-S17,见表1。所取样品密封后于-80℃度冷藏备用。土壤基因组DNA提取试剂盒Solarbio D2600 北京索莱宝科技有限公司;NPK02 PCR试剂盒 购自GREDBIO,山东;胶回收试剂盒SK8132 购自上海生工;TA克隆试剂盒D102A及大肠杆菌感受态细胞DH5α(Code No.9027)购自大连宝生物;LB培养基成分、分子量标记DL2000等其他生化试剂 均购自上海生工。
普通PCR仪TP600 TaKaRa;生化恒温培养箱SPX-250B-Z 博讯;震荡培养箱THZ-92A 博讯;超净工作台SW-CJ-1F 博讯;普通离心机TGL-16GB 生工;凝胶成像分析仪WD-9413C 六一。
1.2 实验方法
1.2.1 窖泥样品基因组总DNA提取 每种窖泥样品称取200mg,采用Solarbio试剂盒方法提取基因组,最后洗脱体积为50μL。基因组样品-80℃冷藏备用。
1.2.2 细菌16S rDNA的PCR扩增 扩增引物为27F(5'-AGAGTTTGATCCTGGCTCAG-3')和 1492R(5'-GGTTACCTTGTTACGACTT-3')。PCR 反应12μL 体系:6μL 2 × NPK02(GREDBIO)buffer,1μL(50ng)DNA 模板,0.8μL each primer(2μmol/L),0.2μL Taq DNA polymerase(5U/μL),4μL ddH2O.PCR反应程序:94℃预变性5min;94℃ 1min,55℃1min,72℃ 1min,38 个循环;72℃延伸2min。
1.2.3 细菌16S rDNA克隆文库的构建及阳性克隆的筛选 PCR产物经1.2% 琼脂糖凝胶电泳,将位于1500bp左右的条带切胶纯化。按说明书将纯化片段与载体pMD19-T连接进行TA克隆。连接产物转化大肠杆菌感受态细胞DH5α,涂布于含氨苄(100μg/mL)LB板上37℃过夜培养,进行蓝白斑筛选。每个LB板预先涂有 40μL X-gal(20mg/mL)和 70μL IPTG(20mg/mL)。每种窖泥样品从板上随机挑取80~120个白斑克隆,进行转板备份培养并进行测序。
1.2.4 克隆测序及序列分析 利用PCR引物27F和1492R进行 DNA双向测序(上海生工)。使用CExpress软件完成双向序列拼接。拼接所得序列由EditSeq(Lasergene,DNASTAR,Madison,WI,USA)软件编辑,并由 Seqman(Lasergene)软件去掉来自于pMD19-T载体的多余序列。再经过NCBI的BLAST进行比对,确定与已知序列的同源关系。根据BLAST结果,下载(http://www.straininfo.net/)标准菌的16S rDNA序列,然后每个OTU选用1个代表序列,采用Mega 5软件建立neighbour-joining(Bootstrap method with a 1000 replicates)系统发育树。
1.2.5 克隆文库评价 用C值和稀释曲线对克隆文库的细菌多样性进行分析和评价,C值代表克隆文库中所包含的微生物的种类占样品中全部微生物种类的比例,反映了克隆文库对样品群落多样性的代表程度,数值越大,代表性越强[13]。计算公式为:C(%)=(1-n1/N)×100计算,N代表16S rDNA克隆文库的库容,n1为文库中仅出现过1次的OTU的数量。稀释曲线(rarefactioncurve)是通过aRarefactWin软件,用已知的各种OTU的相对比例来推算抽取n个克隆时出现OTU数量的期望值,然后根据一组n值与其相对应的OTU数量的期望值所做出的曲线,当曲线趋于平缓或达到平台期时可以认为库容已经足够。
2 结果与分析
2.1 窖泥基因组总DNA提取和PCR扩增结果
利用土壤基因组DNA提取试剂盒提取样品基因组总DNA。然后,以样品总DNA为模板,通用引物27F-1492R扩增包含16S rDNA所有V区序列。琼脂糖凝胶电泳显示PCR产物单一,大小约1500bp左右,见图1。

图1 古井窖泥细菌16S rDNA PCR产物电泳图Fig.1 Agarose gel separation of 16S rDNA PCR products of eight GuJingGong pit mud samples
2.2 细菌16S rDNA克隆文库的分析
古井窖泥的八个样本经过克隆测序所取得的阳性克隆数,OUT数,C值等参数见表2。各样本TA克隆文库的稀释曲线见图2。从S10随机挑取的120个克隆中获得112个阳性克隆,归为21个OTU,16S克隆文库的C值为91.07%;从S11随机挑取的120个克隆中获得116个阳性克隆,归为11个OTU,C值为97.41%;从S12随机挑取的120个克隆中获得113个阳性克隆,归为17个OTU,C值为91.05%;从S13随机挑取的120个克隆中获得118个阳性克隆,归为20个OTU,克隆文库的C值为92.37%;从S14随机挑取的80个克隆中获得64个阳性克隆,归为19个OTU,克隆文库的 C值为82.81%(略低);从S15随机挑取的120个克隆中获得92个阳性克隆,归为14个OTU,克隆文库的C值为92.39%;从S16随机挑取的120个克隆中获得109个阳性克隆,归为21个OTU,克隆文库的 C值为89.91%(略低);从S17随机挑取的80个克隆中获得69个阳性克隆,归为11个OTU,克隆文库的C值为89.86%(略低);稀释曲线见图2。尽管S14,S16和S17的C值偏低,相比于以前的研究[19],本研究的阳性克隆数目已扩大了很多倍。汤斌等[19]曾对古井窖泥细菌多样性进行过初步分析,但是他们当时仅探索了池底窖泥的30个阳性克隆。本次研究探索了池壁和池底的793个阳性克隆。结合C值和稀释曲线可知库容已基本满足要求,可以得到较为全面且可靠的群落信息。

表1 古井窖泥样本资料Table 1 GuJingGong pit mud samples
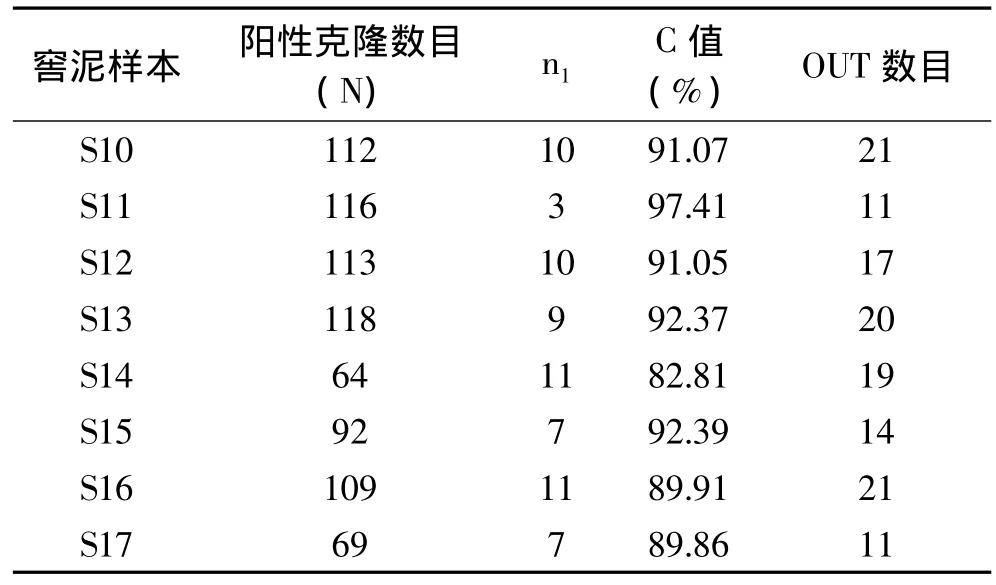
表2 古井窖泥样本细菌克隆文库分析参数表Table 2 Information of TA-cloning library for GuJingGong pit mud samples
2.3 细菌群落多样性分析
将各克隆文库的序列通过NCBI BLAST分析得到与NCBI数据库中相似度最高的菌种。详细统计信息如表3所示。
古井窖泥八个样本所得到的总共793个克隆,归为57个OTU,优势菌类为未知菌类(60.78%)和厚壁菌门(Firmicutes,31.16%)。另外变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、互养菌门(Synergistetes)、放线菌门(Actinobacteria)的奇异菌属(Atopobium)也有相应比例分布。
表4中列出了57个OTU的代表菌株和比对详细信息。结合表3和表4可以看出厚壁菌门对应的前26个OTU部分约有50%的代表菌株的相似度低于97%。而其余大类的代表菌株中,第55个、第33个和第45个OTU代表菌株相似度偏低外,其余代表菌株的相似度菌均大于97%。一般认为同源性小于97%极有可能是新种,所以对于这部分细菌的物种归属需要进一步确定。
表5显示了西厂和四分厂的细菌群落结果分布差异,池底泥和池壁泥的细菌群落结构差异。与西厂相比,四分厂的变形菌门细菌的含量似乎要明显丰富一些。与池壁泥相比,池底泥的细菌群落种类更丰富。
2.4 细菌16S rDNA系统发育分析
根据表3和表4中各OTU代表菌株blast结果的分类,将大致属于厚壁菌门的第1~27个OTU的代表序列和未知菌类的最后8个OTU的代表序列的系统发育分析图如图3所示。第28到57个OTU的代表序列的系统发育分析图如图4所示。

表4 古井贡酒窖泥细菌各OTU代表菌株详细信息Table 4 Representative strains of each bacterium OTU in GuJingGong pit mud samples
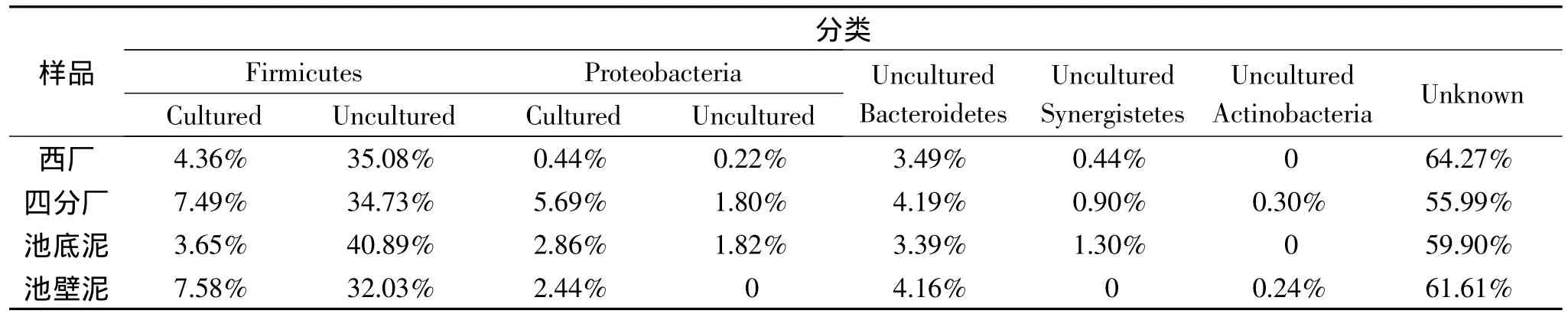
表5 古井贡酒窖泥细菌各类比例分布信息Table 5 Distribution of various types of bacteria in GuJingGong pit mud samples
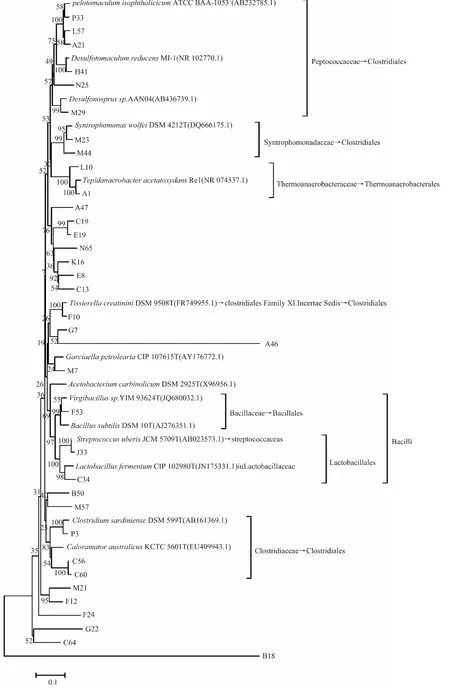
图3 根据代表序列构建的系统发育树。使用了35个代表性OTU序列(第1到27以及第50到57个OTU)和14个标准菌序列Fig.3 Construction of phylogenetic tree based on 16s rDNA sequences using representative OTUs(1 to 27,50 to 57)and 14 standard sequences
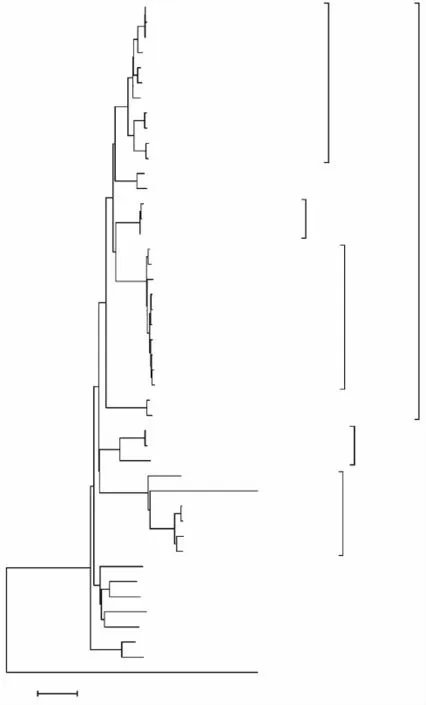
图4 根据代表序列构建的系统发育树。使用了30个代表性OTU序列(第28到57个OTU)和15个标准菌序列Fig.4 Construction of phylogenetic tree based on 16s rDNA sequences using 30 representative OTUs(28 to 57)and 15 standard sequences
从图3来看,可以明显看到B18代表序列起源最早,且其与其它代表菌株的遗传距离很远。从表4得知,B18比对相似度只有83%,可推测B18极有可能并不属于后壁菌门(Firmicutes)。图3中各序列聚类分布,依次属于后壁菌门(Firmicutes)的Peptococcaceae(Clostridiales),Syntrophomonadaceae(Clostridiales),Thermoanaerobacteraceae (Thermoanaerobacterales),Clostridiales FamilyⅪ.Incertae Sedis(Clostridiales),Bacillaceae(Bacillales,Bacilli),Lactobacillales(Bacilli),Clostridiaceae(Clostridiales)。中间部分,代表序列A47、C19、E19、N65、K16、E8、C13 没有与标准菌聚类,可推测它们属于后壁菌门(Firmicutes)的其余科属。最后部分的M21、F12、F24代表菌株与其余菌株遗传距离比较近,有可能属于后壁菌门,而未知菌类的G22和C64两者单成一支,并不属于后壁菌门。
从图4来看,A46代表序列起源最早,且其与其它代表菌株的遗传距离很远。从表4得知,A46序列在NCBI中找不到相似序列,结合图3可推测A46序列与后壁菌门有一定的亲缘关系。很可能为近代进化未鉴定菌类。图4中各序列聚类分布,依次属于变形菌门(Proteobacteria)的 Xanthomonadaceae、Pseudomonadaceae、Enterobacteriaceae、互 养 菌 门(Synergistetes)和拟杆菌门(Bacteroidetes)。
结合图3和图4,可确定8个未知菌代表的大致归属。G22明显属于拟杆菌门(Bacteroidetes);C64属于变形菌门(Proteobacteria)的Xanthomonadaceae;A21、P3、C60、E19、M57 属于后壁菌门(Firmicutes)。其中P3与标准菌Clostridium sardiniense DSM 599T有100%的相似度。A46显示出与后壁菌门有很大亲缘关系,由于进化比较晚,具体归属需要进一步鉴定。
3 结论
本研究利用16S rDNA克隆文库对古井贡酒窖泥微生物群落进行了较大规模的分子鉴定,发现在793个有效测序的阳性克隆中未知菌种占据过半(60.78%),已知菌类分别属于Firmicutes(31.16%)、Proteobacteria(3.55%)、Bacteroidetes(3.79%)、以及Synergistetes(0.63%)、Actinobacteria的 Atopobium(0.13%)。这些结果为进一步研制窖泥微生物群落组成的快速定量技术,继而为实时监测发酵过程提供服务奠定了基础。
目前所有已知的天然微生物群落都是比较复杂的,而且其中的绝大部分微生物都是人类所知甚少的物种。这说明在群落(而不是单个或少数微生物物种)水平上,微生物群落综合研究水平及其在食品工业中的应用在国际范围内都处于初级阶段。古井集团研究人员及合作者已经开展了若干研究[14-19],包括曾经对古井贡窖泥的微生物群落结构做过分析[19],得出了一些重要结论,而本研究是对以前工作的延续和补充。与已报道的其他类似研究一样,研究结果显示了窖泥微生物群落组成的复杂性,其中含量最高的一部分物种或种属除了部分16S rDNA序列外往往没有任何别的分子信息;在这种条件下建立快速实时测定群落结构的方法比较困难;而要建立这种复杂性与酿造风味及质量控制之间的因果关系,更是需要研究人员的长期努力。
[1]李家民,邹永芳,王海英,等.DGGE法初步解析沱牌酒厂窖底泥微生物群落结构[J].酿酒科技,2013,3:36-39.
[2]李恒,孙夏冰,陆震鸣,等.白酒窖池中不同部位窖泥挥发性化合物的差异性分析[J].食品与发酵工业,2013,39(3):158-162.
[3]王明跃,张文学,王海英,等.不同窖龄窖泥细菌的系统发育多样性分析[J].食品科学,2013,34(11):177-181.
[4]李文芳,王海英,张文学,等.川皖地区浓香型白酒窖底泥微生物菌群的多样性分析[J].酿酒科技,2013,1:23-26.
[5]刘琨毅,卢中明,郑佳,等.浓香型白酒窖泥微生物群落PLFA指纹图谱方法[J].应用与环境生物学报,2012,18(5):831~837.
[6]侯小歌,王俊英,李学思,等.浓香型白酒糟醅及窖泥产香功能菌的研究进展[J].微生物学通报,2013,40(7):1257-1265.
[7]熊小毛,杨团员,张明春,等.形成白云边酒风味特征的典型原核微生物菌系的研究[J].酿酒科技,2013,6:51-53.
[8]熊小毛,杨团员,张明春,等.形成白云边酒风味特征的典型真菌菌系的研究[J].酿酒,2013,40(2):25-28.
[9]姚万春,唐玉明,任道群,等.液体窖泥培养过程微生物和香气成份变化趋势[J].中国酿造,2013,32(2):45-48.
[10]张胜利,赵金松.优质窖泥微生物的测定研究[J].酿酒,2013,40(3):50-52.
[11]Jinsong Zhao,Jia Zheng,Rongqing Zhou,et al.Microbial community structure of pit mud in a Chinese strong aromatic liquor fermentation pit[J].Journal of the Institute of Brewing,2012,118:356-360.
[12]Bo Deng,Caihong Shen,Xiaohu Shan,et al.PCR- DGGE analysis on microbial communities in pit mud of cellars used for different periods of time[J].Journal of the Institute of Brewing,2012,118:120-126.
[13]姚粟,葛媛媛,李辉,等.利用非培养技术研究芝麻香型白酒高温大曲的细菌群落多样性[J].食品与发酵工业,2012,38(6):1-6.
[14]周庆伍,穆文彬,汤有宏,等.淡雅型古井贡酒生产工艺探讨[J].酿酒,2009,36(2):48-50.
[15]黄建梅,卢建春,李安军,等.采用气相色谱-质谱分析古井贡酒中的风味物质[J].酿酒科技,2006,7:91-94.
[16]郭文杰,卢建春.古井贡酒特征香味成分的研究[J].酿酒科技,2001,5:83-85.
[17]肖熙佩,张润清,陈华,等.古井贡酒大曲中发酵菌类—曲霉菌、根霉菌和酵母菌的初步研究[J].大连轻工业学院学报,1982,1:60-68.
[18]张会敏,束莹,李安军,等.利用非培养技术初步研究古井贡酒大曲细菌群落结构[J].现代食品科技,2014,30(4):44-49.
[19]汤斌,刘金英,周庆武,等.浓香型白酒窖泥中细菌多样性的免培养技术分析[J].食品与发酵工业,2012,38(7):43-47.
