基于特征耦合泛化的药名实体识别
2014-04-14何林娜杨志豪林鸿飞李彦鹏唐利娟
何林娜,杨志豪,林鸿飞,李彦鹏,唐利娟
(1.大连理工大学计算机科学与技术学院,辽宁大连116024;2.山东省农业管理干部学院机械电子工程系,山东济南250100)
1 引言
生物命名实体识别的直接目的是从文本集中识别出指定类型的名称,例如,蛋白质、基因、核糖核酸、脱氧核糖核酸、细胞、疾病、药物的名称等[1]。郑强等[2]对目前实体识别的研究情况进行了概述,其结论是近年来生物医学命名实体识别在语料库、特征选择、识别方法等方面取得了一定的进展,但由于生物医学领域的命名实体具有一些独有的特点,要使系统达到更高的性能仍面临着很大的挑战。生物命名实体识别的研究方法主要集中在以下几个方面:词典匹配方法[3]、基于规则的方法[4]、基于语言的方法[5]、基于主动学习的方法[6]以及基于机器学习的方法,如支持向量机(SVM)[7]、隐马尔可夫模型(HMM)[8]、最大熵[9]和条件随机场(CRF)[10]、分类器组合[11]以及词典和CRF相结合的半监督学习方法FCG[12]等。
目前制药工业正日益成为以知识为基础的行业,科学家们在发明药物的过程中需要获取相关的信息和知识。药物相关的研究呈爆炸式发展,远远超出了维护人员更新数据库的速度,这就迫切需要有一种自动提取药物信息的技术。Segura-Bedmar等[13]构建的药名识别系统可以同时对识别出的药名进行分类,该系统用MetaMap Transfer(MMTx)工具对文本中的信息进行抽取,将每个句子中的短语抽取出来并赋予相关的语义类型,然后将语义类型为“Pharmacological Substance”(PHSU)和“Antibiotics”(ANTB)的短语看作是药名,之后再用Stem方法(所有与药相关的词干均可从文献[14]中获取)对MMTx的结果进行补充和分类。徐博等[15]采用上下文模板抽取的方法构造了一个药名词典,然后用词特征的方法对药名词典进行去噪。本文中我们用同样的方法构造了一个药名词典,然后用半监督学习方法FCG[12]对药名词典进行去噪过滤,最后用去噪后的药名词典和CRF相结合的方法进行药名实体识别。
本文提出了一种词典查找和CRF相结合的实体识别方法。首先,我们利用上下文模板匹配的方法从外部资源PUBMED中进行药名提取,从而构成一个药名词典;然后再利用半监督学习方法——特征耦合泛化(FCG)对词典进行去噪;最后我们将去噪后的词典和CRF相结合在测试集上进行药名实体识别。
2 方法
我们的方法主要包括两个处理过程,一个是构造药名词典,另一个是用生成的药名词典结合CRF进行药名实体识别。
2.1 药名词典生成
由于已有的药名词典中包含的药名很少,而且药名词典不能得到及时的更新,所以必须对原有的词典进行扩充。本文中对于药名词典的生成,用的是上下文模板匹配方法[15]。此方法不依赖于已有的数据库,所以它识别出的实体名可以超出已有数据库,能够满足扩充词典的要求。然后我们用半监督学习方法FCG对扩充后的词典进行去噪,最后生成药名词典,方法流程如图1所示。
该流程图可以概括如下:
1.选取药名种子以及用于抽取模板和药名的语料。药名种子均选自Drugbank,我们把Drugbank中能下载下来的药名均作为种子。抽取模板和药名用的语料是1979年到2009年的PUBMED摘要;

图1 药名词典生成流程
2.从PUBMED语料中找到每个药名种子对应的上下文,组成上下文集合。
我们用Drugbank中的药名作为种子,从PUBMED摘要中抽取每个药名的上下文内容。本文在药名前后确定了窗口大小(窗口大小即为单词的数量),我们确定的药名前的窗口大小为3,药名后的窗口大小为2,并用-ENT-来代替药名。图2为本文方法抽取到的上下文的例子;

图2 由药名种子抽取到的上下文
3.根据上下文集合确定触发词,因为不同的上下文可能对应相同的触发词,所以每个触发词都对应一个上下文集合。
每个模板都由几个词组成,然而并非每个词的重要性都一样,图2所示例子中单词“treatment”的重要性就比其他的词大,因为这个词后面出现药名的概率比较大。我们把像“treatment”这样的词定义为触发词。触发词满足以下两个条件:(1)在上下文集合中出现的次数比较多;(2)在其周围可以确定药名;
4.根据每个触发词确定的上下文集合构建有向连通图。图3为所构建有向图的示意;
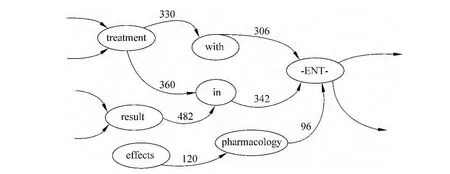
图3 根据选定的上下文模板构造的有向图
5.根据有向图中的权重进行剪枝,确定最后的上下文模板。本文中我们把权重小于150的分枝去掉,然后对模板进行排序,取排名前4 000的模板做为最终模板。
上面所述的步骤与徐博等[15]描述的基本相同,因此详细信息可参考文献[15]。
2.2 药名词典去噪
本文中我们把药名词典去噪看作分类问题,采用的是基于半监督学习的FCG方法,此方法由Li[12]提出,可以有效地解决数据稀疏的问题。
FCG算法的基本思想是将特征共现的信息转化为新的特征。特征共现要涉及两种特征,一种是低频特征,一种是区分性较强的类别特征。本文中将低频特征推广到任何特征,根据这两种类型特征在未标注语料(PUBMED摘要)中的共现情况我们得到新的特征。这些新的特征融合了未标注语料信息,所以弥补了有限训练集中信息量的不足。
在FCG方法中称第一类特征为实例识别特征(EDF),第二类特征为类别识别特征(CDF),而共现的值用特征耦合度(FCD)来表示。FCD的定义如下
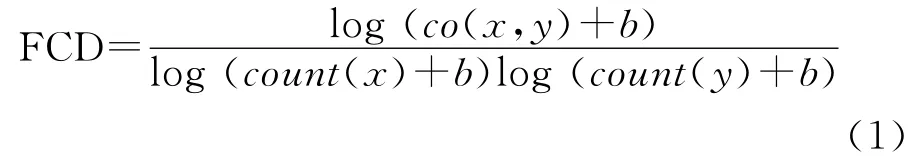
其中x代表EDF,y代表CDF,b在本文中设置为常量1。
因为EDF可以是任何特征,所以一个样本中可以含有很多EDF,每个EDF可以与多个CDF计算得到FCD。FCG方法的基本思路是:新特征是由EDF的上层“抽象概念”、CDF以及FCD类型组合而成,特征的值是当前实例中隶属于同一“抽象概念”的FCD的和。其中“抽象概念”我们用EDF root来表示。图4与图5是这几个概念的一个例子。

图4 EDF和EDF root的层次概念

图5 FCG方法中新特征的形成
本文中我们用的EDF root主要包含以下几种:{Name,Left-1,Left-2,Left-3,Right-1,Right-2,Right-3}其中Name是待去噪的药名,Left-n和Right-n分别表示该药名最左边和最右边的n个词,即边界n-gram。表1是EDF的一个实例。
本文中的CDF是根据卡方特征选择方法从EDF中选择出的,其中Left n-gram 300个,Right n-gram 300个。关于FCG算法的详细描述,以及EDF、CDF的选择可参考文献[12]。
最后我们将去噪后的词典和Drugbank中的词典融合在一起作为最终的药名词典。

表1 本文用到的EDF特征
2.3 药名实体识别
这里我们采用两种方法,并结合前面生成的药名词典进行命名实体识别。一种方法是词典匹配,所用到的算法是最大长度匹配,此方法简单快速;另一种方法是条件随机场,在条件随机场中主要用到了以下几种词特征:
1.词典前缀。以药名的前3~4个字母作为特征。
2.词典后缀。以药名的后3~4个字母作为特征。
3.以药名本身作为特征。
4.以词典查找结果作为特征(详细信息参考文献[12])
3 实验
3.1 实验数据
在药名词典生成中我们用Drugbank中的药名作为种子从PUBMED摘要中提取模板,用最终选出的模板再到PUBMED摘要中提取药名,从而生成初步的药名词典。然后用Drugbank中药名、疾病名和常用词构造一个训练集,通过FCG方法对生成的药名词典进行去噪。
在命名实体识别中用到的训练集语料是DDIE-xtraction 2011评测的训练集语料(http://labda.inf.uc3m.es/DDIExtraction2011/dataset.html)。该语料是用于提取药物与药物之间的关系的。语料是XML格式,每个句子中的药名已经识别出,所以我们可以将此语料作为训练集。实验一是用该评测的测试集语料作为药名实体识别的测试集,以检验生成的药名词典的性能;实验二用Segura-Bedmar等[13]用的语料(http://labda.inf.uc3m.es/DrugDDI/DrugNer.html)作为测试集,以便与其方法进行比较;实验三中我们将DDIExtraction 2011评测的训练集和测试集共同作为药名实体识别的训练集,将Segura-Bedmar等用的语料作为测试集,以检验在增加训练集的情况下我们方法的性能是否有所提高。
3.2 实验结果
表2给出了实验一的结果,其中CRF和词典相结合的方法(即表中“CRF+词典”项)是指将词典查找的结果作为特征加入到CRF中去。从表2中我们可以看到只用CRF的方法优于词典匹配的方法。因为词典匹配使用的算法是前向最大匹配和后向最大匹配,所以识别出来的很多实体都只是部分匹配。另外由于我们把词典中的每个实体都转化成了它的小写形式,这样就加入了一些噪音,例如,“THE”是一个药名的缩写,但是其小写形式“the”则只是英语中常用的定冠词。因此,词典匹配方法识别出的实体很多,但准确率和召回率却并不高。从表2我们看出CRF的效果优于词典匹配的方法,而CRF和词典相结合的方法又优于前两种方法,这充分说明词典在药名实体识别中具有一定的辅助作用,而且词典和CRF具有互补效果。

表2 本文中的方法在DDIExtraction 2011评测语料上的结果
表3显示了实验二和实验三的结果。此表中前3行是实验二的结果,该结果进一步验证了我们对上一个实验的分析。表3将我们的方法和Segura-Bedmar等的Stem方法进行了比较,所用的语料是Segurar-Bedma I.等在文献[13]用到的。这些方法的比较是在文献[13]中MMTx识别出的药名作为标准答案的基础上进行的。通过表3可以看出,我们所用的任何一个方法都比文献[13]中的Stem方法要好,这是因为Stem方法在一定程度上限制了药名的范围,有些药名并不包含给定的词干,对于这样的药名Stem方法就无法识别出。另外有些词包含给定的词干但它并不是药名,这样的词相对于前者比较少。
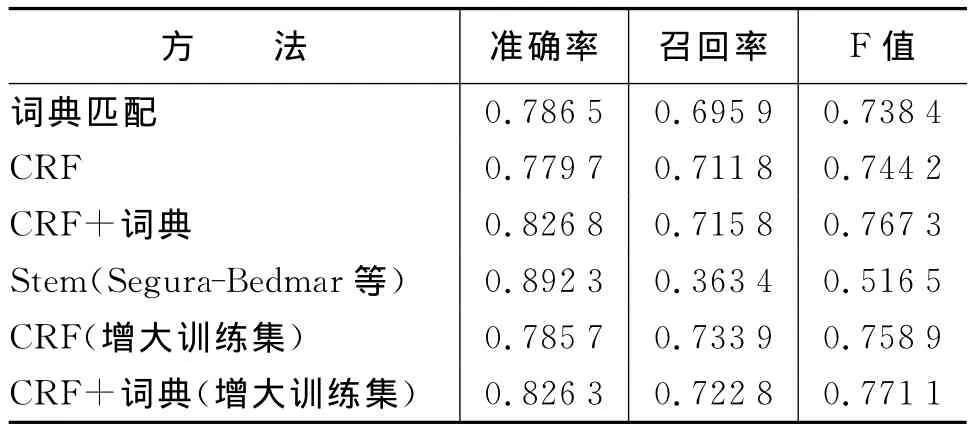
表3 本文中的方法在Segura-Bedmar等语料上的结果
除此之外,DDIExtraction 2011评测语料很有限,这也可能是影响性能的原因。于是我们做了第三个实验,将评测中的训练集和测试集一起作为药名实体识别的训练集。在第三个实验中,我们对CRF和CRF结合词典匹配两种方法进行了测试,结果如表3中的最后两行所示。从中我们可以看到,CRF和CRF结合词典匹配这两种方法在训练集增加的情况下,结果都有所提高,这证实了我们之前的推测。我们之所以没有在词典匹配方法上进行测试,是因为词典匹配方法用不到训练集,它只是以词典为基础在测试集上进行查找匹配。实际上这些方法的结果应该更好,因为训练集中存在一些错误,即有些非药名的词也被标注为药名,如“drug”、“n=7”等。然而,对比表2和表3我们可以发现测试集不同时性能差别比较大,这主要是因为表2的训练集和测试集均是DDIExtraction 2011评测语料,它们来都自于Drugbank数据库,有着相似的分布;而表3中我们用到的测试集是从PUBMED摘要中选出的,训练集和表2实验中的一样,分布情况差别较大,因而会在性能上存在差别。
Segura-Bedmar等用Stem方法除了识别MMTx识别出的药名外,还对MMTx未识别出来的药名进行了识别,本文中我们的方法也对MMTx未识别出的药名进行了识别。表4列出了我们的方法和Stem方法的比较。因为在通常情况下训练集是有限的,所以此实验中我们用的训练集是DDIE- xtraction 2011评测语料的训练集而没有加入评测的测试集。从表4中可以看出Stem方法在准确率上优于词典匹配方法和只用CRF的方法,因为Stem方法用的是词干,它使得两种药名之间的区别性更强。不过这两种方法识别出的药名总数多于Stem方法,因为我们构造的词典包含了很多数据库中所没有的药名,所以找出的药名数量比较大。由于CRF方法是根据词特征来进行识别的,它将具有某些特征的词均看作药名,因而识别出的药名数量更多。我们的另一种方法——CRF结合词典的方法不仅找到药名总数比Stem方法的多,而且在准确率上也比Stem方法好。然而它找出的药名总量少于CRF方法,这是因为在CRF的特征中加入了词典查找结果的特征,而此特征的加入增强了CRF的识别能力。对于数据库管理员来说,他们比较倾向于找到的药名多而准确率又高的方法,因此我们的方法比较有优势。
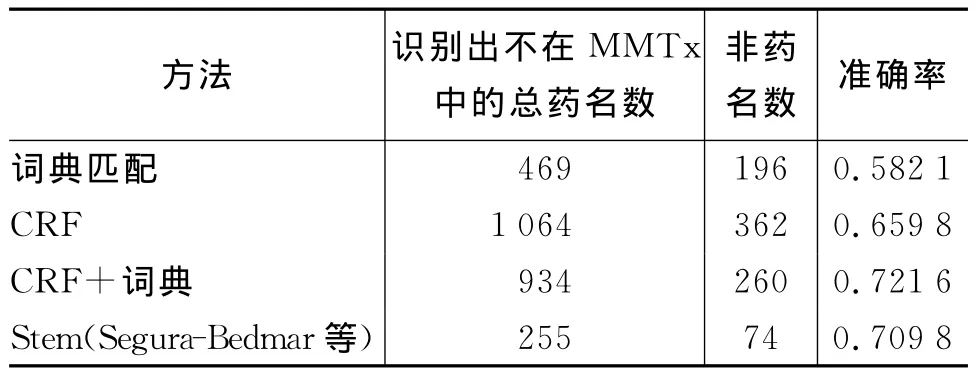
表4 MMTx之外的药名识别结果
4 总结和展望
药物相关研究不断出现,各种新药层出不穷,远远超出了维护人员对更新数据库的速度,这就迫切需要有一种自动提取药物信息的技术,本文提出一种基于特征耦合泛化的半监督学习技术,可以在很大程度上提高数据库维护人员手工检索的效率。通过上述实验的结果可以看出,用本文所描述的方法进行药名词典的生成,对药名实体识别有较好的效果。我们的方法除了识别出已有的药名外,还识别出了一些标准答案中所没有的药名,我们对其进行人工评测,准确率达到70%。
本文的工作还存在改进的地方,例如在词典去噪的时候,我们构造的训练集样本数量不足,加入更多的负例时词典的去噪结果会更好,在下一步工作中我们会尝试增加更多负例,例如,蛋白质名、基因名等,以进一步提高性能。
[1] 王浩畅,赵铁军,于浩.生物文本中蛋白质名称的识别[J].计算机应用,2007,24(1):100-102.
[2] 郑强,刘齐军,王正华,等.生物医学命名实体识别的研究与进展[J].计算机应用研究,2010,27(3):811-815.
[3] Tuason O,Chen L,Liu H,et al.Biological nomenclatures:A source of lexical knowledge and ambiguity[J].Pac Symp Biocomput,2004,9:238-249.
[4] Fukuda K,Tamura A,Tsunoda T,et al.Toward information extraction:Identifying protein names from biological papers[J].Pac Symp Biocomput,1998:707-718
[5] Sang EFTK,Meulder F D.Introduction to the CoNLL-2003shared task:language-independent named entity recognition[C]//Proceedings of the seventh conference on Natural language learning at HLT-NAACL.Edmonton,Canada,2003:142-147.
[6] Kim S,Song Y,Kim K,et al.MMR-based Active Machine Learning for Bio Named Entity Recognition[C]//Proceedings of the Human Language Technology Conference of the NAACL,New York,2006:69-72.
[7] Tsochantaridis I,Hofmann T,Joachims T,et al.Support vector machine learning for interdependent and structured output spaces[C]//Proceedings of the twenty-first international conference on Machine learning.Banff,Alberta,Canada,2004:104.
[8] Zhou G D,Su J.Named entity recognition using an HMM-based chunk tagger[C]//Proceedings of the 40th Annual Meeting on Association for Computational Linguistics,Philadelphia,Pennsylvania,2002:473-480.
[9] Lin Y F,Tsai T H,Chou W C,et al.A maximum entropy approach to biomedical named entity recognition[C]//Proceedings of the 4th ACM SIGKDD Workshop on Data Mining in Bioinformatics.Seattle,WA,2004:56-61.
[10] Settles B.Biomedical named entity recognition using conditional random fields and rich feature sets[C]//Proceedings of the International Joint Workshop on Natural Language Processing in Biomedicine and its Applications,Geneva,Switzerland,2004:104-107.
[11] Florian R,Ittycheriah A,Jing H Y,et al.Named entity recognition through classifier combination[C]//Proceedings of the seventh conference on Natural language learning at HLT-NAACL 2003,Edmonton,Canada,2003:168-171.
[12] Li Y P,Lin H F,Yang Z H.Incorporating rich background knowledge for gene named entity classification and recognition[J].BMC Bioinformatics,2009,10:223.
[13] Segura-Bedmar I,Martínez P,Segura-Bedmar M.Drug name recognition and classification in biomedical texts:A case study outlining approaches underpinning automated systems[J].Drug Discovery Today,2008,13(17-18):816-823.
[14] World Health Organization Programme on International Nonproprietary Names.(2006)The use of stems in the selection of International Nonproprietary Names(INN)forpharmaceutical substances.WHO Press,World Health Organization.[DB/OL].http://www.who.int/medicines/services/inn/Revised-FinalStemBook2006.pdf.
[15] 徐博,林鸿飞,杨志豪.基于模板抽取和丰富特征的药名词典生成[C].第五届全国信息检索学术会议论文集,2009.
