对基因组内不同序列分布差异进行量化的探讨△
2014-04-03崔跃华张岩波
李 治 崔跃华,2 张岩波,△
基因组常被比喻为一本“生命天书”,如果有几个关键词在书中出现的位置比较接近,则其关系可能比较密切。反之,若其出现的位置具有明显差异,则其可能关系较远。如果把序列看作是基因组内的关键词,则序列在基因组上出现的位置差异则可能说明这些序列之间的功能差异。如果两个重复序列在同一基因组上的分布相同,则说明这些序列之间可能有非常密切的关系。如果不同,则可以通过分布一致性检验来判断其差异是否具有统计学意义。当其差异具有统计学意义时,我们一般很想知道其差别到底有多大,以此来推断两者之间功能的差异。由于P值受样本含量的影响,其大小难以反应分布之间的差异,因此需要寻找一个合适的指标对分布差异进行量化。相对熵(relative entropy),又称KL散度(Kullback-Leibler divergence),是衡量不同分布之间差异的常用方法。但其有两个缺陷:①当计算的顺序不一样时,其结果不同;②对于定量数据,一般需将抽样数据进行适当分组再计算,而分组会损失一部分样本信息。
Kolmogorov-Smirnov检验(KS检验)一般用于两种分布之间是否有差异的假设检验[1]。该方法完全避免了相对熵计算所存在的两个缺陷。那么是否可以利用KS检验的统计量对分布之间差异进行量化呢?另外,不同的分布具有不同累积概率曲线,而图心(centroid)可以视为一个图形的中心,那么累积概率曲线下图形的图心差异也有可能用于衡量分布之间的差异。本文就对这两个指标进行了一些探讨。
研究方法
1.赋值方法
如果将基因组看作[0,1],则基因组上序列出现的位置即可表示为[0,1]内的数字,如:

基因组序列:CTTTACGCCT碱基T位置:23410数字转换:0.20.30.41.0
其他碱基以此类推。当基因组很长时,若干碱基组成的序列就可能大量重复出现在基因组的不同位置上,这些位置也都可以转为[0,1]上的数字,这样不同序列的分布就可转化为[0,1]上的各种分布。基因组越长,所需要的精度越高,如基因组长度为106的,则精度为10-6,这样就可对不同序列的分布进行比较。
2.KS检验原理
KS检验是比较样本与理论分布之间,或两样本之间,累积概率的最大差异。在R语言[2-3]中KS检验的统计量表示为D。该统计量D即累积概率的最大差异,由D值计算两样本相同的概率。
3.图心的计算
图心横坐标的计算:作一条垂直于横轴的线,若该线可以将图形面积二等分,则该线与横轴的交点为图心的横坐标。图心纵坐标的计算:作一条垂直于纵轴的线,若该线可以将图形面积二等分,则该线与纵轴的交点为图心的纵坐标。如图1所示,图中曲线为β(3,3)的累积概率曲线,其曲线下面积分别被两条平行于x轴和y轴的虚线平分,这两条虚线的交点即该图形的图心。对于理论分布,采用迭代的方法查找图心坐标,最终坐标的误差小于10-10。对于抽样数据,则直接计算二分面积的位置。
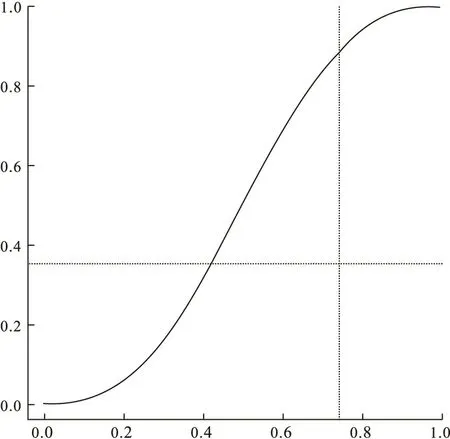
图1 图心示意图
数值模拟结果分析
采用数值模拟的方法对[0,1]上的若干分布进行了分析。所有随机数字的获取、统计分析和作图都使用R语言完成。数据模拟形式如上述。
1.样本含量对两个指标的影响
(1)样本含量对统计量D的影响
数值模拟:分别根据5个β分布生成随机数字,然后利用KS检验进行均匀分布一致性检验。5个分布分别为β(1,1)、β(3,3)、β(1.5,0.8)、β(2,0.5)、β(0.5,2),分别代表5条不同的序列。β(1,1)即均匀分布。5个分布的累积概率曲线如图2所示。

图2 五种分布的累积概率曲线
抽样的样本含量从100开始,逐次加100,直至10000。每个样本含量下进行1000次抽样。记录每组抽样数据均匀分布一致性检验所得的D值。每个样本含量根据所获得的1000个D值计算5个百分位数,即2.5%,25%,50%,75%,97.5%。计算结果如图3所示。

图3 样本含量与D统计量的关系(100~10000)
在图3中,每个分布由5条曲线构成,从下到上,分别表示D值的5个百分位数。从图中可见,随着样本含量的增加,D值的离散趋势逐渐减小,但其集中趋势并没有受到明显影响,且不同的分布集中于不同的位置。表明D值可以看做分布差异的一种量化指标。另外,从图中可以看到β(2,0.5)和β(0.5,2)两个分布的数据基本重合。
(2)样本含量对图心差异的影响
图心差异采用图心之间的距离表示,数值模拟过程同前。计算结果如图4所示。

图4 图心差异与样本含量的关系(100~10000)
从图中可见,样本含量对图心之间距离的影响和对统计量D的影响相似。即随着样本含量的增加,图心距离的离散趋势逐渐减小,但集中趋势并没有受到明显影响,且不同的分布集中于不同的位置。表明图心距离也可以看做分布差异的一种量化指标。
2.不同样本含量下两指标能判别的最小差异
(1)KS检验的统计量D

从表1可见,当样本含量大于100时,所能判别的最小D值差异已小于0.1。虽然随着样本含量的增加,所能判别的最小D值差异不断减小,但其差异是否具有实际意义可能值得商榷。
(2)图心距离
计算方法同前,计算结果见表1。当样本含量为100时,其最小判别差异在0.02附近。虽然在绝对值上比统计量D小,但需注意理论上此时图心距离的最大差异小于0.5,而统计量D的最大差异是小于1。另外,从变异程度上看,图心距离的变异程度约为统计量D的两倍。
3.在同一坐标系内标记不同分布
(1) KS检验的统计量D
从前一部分结果可以看到,虽然统计量D可以用于区分两个分布的差异,但正如β(2,0.5)和β(0.5,2)两个分布和β(1,1)的关系一样,当使用一个分布作为基准,存在两个不同分布的D值相同的情况。为能够区分各种不同的分布,可以选择两个分布作为基准分布。本实验选择β(2,1)和β(1,2)为基准分布。
数值模拟:分别根据5个分布生成随机数字,然后针对两个基准分布分别进行分布一致性检验。5个分布分别为β(1,1)、β(3,3)、β(1.5,0.8)、β(2,0.5)、β(0.5,2)。抽样的样本含量为1000,每个分布进行1000次抽样,记录所得D值的平均值。计算结果如图5所示。
在图2中,由于β(1,1)累积概率曲线是β(2,0.5)和β(0.5,2)两个累积概率曲线的对称轴,因此使用β(1,1)作为基准就很难区分二者的差别。从图5可见,当采用β(2,1)和β(1,2)两个基准分布时,即可将5个分布分别标记在不同的位置。
(2)图心
5个分布的累积概率曲线下图形的图心采用迭代法计算,计算结果如图6所示。由于不同分布具有不同的累积概率曲线,且累积概率曲线只能是单调递增曲线。因此不同的分布会对应不同的图心。
实例分析
选取大肠杆菌K-12(MG1655)参考基因组序列,该序列从NCBI网站获取,全长4639675bp。待测序列为GTGGGCG、GGCGGGG、CCCTCTT、CCCTGCC、CCGCCCT、GCCACCG。6条序列可分别获取其在基因组中的位置,其重复出现的次数即检验所需的样本含量。根据这些位置进行均匀分布一致性检验,并在转换为[0,1]内的数字后计算图心。
6条序列的统计结果见表2。在表2中,P和D分别为均匀分布一致性检验的P值和统计量,x和y分别为计算所得图心的横坐标和纵坐标。其图心在直角坐标系中的表示见图7。从图中可见6条序列大致分为3群。

表1 不同样本含量下统计量D和图心距离能判别的最小差异
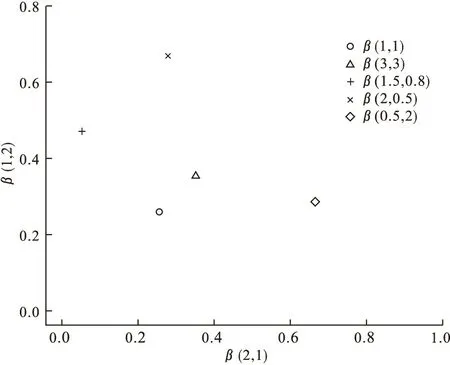
图5 利用两个基准分布和统计量D 显示五个不同分布的差异
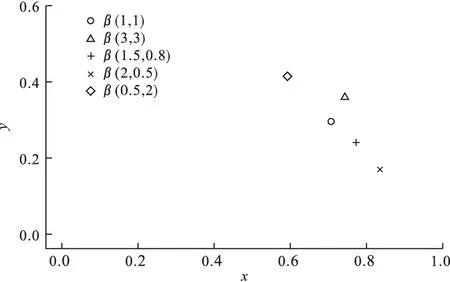
图6 利用图心显示五个不同分布
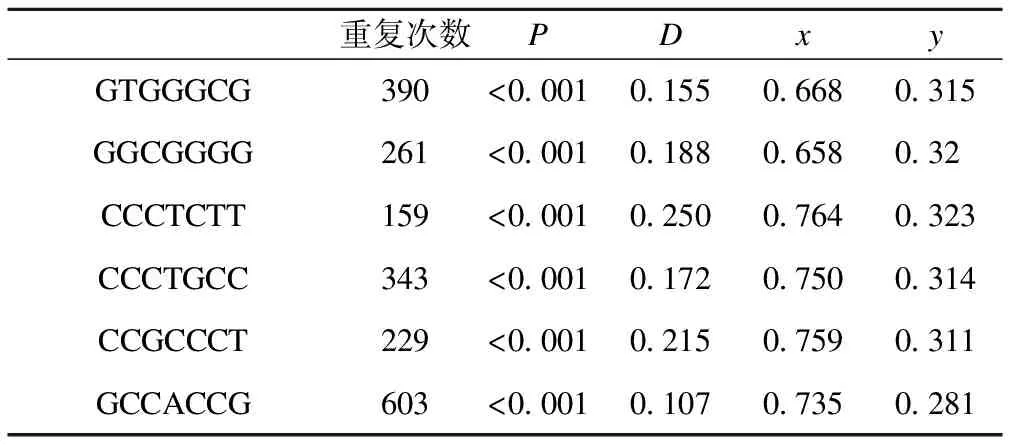
表2 6条序列的分布差异
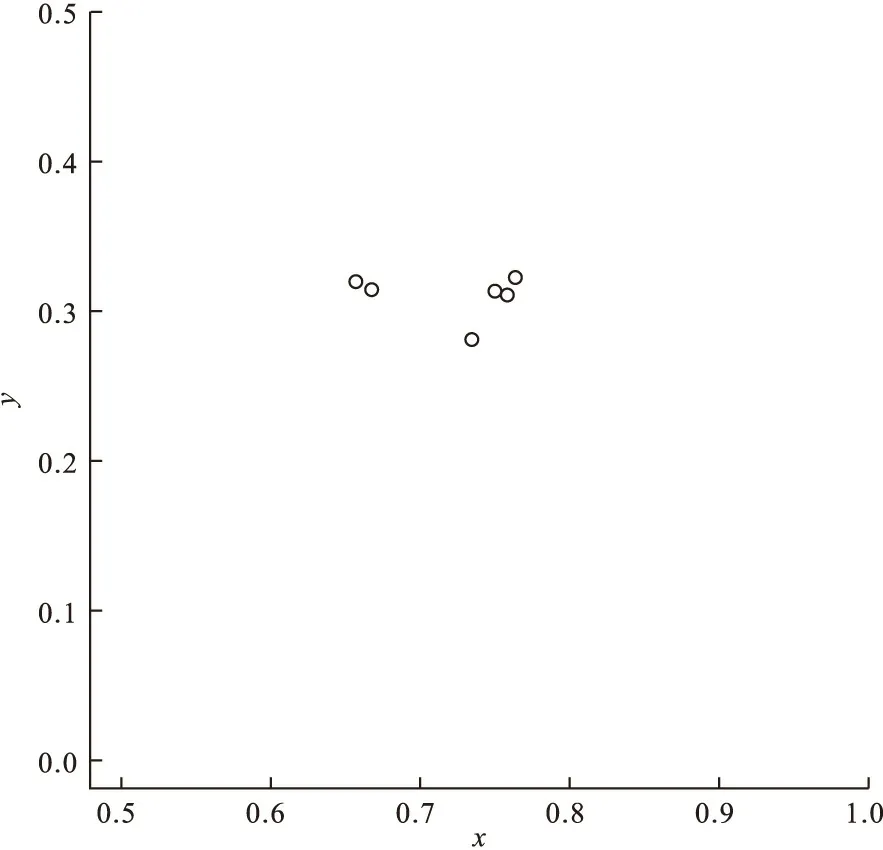
图7 6条序列的图心表示
讨 论
如前所述,基因组上任一序列的分布都可转为[0,1]分布,这样就可以比较不同序列在基因组上的分布差异。但在计算分布差异时,若使用相对熵来衡量分布之间的差异,由于其对概率差异进行了加权平均,导致计算顺序不同会得出不同的结果,而这样的结果往往不是我们所预期的。KS检验的统计量D以及累积概率曲线下图形的图心差异则与此不同。由前面实验结果可知,二者随着样本含量的增加,离散趋势逐渐减小,而集中趋势没有明显影响。因此认为这两个指标可以用于衡量不同分布的差异。

当考察同一基因组上多个序列之间的关系时,若能将所有序列的分布画在一个坐标系内,则可以使序列分布之间的差异比较直观。因此本实验还探讨了在同一坐标系标记任意两个分布差异的可行性。当使用D值差异时,由于对称性的存在,采用单个基准分布将有可能出现两个分布D值差异相同的情况。此时采用两个基准分布可消除这一缺陷,而且可以将不同分布表示在同一坐标系,但该坐标系内分布之间的差异不一定等于D值差异。但此时需注意,若采用不同的基准分布,则相当于换了不同的坐标系,分布之间的关系也将发生改变。若采用图心指标,不同的累积概率曲线会对应不同的图心,而且图心不会像D值那样随基准分布而变。另外由于D值反映的是最大累计概率差异,而图心则与整个图形有关,因此采用图心指标作图可能会更好地反映分布之间的差异。
在实例分析中,我们选取了同一基因组上的6条序列进行了分析。正如本文开头所言,分布之间的关系可能作为功能联系密切程度的一个指标,因此该实例分析结果显示图心有可能作为序列功能聚类的一种指标。
最后,由于本研究采用数值模拟的方法,并不能完全反应各种实际情况,有待在实际运用中探讨证实。
参 考 文 献
1.Frank J,Massey Jr.The Kolmogorov-Smirnov test for goodness of fit.Journal of the American Statistical Association,1951,46(253):68-78.
2.邓宇,彭斌,田考聪.R统计计算环境简介.中国卫生统计,2008,25(4): 421-422.
3.R Core Team (2013).R: A language and environment for statistical computing.R Foundation for Statistical Computing,Vienna,Austria.ISBN 3-900051-07-0,URL http://www.R-project.org/.
4.Pham-Gia T,Hung TL.The mean and median absolute deviations.Mathematical and Computer Modelling,2001,34(7): 921-936.
