早酥梨及其红色芽变Ty1-copia反转录转座子逆转录酶的克隆与分析
2014-03-25仇宗浩徐凌飞
周 鹏,仇宗浩,翟 锐,梁 东,徐凌飞
(西北农林科技大学 园艺学院,农业部西北地区园艺作物生物学与种质创制重点实验室,陕西 杨凌712100)
反转录转座子是植物基因组中可以自由移动的基因片段,转座子通过DNA-RNA-DNA的方式,发生逆转录后整合到基因组中的新位点,可以影响其他基因的表达和基因组的变化,创造稳定的插入突变[1-2]。反转录转座子在植物基因组分析[3]、连锁作图[4]、系统进化[5-6]及生物多样性研究[7]等方面具有广泛的应用前景。逆转录酶是反转录转座子发生转座的重要调控因子,其序列的保守性影响着转座的发生。
迄今为止,已有许多反转录转座子在果树中被分离和鉴定,用以研究其对果树基因组组成、系统进化以及基因表达调控方面的重要作用[8-10]。孙俊等[11]应用改良的Pearce方法,分离了苹果Ty1-copia反转录转座子RNaseH-LTRs序列;Tao等[12]发现,反转录转座子是导致柑橘基因组突变和进化的重要原因,芽变的逆转录酶序列存在移框、缺失等多种突变情况;Rico-Cabanas等[13]首次从柑橘中获得完整的copia反转录转座子CIRE1,并发现其在根中特异表达。另有研究表明,反转录转座子的插入导致果树性状发生了改变,如Harada等[14]研究发现,苹果一个耐贮藏突变品种可能是ACC合成酶基因启动子-781 bp处插入了一个162 bp的类似反转录转座子片段所致;Yao等[15]研究发现,RaeIme等单性结实的苹果芽变是由于反转录转座子插入MdPI基因内含子影响其表达而产生的性状;Kobayashi等[16]研究表明,红皮葡萄芽变Ruby Okuyama和Flame Muscaty,分别是由Gret1反转录转座子插入白皮葡萄Italia和Muscat of Alexandria的Vvmyb1基因中所致。Ty1-copia反转录转座子逆转录酶具有异质性和高拷贝的特点,已经在火龙果[17]、苹果[18]、草莓[19]、黄瓜[20-21]、白菜[22]等植物中得到验证。目前,只有Shi等[23]和Kim等[24]对日本梨Ty1-copia反转录转座子进行了鉴定分析,但从梨母本和芽变基因组中分离逆转录酶序列并进行分析的研究却鲜见报道。本研究拟从早酥梨及其红色芽变基因组中克隆Ty1-copia反转录转座子逆转录酶序列,并运用生物信息学技术对其进行对比分析,以期通过研究Ty1-copia反转录转座子探索早酥梨与其红色芽变的遗传关系,进而为早酥梨的遗传进化提供信息支持。
1 材料与方法
1.1 材 料
以白梨品种“早酥”及其红色芽变叶片为试材,2011-04从西北农林科技大学梨种质资源圃采集幼叶,立即用液氮速冻后,-70 ℃保存备用。试验在农业部西北地区园艺作物生物学与种质创制重点实验室进行。采用改良的SDS酚法提取叶片总DNA[25],用8 g/L琼脂糖凝胶电泳和NanoDrop2000(Thermo)仪器检测DNA质量浓度和纯度。用TE缓冲液稀释DNA至100 ng/μL,保存于-20 ℃冰箱中备用。
1.2 逆转录酶序列的克隆
根据Ty1-copia反转录转座子逆转录酶保守基序Ⅰ(TAFLHG)和Ⅲ(YVDDML)设计扩增基因片段的简并引物[26],上游引物NM1的序列为5′-ACNGCNTTPyPyTNCAPyGG-3′,下游引物NM2的序列为5′-APuCATPuTCPuTCNACPuTA-3′,其中N=A+T+C+G,Pu=A+G,Py=T+C。
PCR反应体系25 μL:10×TaqBuffer 2.5 μL,25 mmol/L MgCl21.5 μL,2.5 mmol/L dNTP 0.8 μL,5 U/μLTaq聚合酶(Fermentas) 0.2 μL,10 μmol/L引物各1 μL,ddH2O补足至25 μL。以早酥梨DNA为模板,建立并优化梨Ty1-copia反转录转座子逆转录酶序列的PCR反应体系。将PCR反应体系中的MgCl2、dNTP、引物、Taq酶等成分设置不同的用量(表1),当一种成分的用量发生改变时,保持其他成分用量不变,通过扩增效率的对比分析确定各成分的最佳用量。

表1 早酥梨Ty1-copia反转录转座子逆转录酶序列PCR体系的优化
PCR反应程序为:94 ℃预变性2 min;94 ℃变性1 min,设置不同的退火温度(45.9,47.5,49.5,51.5,53.2,55.0,57.0 ℃)复性50 s,72 ℃延伸50 s,35个循环;最后72 ℃延伸5 min。整个反应在Life-Express PCR反应仪中完成。PCR产物用15 g/L琼脂糖凝胶进行电泳检测。
1.3 逆转录酶序列的获得及分析
在紫外灯下对凝胶拍照后,用琼脂糖凝胶回收试剂盒(Biomiga)回收目的片段,与pGM-T (TIANGEN)载体于16 ℃连接8~10 h。连接产物转化到Top10大肠杆菌感受态细胞中,涂于含有Amp/X-gal/IPTG 的LB 固体培养基上,37 ℃过夜培养后,挑取白色克隆,在含有100 mg/L氨苄青霉素的 LB 液体培养基中培养。将每个经过检测的阳性克隆分装成2个,送往北京奥科鼎盛生物科技有限公司利用T7或SP6引物测序,获得目的基因序列。
在NCBI网站对测序结果进行BLASTN,用BLASTX程序进行同源比对,搜索库中已知物种的逆转录酶序列,利用DNAStar软件对序列进行聚类分析,用Mega 5.0软件构建系统进化树。
1.4 Ty1-copia反转录转座子转录活性的检测
采用改良的SDS酚法提取叶片RNA,利用PrimeScript RT reagent Kit with gDNA Eraser(TaKaRa)试剂盒去除基因组DNA,将RNA反转录成cDNA,分别以RNA和cDNA为模板PCR扩增Actin(GU830958)基因用以检测样品质量。以cDNA为模板,利用已经优化好的体系扩增逆转录酶序列,检测早酥梨及其红色芽变Ty1-copia反转录转座子的转录活性。
2 结果与分析
2.1 早酥梨逆转录酶序列PCR扩增体系的建立与优化
以早酥梨DNA为模板,根据表1将PCR反应体系中的MgCl2、dNTP、引物、Taq酶等成分设置不同用量进行扩增,结果如图1所示。
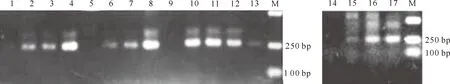
图1 不同组分PCR反应体系扩增早酥梨逆转录酶的检测结果
由图1可以看出,不同的PCR扩增体系中,随组分及用量的变化,PCR扩增效率亦会随之改变。随着dNTP用量的增加,PCR产物增多,以1.2 μL dNTP时的扩增产物最多;虽然取用0.3 μLTaq酶时逆转录酶的扩增效率较高,但同时出现了非特异性产物,故选用0.2 μLTaq酶进行扩增;不同的引物用量对逆转录酶序列的扩增也有影响,随着引物用量增大扩增产物减少,故确定引物用量为0.8 μL;当MgCl2用量为 1.5 μL时,扩增产物具有特异性。所以,经过优化后的PCR反应体系为:10×TaqBuffer 2.5 μL,25 mmol/L MgCl21.5 μL,2.5 mmol/L dNTP 1.2 μL,5 U/μLTaq聚合酶0.2 μL,10 μmol/L 引物各0.8 μL。
同时,为进一步优化反应程序,设置不同的退火温度进行扩增试验,结果(图2)显示,不同退火温度时的PCR扩增效率不同,51.5 ℃为逆转录酶PCR扩增体系的最佳退火温度。
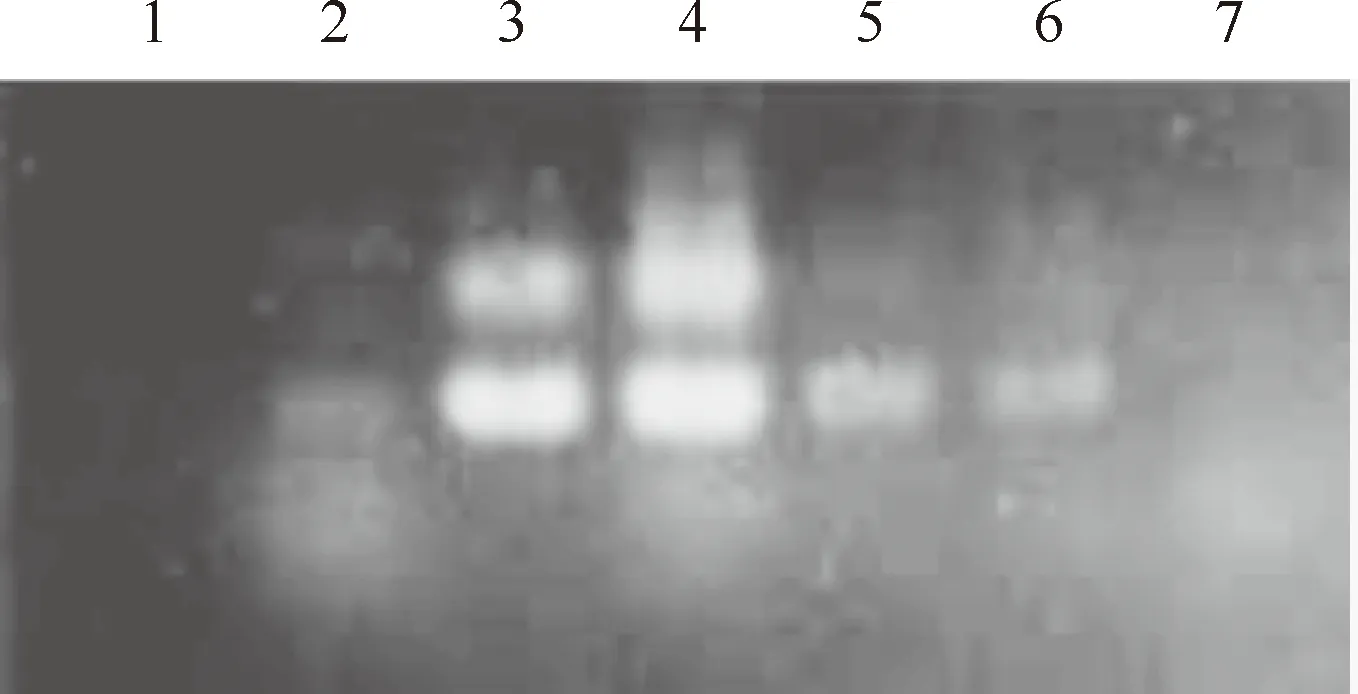
图2 退火温度对早酥梨逆转录酶PCR扩增结果的影响
2.2 早酥梨及其红色穿变逆转录酶的扩增与检测
利用优化后的PCR体系在早酥梨及其红色芽变基因组中都能得到扩增产物(图3),长度约为260 bp,从片段大小尚未看出二者有何差异。经过测序比对,成功从早酥梨基因组中获得45条逆转录酶序列,从芽变基因组中获得60个克隆。所得序列中,除早酥梨2个序列的BLASTX比对结果与一种转座酶相似外,其余序列均与Ty1-copia反转录转座子逆转录酶序列有很高的相似性。经聚类分析,将小于200 bp的序列去掉,并将相同的序列归为1个,最后按照顺序,将所得的29条早酥梨Ty1-copia反转录转座子逆转录酶序列命名为PbRTzs1~PbRTzs29,将30条红色芽变逆转录酶序列命名为PbRTyb1~PbRTyb30。现已将59条序列全部提交到NCBI数据库,登录号见表2。
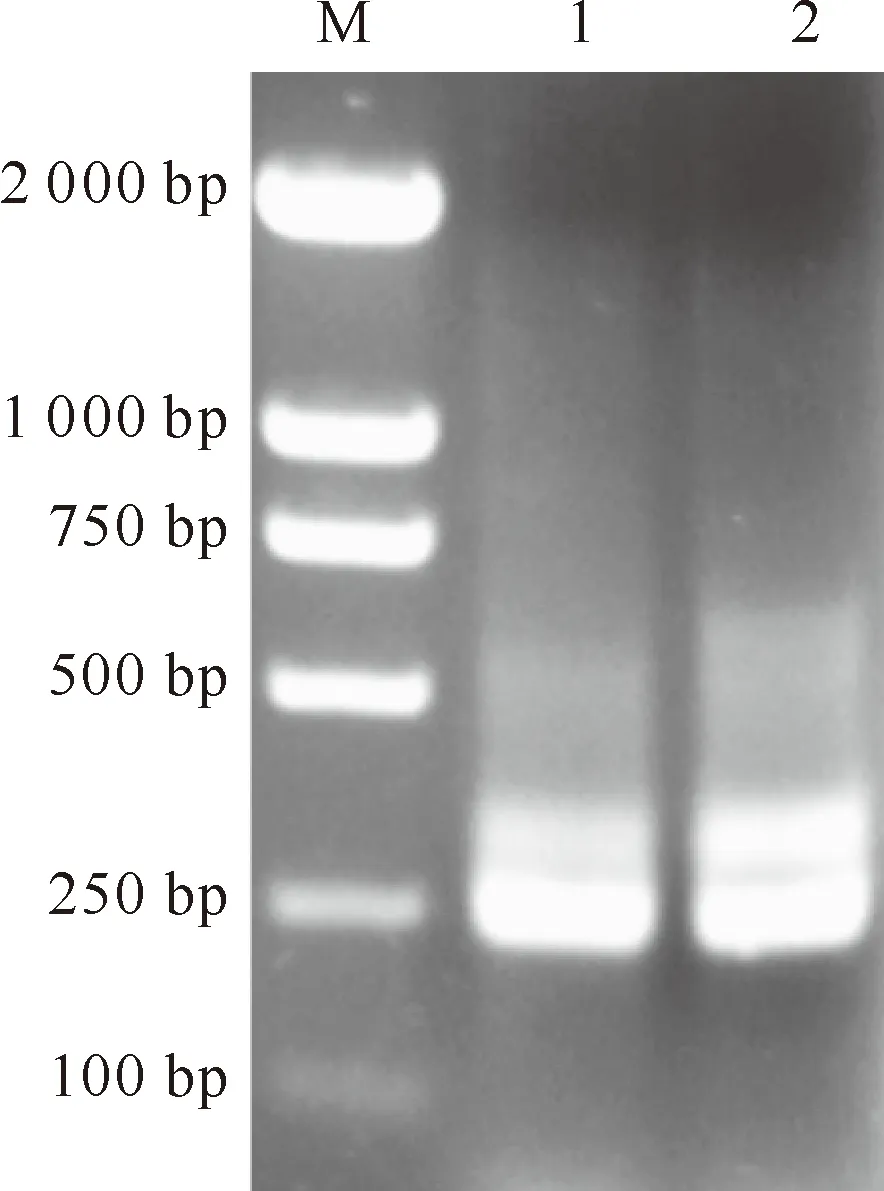
图3 早酥梨及其红色芽变逆转录酶序列的 PCR 扩增
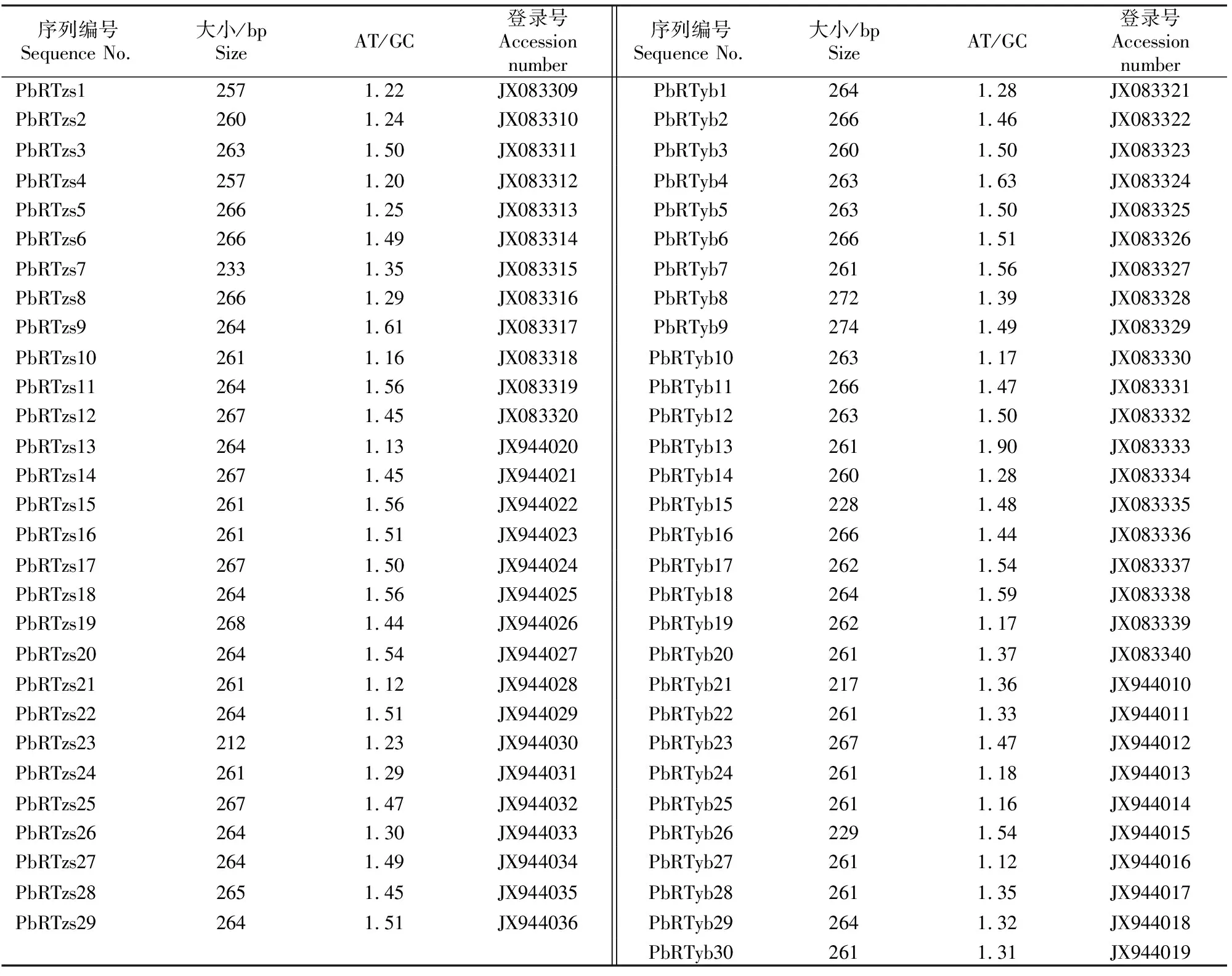
表2 早酥梨及其红色芽变逆转录酶序列的组成
2.3 早酥梨及其红色芽变Ty1-copia反转录转座子逆转录酶的序列对比
从早酥梨及其红色芽变基因组中克隆到的逆转录酶,序列大小与前人的报道一致[18,22]。其中,PbRTyb8和PbRTyb9的长度分别为272和274 bp,虽比较特殊,但在Voytas等[26]的研究中也有发现。从表2可以看出,早酥梨基因组中的逆转录酶序列大小为212~268 bp,AT/GC为1.12~1.61,而红色芽变的逆转录酶序列大小为217~274 bp,AT/GC为1.12~1.90。将2组序列分别导入DNAStar软件运用ClustalW[27]方法计算序列之间的差异性和同源性,结果显示,早酥梨逆转录酶序列的差异度为0.4%~89.8%,相似性为48.3%~99.6%;其芽变逆转录酶序列之间的差异度为 1.5%~96.3%,相似性为45.8%~98.5%。2种材料逆转录酶序列的大小及碱基变化,反映了梨基因组中的Ty1-copia反转录转座子逆转录酶同样具有异质性特点。
利用DNAStar软件将逆转录酶序列模拟合成氨基酸序列,发现均不含内含子。参考BLASTX比对结果中的Ty1-copia反转录转座子逆转录酶氨基酸序列,对早酥梨及其红色芽变逆转录酶氨基酸序列进行分析,结果(图4和图5)发现,早酥梨Ty1-copia反转录转座子逆转录酶保守氨基酸的一致性为62.22%,芽变的一致性为53.30%,且均可以找到逆转录酶序列的3段保守区域“TAFLHG”、“KSLYGLKQ”、“LLYVDDM”以及相对保守的“QP”、“GF”片段。
对早酥梨及其红色芽变逆转录酶氨基酸序列突变情况进行分析,结果见表3。由表3可知,各序列发生了不同种类的突变,主要包括终止子突变、缺失突变和替换突变。具体而言,早酥梨逆转录酶序列PbRTzs3由于碱基的替换,出现1个终止子突变(第46个氨基酸),而且发生在“KSLYGLKQ”保守区域;序列PbRTzs19由于1个碱基的插入,导致后面的序列发生移框,出现了2个终止子突变(第16和86个氨基酸);PbRTzs28第38个氨基酸发生缺失突变;PbRTzs1和PbRTzs4的保守区域均发生了1个氨基酸替换突变(L变为F),但未发现缺失突变和移框突变情况。而在红色芽变的逆转录酶序列中,由于碱基替换序列PbRTyb7(第80个氨基酸)、PbRTyb14(第15个氨基酸)、PbRTyb24(第54个氨基酸)、PbRTyb25(第30个氨基酸)存在1个终止子突变,同时序列PbRTyb7由于碱基的替换导致后面序列的移框突变,PbRTyb4(第41和46个氨基酸)存在2个终止子突变,PbRTyb15(第53,55和61个氨基酸)、PbRTyb26(第53,55和61个氨基酸)存在3个终止子突变,PbRTyb9(第9,33,53,67和73个氨基酸)、PbRTyb17(第39,57,78,81和84个氨基酸)、PbRTyb19(第64,65,70,76和84个氨基酸)序列存在5个终止子突变;PbRTyb9序列由于第37位碱基“A”的插入,导致后面序列发生移框突变,PbRTyb7由于缺失突变(第71个氨基酸处)导致随后的序列发生移码丧失基因功能,PbRTyb27由于第153处单碱基的插入导致后面的序列发生了移框突变;PbRTyb15(第51个氨基酸开始)、PbRTyb17(第33个氨基酸)、PbRTyb19(第63个氨基酸处)序列同样发生了缺失突变;PbRTyb1(第1个氨基酸)、PbRTyb4(第34个氨基酸)、PbRTyb6(第4个氨基酸)、PbRTyb9(第34个氨基酸)、PbRTyb11(第4个氨基酸)、PbRTyb17(第34个氨基酸)、PbRTyb19(第34个氨基酸)都发生了替换突变。由此推测,逆转录酶序列的异质性是由于终止子突变、缺失突变、替换和移框突变造成的,并且红色芽变的变异程度明显高于早酥梨。

图5 早酥梨红色芽变Ty1-copia反转录转座子逆转录酶氨基酸序列的同源性比较
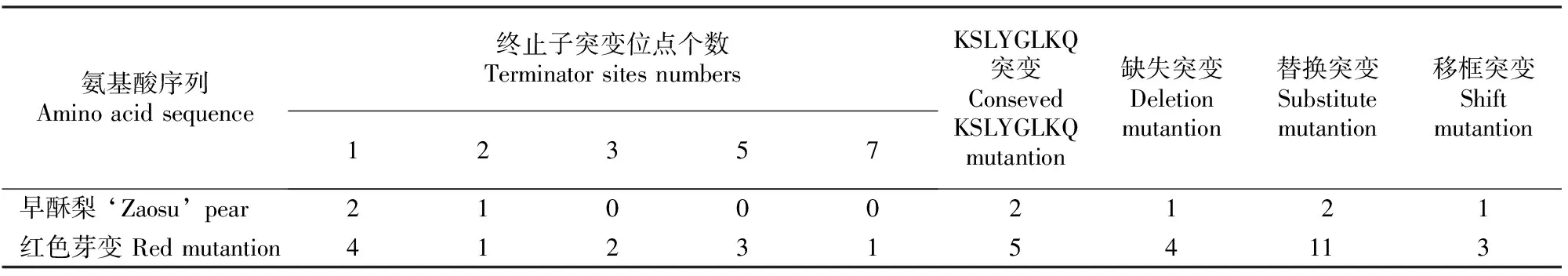
表3 早酥梨及其红色芽变逆转录酶氨基酸序列突变情况的比较
2.4 早酥梨及其红色芽变逆转录酶基因的系统进化树
为探明早酥梨基因组中克隆得到的59条序列之间的关系,利用Mega 5.0软件以Kimura 2-parameter算法,采用邻接法构建反转录转座子逆转录酶的系统进化树(图6)。根据进化树分枝程度可将所有序列分为7个家族(Family 1~7),代表了来自同一祖先的7个独立的遗传家系。其中家族1包括15条序列,家族2包括1条序列,家族3包括14条序列,家族4包括1条序列,家族5包括12条序列,家族6包括2条序列,家族7包括14条序列。
通过进化树分析,可以找到亲缘关系相近的早酥梨及其红色芽变的逆转录酶序列。在各家族的核苷酸序列当中,都有不同程度的突变情况发生。第1家族的序列主要来自于早酥梨,第5家族的序列主要来自于红色芽变,而在第3和第7家族中,早酥梨和红色芽变的逆转录酶序列各占1/2,第2、第4和第6家族所包含的序列较少且均来自于红色芽变材料,这3类与其余家族序列的遗传距离较大,而且第6家族的PbRTyb15和PbRTyb26均是含有3个终止子突变的序列。在这些家族中,由于缺失突变造成序列严重不完整,可能已经不具有转录活性。
为研究早酥梨与其他物种Ty1-copia反转录转座子的进化关系,从NCBI网站上下载参考序列,包括苹果(Malus×domestica)ABD76549.1、拟南芥(Arabidopsisthaliana)AAF02855.1、鹰嘴豆(Cicerarietinum)CAD59770.1、柑橘(Citrussinensis)CAJ09951.2、果蝇(Drosophilamelanogaster)CAD27357.1、番薯(Ipomoeabatatas)AAV88069.1、粳稻(OryzasativaJaponica Group)ABA98656.1、杨(Populusdeltoides)CAC95126.1、李(Prunusmume)ACZ36927.1、豇豆(Vignaradiata)AAT90460.1、茄属(Solanumchilense)AAK29467.1、烟草(Tobacco)P10978.1和玉米(Zeamays)T02087。从每个家族中各选取早酥梨及其红色芽变的逆转录酶序列与其他植物建立进化树,结果见图7。从图7可以看出,早酥梨不同家族的逆转录酶氨基酸序列与其他物种的遗传距离为0.023~1.020。第1,2,4,5和6家族与其他物种的遗传距离较远,第3家族的序列与拟南芥(Arabidopsisthaliana)AAF02855.1的序列和玉米(Zeamays)T02087的序列遗传距离较近;第7家族的氨基酸序列与苹果(Malus×domestica)和李(Prunusmume)的进化关系非常近,PbRTzs6与二者的遗传距离分别为 0.023 和 0.035,PbRTyb11与二者的遗传距离分别为0.083和0.095。不同逆转录酶序列与不同物种遗传距离的差异,可能是由于反转录转座子在进化过程中发生横向传递所致。
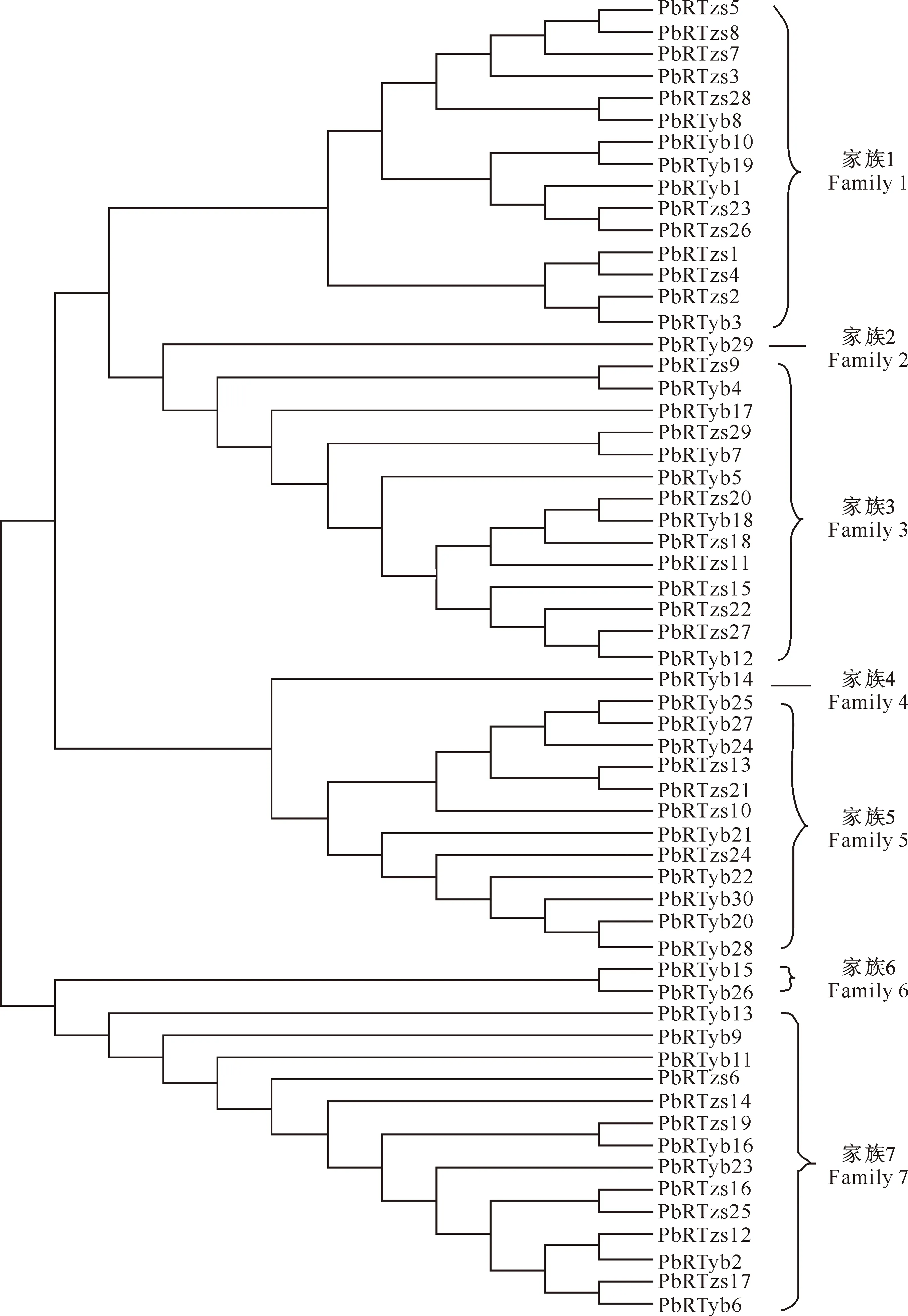
图6 早酥梨及其红色芽变Ty1-copia反转录转座子逆转录酶核苷酸序列的系统进化树
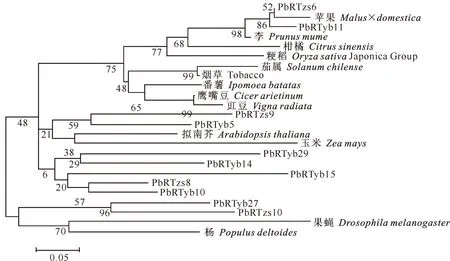
图7 早酥梨及其红色芽变与其他植物Ty1-copia反转录转座子逆转录酶氨基酸序列的进化树
2.5 早酥梨及其红色芽变Ty1-copia反转录转座子转录活性的检测
由图8可知,Actin内参基因在早酥梨及其红色芽变中扩增条带明亮、无杂带,说明反转录合成的品质很好;以RNA为模板无任何扩增产物产生,说明DNA杂质去除彻底。以cDNA为模板扩增逆转录酶序列,则无扩增产物出现,说明Ty1-copia反转录转座子在早酥梨及其红色芽变植物中已经失去转录活性,从而稳定地存在于植物基因组中。
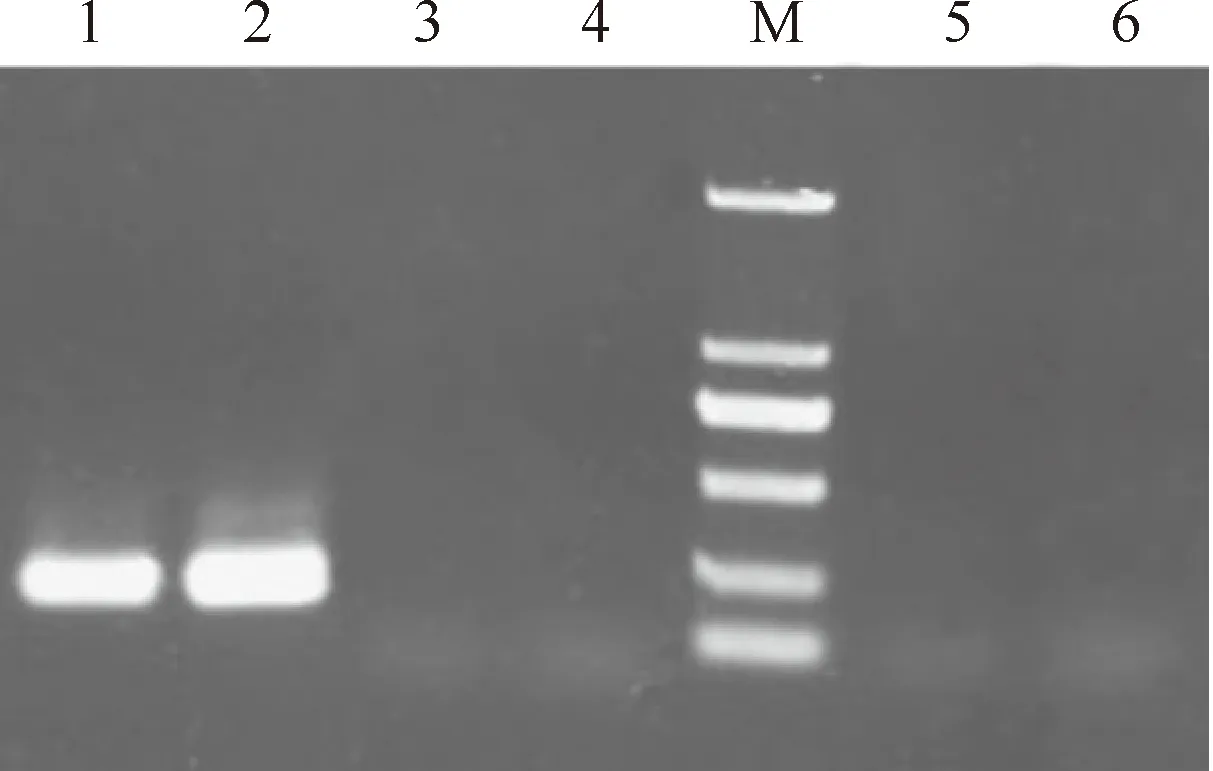
图8 早酥梨及其红色芽变Ty1-copia反转录转座子转录活性的检测
3 讨 论
Liu等[28]利用早酥梨及其红色芽变材料构建SSH文库,发现了与苹果(NCBI Accession No.AEX97092.1)、麝香百合(NCBI Accession No.ABM68560.1)和水稻(NCBI Accession No.ABA95643.1)Ty1-copia反转录转座子具有较高同源性的EST序列(NCBI Accession No.HO774934),并以此为切入点探索反转录转座子与芽变发生的关系。前人对苹果[15]、葡萄[16]等果树芽变机理的研究结果,已为反转录转座子插入是果树芽变的重要原因提供了一些证据。已有大量相关报道指出,反转录转座子在植物基因组中发生转座,是对各种生物或非生物逆境的响应[29-30]。
本研究建立并优化了逆转录酶的PCR扩增体系,并成功从早酥梨及其红色芽变基因组中分离克隆了反转录转座子逆转录酶序列。序列分析显示,碱基替换、插入、缺失所导致的突变,是其具有很高异质性的重要原因,与其他物种逆转录酶序列的特点相似[20,22,31]。这可能是由于反转录转座子发生转座时,逆转录酶无错读校对功能,从而在转座过程中易发生高频突变造成的[30]。另外,反转录转座子之间的同源重组也是导致反转录转座子变异的原因之一[32]。
对逆转录酶序列进行分析,发现红色芽变逆转录酶序列突变的复杂程度明显高于早酥梨,以致大多序列都已经不具有转录活性。由于在反转录转座子的转座过程时,序列很容易突变丧失活性,所以这可作为早酥梨红色芽变反转录转座子发生了转座的证据。序列突变导致反转录转座子丧失活性是有积极意义的,可以保证物种的稳定遗传。植物一方面抑制反转录转座子转座,防止产生致死突变;另一方面,反转录转座子在逆境中的不断转座为基因组的多样性和遗传变异奠定了基础[25]。
利用软件建立进化树,分析早酥梨及红色芽变的逆转录酶序列与其他物种逆转录酶序列的进化关系,发现只有个别序列(家族7)与苹果和李具有较近的遗传距离,甚至小于同一物种逆转录酶序列之间的遗传距离,这可能是反转录转座子横向传递的结果,不同物种可能来自于同一祖先,而纵向传递的不稳定性导致同一物种的序列产生了较大的差异。
4 结 论
本研究利用简并引物首次从早酥梨及其红色芽变基因组中成功克隆了Ty1-copia反转录转座子逆转录酶序列。序列分析结果表明,二者的逆转录酶序列都具有高拷贝数和异质性的特点,这主要是由于终止子突变、缺失突变和移框突变所致,而且红色芽变的逆转录酶序列的突变情况更为复杂。根据进化树分析可知,早酥梨及其红色芽变与苹果和李具有很近的遗传距离,这可能是反转录转座子横向传递的结果。
[参考文献]
[1] Flavell A J,Pearce S R,Kumar A.Plant transposable elements and the genome [J].Current Opinion in Genetics & Development,1994,12(4):838-844.
[2] Park Y J,Dixit A,Yoo J W,et al.Further evidence of microcolinearity between barley and rice genomes at two orthologous regions [J].Mol Cells,2004,17:492-502.
[3] Koo D H,Hong C P,Batley J,et al.Rapid divergence of repetitive DNAs inBrassicarelatives [J].Genomics,2011,97:173-185.
[4] Ochogavía A C,Seijo J G,González A M,et al.Characterization of retrotransposon sequences expressed in inflorescences of apomictic and sexualPaspalumnotatumplants [J].Sex Plant Reprod,2011,24:231-246.
[5] Khaliq I,Khan M A,Pearce S.Ty1-Copia retrotransposons are heterogeneous,extremely high copy number and are major players in the genome organization and evolution ofAgavetequilana[J].Genet Resour Crop Evol,2012,59(4):575-587.
[6] Cenci A,Combes M C,Lashermes P.Genome evolution in diploid and tetraploid Coffea species as revealed by comparative analysis of orthologous genome segments [J].Plant Mol Biol,2012,78:135-145.
[7] Woodrow P,Pontecorvo G,Ciarmiello L F.Isolation of Ty1-copiaretrotransposon in myrtle genome and development of S-SAP molecular marker [J].Molecular Biology Reports,2012,39(4):3409-3418.
[8] Du X,Zhang Q,Luo Z.Development of retortransposon primers and their utilization for germplasm identification inDiospyrosspp.(Ebenaceae) [J].Tree Genetics & Genomes,2009,5:235-245.
[9] Biswas M K,Baig M N R,Cheng Y J,et al.Retrotransposon based genetic similarity within the genusCitrusand its relatives [J].Genetic Resources and Crop Evolution,2010,57:963-972.
[10] Ma Y,He P,Sun H Y,et al.Isolation and characterization of transcriptionally active Ty1-copiaretrotransposons inFragaria×ananassa[J].Agricultural Sciences in China,2010,9 (3):337-345.
[11] 孙 俊,房经贵,王 飞,等.苹果Ty1-copia类逆转座子LTR10序列及其在苹果属植物中的遗传多样性分析 [J].南京农业大学学报,2010,33(1):43-48.
Sun J,Fang J G,Wang F,et al.Analysis on the sequence of Ty1-copia retrotransposon LTR10 and its genetic diversity inMalusgenus [J].Journal of Nanjing Agricultural University,2010,33(1):43-48.(in Chinese)
[12] Tao N G,Xu J,Cheng Y J,et al.Isolation and characterization of copia-like retrotransposons from 12 sweet orange(CitrussinensisOsbeck) cultivars [J].Journal of Integrative Plant Biology Formerly Acta Botanica Sinica,2005,47(12):1507-1515.
[13] Rico-Cabanas L,Martínez-Izquierdo J A.CIRE1,a novel transcriptionally active Ty1-copia retrotransposon fromCitrussinensis[J].Mol Genet Genomics,2007,277(4):365-377.
[14] Harada T,Sunako T,Wakasa Y,et al.An allele of the 1-aminocyclopropane-1-carboxylate synthase gene(Md-ACS1) accounts for the low level of ethylene production in climacteric fruits of some apple cultivars [J].Theoretical and Applied Genetics,2000,101:742-746.
[15] Yao J,Dong Y,Morris B A.Parthenocarpic apple fruit production conferred by transposon insertion mutatons in a MADS-box transcription factor [J].Proc Natl Acad Sci USA,2001,98(3):1306-1311.
[16] Kobayashi S,Goto-Yamamoto N,Hirochika H.Retrotransposon-induced mutation in grape skin color [J].Science,2004,304:982.
[17] 范付华,乔 光,郑思成,等.火龙果Ty1-copia类反转录转座子反转录酶序列的克隆及分析 [J].园艺学报,2012,39(2):265-272.
Fan F H,Qiao G,Zheng S C,et al.Cloning and analysis of reverse transcriptase of Ty1-copiaretrotransposons inHylocereusundatus[J].Acta Horticulturae Sinica,2012,39(2):265-272.(in Chinese)
[18] 孙 俊,房经贵,孙其宝,等.苹果Ty1-copia类逆转座子家族鉴定及特性分析 [J].西北植物学报,2009,29(12):2373-2377.
Sun J,Fang J G,Sun Q B,et al.Identification and characteristics of apple Ty1-copia-like retrotransposons [J].Acta Bot Boreal Occident Sin,2009,29(12):2373-2377.(in Chinese)
[19] Ma Y,Sun H Y,Zhao G L,et al.Isolation and characterization of genomic retrotransposon sequences from octoploid strawberry (Fragaria×ananassaDuch.) [J].Plant Cell Reports,2008,27:499-507.
[20] Jiang B,Wu Z M,Lou Q F,et al.Genetic diversity of Ty1-copia retrotransposons in a wild species ofCucumis(C.hystrix) [J].Scientia Horticulturae,2010,127:46-53.
[21] Jiang B,Lou Q F,Wu Z M,et al.Retrotransposon-and microsatellite sequence-associated genomic changes in early generations of a newly synthesized allotetraploidCucumis×hytivusChen & Kirkbride [J].Plant Molecular Biology,2011,77(3):225-233.
[22] 肖 栋,侯喜林,马景蕃,等.不结球白菜Ty1-copia逆转座子序列的克隆及表达 [J].西北植物学报,2009,29(4):643-649.
Xiao D,Hou X L,Ma J F,et al.Cloning and expression analysis of Ty1-copia-like retrotransposons inBrassicacampestrisssp.chinensis[J].Acta Bot Boreal Occident Sin,2009,29(4):643-649.(in Chinese)
[23] Shi Y,Yamamoto T,Hayashi T.Characterization of copia-like retrotransposons in pear [J].J Jpn Soc Hort Sci,2002,71:723-729.
[24] Kim H,Yamamoto M,Hosaka F,et al.Molecular characterization of novel Ty1-copia-like retrotransposons in pear(Pyruspyrifolia) [J].Tree Genetics & Genomes,2011,7:845-856.
[25] Fonseca S,Hackler L,Zvara,et al.Monitoring gene expression along pear fruit development,ripening and senescence using cDNA microarrays [J].Plant Science,2004,167:457-469.
[26] Voytas D F,Cummings M P,Konieczny A,et al.Copia-like retrotransposons are ubiquitous among plants [J].Proc Natl Acad Sci,1992,89:7124-7128.
[27] Ellis T H N,Poyser S J,Knox M R,et al.Polymorphism of insertion sites of Ty1-copia class retrotransposons and its use for linkage and diversity analysis in pea [J].Molecular Gennetics and Genomics,1998,260:9-19.
[28] Liu Y J,Du Y M,Xu L F,et al.A suppression subtractive hybridization library construction for red skin mutant of ‘Zaosu’ pear and MYBR gene analysis [J].New Zealand Journal of Crop and Horticultural Science,2012,40(2):87-101.
[29] Casacuberta J M,Santiago N.Plant LTR-rotrotransposons and MITES:Control of transposition and impact on the evolution of plant genes and genomes [J].Gene,2003,311(5):1-11.
[30] Pouteau S,Huttner E,Grandbastien M A,et al.Specific expression of the tobacco Tntl retrotransposon in protoplasts [J].EMBO,1991,10(7):1911-1918.
[31] Flavell A J,Smith D,Kumar A.Extreme heterogeneity of Ty1-copia group retrotransposons in plants [J].Molecular Genetics and Genomics,1992,231:233-242.
[32] Beguiristain T,Grandbastien M A,Puigdomènech P,et al.Th-ree Tntl subfamilies show different stress-associated patterns of expression in tobacco,consequences for retrotransposon control and evolution in plants [J].Plant Phvsiol,2001,127:212-221.
