裂殖壶菌OUC88及10个派生菌株18S rDNA基因克隆和分析
2014-03-07臧晓南张学成宋晓金
李 清, 臧晓南, 张学成 , 宋晓金, 杨 青
(1. 中国海洋大学 海洋生命学院, 山东 青岛 266003; 2. 中国科学院 青岛生物能源与过程研究所, 山东 青岛 266101; 3. 青岛科技大学, 山东 青岛 266042)
裂殖壶菌OUC88及10个派生菌株18S rDNA基因克隆和分析
李 清1, 臧晓南1, 张学成1, 宋晓金2, 杨 青3
(1. 中国海洋大学 海洋生命学院, 山东 青岛 266003; 2. 中国科学院 青岛生物能源与过程研究所, 山东 青岛 266101; 3. 青岛科技大学, 山东 青岛 266042)
用PCR方法从裂殖壶菌Schizochytrium limacinumOUC88及以其为出发菌株经紫外诱变筛选的10个菌株中扩增出 18S rDNA 基因序列(1751bp到 1758bp)进行序列测定, 以上序列已登录GenBank(HM042904-HM042914)并与已登录的裂殖壶菌属5条18S rDNA 序列比对分析。结果表明,S. limacinumOUC88以及10个派生菌株间18S rDNA的遗传距离是0.000~0.013, 与Schizochytriumsp. FJU-512 18S rDNA (GenBank No.AY758384)的同源性最高, 为 98%~99%; 与S. limacinum(GenBank No.AB022107)的同源性为96%; 与同属异种S. mangrovei(GenBank No.DQ100293)只有93%的同源性。并运用序列比对分析和 MEGA4.0系统进化树, 结果显示种内诱变产生的细微变异小于同属内不同物种之间的变异。本研究除了为裂殖壶菌这种重要的经济海洋真菌提供分子生物学资料以外, 同时表明18S rDNA序列不仅在分子分类上是一个重要的标志, 也可分析由突变引起的物种内细微的遗传变异。关键词: 裂殖壶菌OUC88; 18S rDNA; 基因克隆; 序列比对分析; 进化树
二十二碳六烯酸(docosahexaenoic acid, DHA)是一种极其重要的 ω-3系列不饱和脂肪酸, 具有抗心血管病、抗癌、抗血脂、促进智力发育和保护视力等重要生理作用[1-5]。DHA的传统来源是海洋鱼油[6],而海洋鱼油的产量和脂肪酸组成随捕捞的季节、产地、鱼种不同而发生改变。鱼油无论是数量还是质量都难以满足人们对DHA的需求。利用破囊壶菌科的海洋真菌生产DHA已成为海洋生物技术的研究热点[7-11]。
裂殖壶菌(Schizochytrium)是属于网粘菌门(Labyrinthulomycota)、网粘菌纲(Labyrinthulomycetes)、破囊壶菌目(Thraustochytriales)、破囊壶菌科(Thraustochytriaceae)的一类类藻的海洋真菌。目前已有5个种被命名, 分别是:S. aggregatum、S. minutum、S. octosporum、S. limacinum和S. mangrovei。其中,S. limacinum从西太平洋沿岸的红树林地区分离得到[12],单细胞, 球形, 细胞内积累了大量的油脂, 总脂肪酸中不饱和脂肪酸含量很高, 主要为 ω-3不饱和脂肪酸—DHA, 而其他的不饱和脂肪酸含量甚微; 且细胞中 90% 以上的油脂以人体易吸收的中性油脂——甘油三脂(TG)的形式存在[13]。因此, 裂殖壶菌成为一种理想的DHA新生资源而备受关注。
裂殖壶菌研究时间不长, 又有重要的经济价值,鉴定及分类是一个重要的问题。海洋真菌传统分类方法只是基于简单的形态学特征, 存在许多争议,制约了相关研究的深入。分子标记技术的发展, 使DNA分子成为区分物种、变种、地理株的有效手段。目前 18S rDNA由于其在一级结构中的多拷贝及高变区域的高度保守等优点而被广泛用于分子系统学研究[14-18], 利用18S rDNA序列变化可以探究生物种间和种内的亲缘关系、系统进化以及鉴定未知的种属等问题, 已成为海洋真菌重要的分子标记[19-25]。在海洋真菌裂殖壶菌属中除了S. octosporum没有得到18S rDNA序列, 其他四个种都得到了一条或多条18S rDNA序列。利用这些序列信息可以确定一些新发现菌株的分类地位, 以及与其他菌种的亲缘关系和变异情况。
目前的研究报道表明, 在真菌类鉴定中, 用18S rDNA序列分析方法一般只能将菌株鉴定到属, 能否鉴定到种一直没有定论。本实验用S. limacinumOUC88[8-9]及由其连续诱变筛选的 10个生长性状、DHA产量差异较大的突变菌株为材料, 克隆扩增18S rDNA, 分析比较该基因在突变菌株之间、突变菌株与出发菌株之间的异同。因为基因突变是产生新生物基因的根本来源, 也是产生生物多样性的根本来源。经过诱变筛选, 突变菌株是否突破了种间距离有待于深入研究。同时以裂殖壶菌Schizochytriumsp. FJU-512(GenBank No.AY758384)[22-23],S. limacinum(GenBank No.AB022107)[24],S. mangroveei(GenBank No.DQ100293),S. aggregatum(GenBank No. DQ023617) ,S. minutum(GenBank No.AB022108)[24],破囊壶菌Thraustochytriumsp. FJN-10(GenBank No. AY773276)[25],酿酒酵母Saccharomyces cerevisiaestrain NRRL Y-12632(GenBank No.EU011664)[26]的18S rDNA 序列作为对照, 比较他们之间的遗传距离, 并为18S rDNA序列分析方法在真菌菌株鉴定中的应用提供实验依据。
1 材料和方法
1.1 材料
以S. limacinumOUC88[8-9]为出发菌株, 经紫外诱变筛选得到OUC101、OUC109、OUC166、OUC168、OUC169、OUC174、OUC175、OUC191、OUC192和OUC196等10个派生菌株(表1)。其中OUC 175产量高, 壁薄, DHA含量高; OUC168产量高, 壁厚, DHA含量高, 沉降好; OUC 101, OUC109, OUC191, OUC192和 OUC196的脂肪酸和 DHA含量介于OUC88和OUC168, OUC175之间; OUC166, OUC169和OUC174的脂肪酸和DHA含量相对较低。以上材料都由本实验室提供。
E. coliDH 5α, PCR Amplification Kit和pMD-18-T Simple Vector购自 TaKaRa公司, 琼脂糖凝胶DNA 回收试剂盒购自天根公司; PCR扩增采用BIO-RAD PCR仪, PCR产物由北京六合华大基因科技股份有限公司测序。
1.2 方法
1.2.1 培养
固体培养基(g/L) : Glucose 60, Agar powder 20, Yeast extract 20, 蒸馏水和海水体积比为1∶1; 发酵培养基 (g/L): Glucose 70, Yeast extract 20, Monosodium glutamate 20, 蒸馏水和海水比为1∶1, 培养温度23℃, 摇瓶转速200 r/min, pH (6.0), 培养时间72 h。

表1 实验菌株的来源、方法和参考文献Tab.1 Source and references of experimental strains
1.2.2 总DNA的提取[8]
采用裂解法(Lysis法)提取总DNA。取适量培养2~3 d的菌体, 液氮研磨后加入10 mL预热的CTAB提取液(0.1 mol/L Tris-Cl pH 8.0, 0.02 mol/L EDTA, 0.5 mol/L NaCl, 1.5%SDS), 剧烈混匀, 60℃水浴30~60 min, 酚/氯仿去蛋白, 乙醇沉淀, 加 TE溶解沉淀, RNaseA去RNA, 电泳检测后-20℃保存备用。
1.2.3 18S rDNA的扩增和测序
采用TaKaRa PCR Amplification Kit, 按操作说明进行PCR, 扩增引物为18SF: 5′CCAACCTGGTTGATCCTGCCAGTA 3′和18SR: 5′CCTTGTTACGACTTCACCTTCCTCT 3′[17], 扩增参数为: 94℃预变性5 min; 94℃变性30 s, 52℃复性30 s, 72℃延伸2 min, 30个循环; 72℃充分延伸10 min, 4℃保存。PCR扩增产物采用天根公司的琼脂糖凝胶 DNA回收试剂盒回收,连接体系依据 TaKaRa连接试剂盒 pMD18-T Simple Vector的使用说明进行。连接产物转化E.coliDH 5α感受态细胞, 用通用引物M13筛选阳性重组子, 阳性克隆送北京六合华大基因科技股份有限公司测序。
1.2.4 序列分析和系统树的构建
将测序得到的基因序列, 用NCBI 网站上Blast软件进行序列比对。然后采用MEGA4.0软件将其与GenBank数据库中已报道的相似序列进行比对并采用邻接法 (neighbor-joining method) 获得系统树,重复计算1000次得到系统树拓扑结构的支持。
1.3 对比序列
为了比较, 还从 Genbank数据库及参考文献得到裂殖壶菌属中5条18S rDNA基因序列, 即Schizo
chytriumsp. FJU-512(GenBank No.AY758384)[22-23],S. limacinum(GenBank No.AB022107)[24],S. mangrovei(GenBank No.DQ100293),S. aggregatum(GenBank No.DQ023617),S. minutum(GenBank No.AB022108)[24],以及作为群外参照的破囊壶菌Thraustochytriumsp. FJN-10(GenBank No.AY773276)[25]和 酿 酒 酵 母Saccharomyces cerevisiaestrain NRRL Y-12632(Gen-Bank No.EU011664)[26]的18S rDNA 序列各一条。这些资料也列于表1中。
2 实验结果
2. 1 18S rDNA基因PCR扩增
取Lysis法提取的11株裂殖壶菌的总DNA为模板进行 PCR扩增, 扩增产物通过 1%的琼脂糖凝胶(EB染色)电泳检测, 结果如图1, 条带清晰且没有杂带, 长度约1 750bp。将片段回收并转化到大肠杆菌DH5α感受态细胞中, 筛选阳性克隆测序。

图1 18S rDNA电泳图像Fig. 1 Electrophoresis patterns of 18S rDNA
2.2 18S rDNA基因序列分析
经DNAMAN整理后得到11株裂殖壶菌完整的18S rDNA基因序列, 序列大小在1 751bp到1 758bp,将其提交到GenBank数据库中得到GenBank 号如表2所示。经Blast同源性比对, 它们与Schizochytriumsp. FJU-512(GenBank No.AY758384, 破囊壶菌科, 裂殖壶菌属)[22-23]同源性最高, 在 98%~99%; 与S.limacinum(GenBank No. AB022107)[24]具有96%的同源性;与S. mangrovei(GenBank No.DQ100293)、S. minutum(GenBank No.AB022108)[24]分别具有93%和86%的同源性; 与S. aggregatum(GenBank No.DQ023617)具有 82%~85%同源性; 与Thraustochytriumsp. FJN-10(GenBank No.AY773276, 破囊壶菌科, 破囊壶菌属)[25]具有91%的同源性; 与Saccharomyces cerevisiaestrain NRRL Y-12632(GenBank No. EU011664,酵母菌科, 酵母菌属, 酿酒酵母菌)[26]具有 81%~84%的同源性, 如表2所示。
将S. limacinumOUC88等11株裂殖壶菌18S rDNA基因序列与Genbank数据库中已知的Schizochytriumsp. FJU-512、S. limacinum(GenBank No.AB022107)[24]、S. mangrovei(GenBank No.DQ100293)、S. aggregatum(GenBank No.DQ023617)、S. minutum(GenBank No. AB022108)[24]、Thraustochytriumsp. FJN-10和Saccharomyces cerevisiaestrain NRRL Y-12632的18S rDNA基因序列进行比对, 利用 MEGA4.0 做出了它们的遗传距离表,如表3。
表 3显示 11株裂殖壶菌的 18S rDNA序列与Schizochytriumsp. FJU-512的遗传距离在0.001~0.010之间; 与S. limacinum的遗传距离为 0.019~0.026; 与S. mangrovei的遗传距离为0.046~0.052; 与S. aggregatum的遗传距离为0.239~0.241; 与S. minutum的遗传距离为0.212~0.216; 与Thraustochytriumsp. FJN-10间的遗传距离为0.072~0.077; 与Saccharomyces cerevisiaestrain NRRL Y-12632的遗传距离为0.265~0.272。
11株裂殖壶菌之间也存在差异, 其中 OUC88与OUC174、OUC192、OUC101、OUC196的遗传距离是0.007, 与OUC166的遗传距离是0.008, 与OUC168的遗传距离是0.010, 与OUC109、OUC191的遗传距离是0.012, 与OUC169、OUC175的遗传距离是0.013。
2.3 分子系统树的构建
基于所得到的数据, 以酿酒酵母菌Saccharomyces cerevisiaestrain NRRL Y-12632为外类群进行系统发育分析。应用NJ 法(Kimura 2-parameter) 构建了分子系统发育关系树(NJ 树, 图2), “Bootstrap”进行1 000次以检验分枝的置信度。枝上数字为1 000 次bootstrap分析的支持百分率, 其数值越高, 支持率越高, 树的置信度越高。
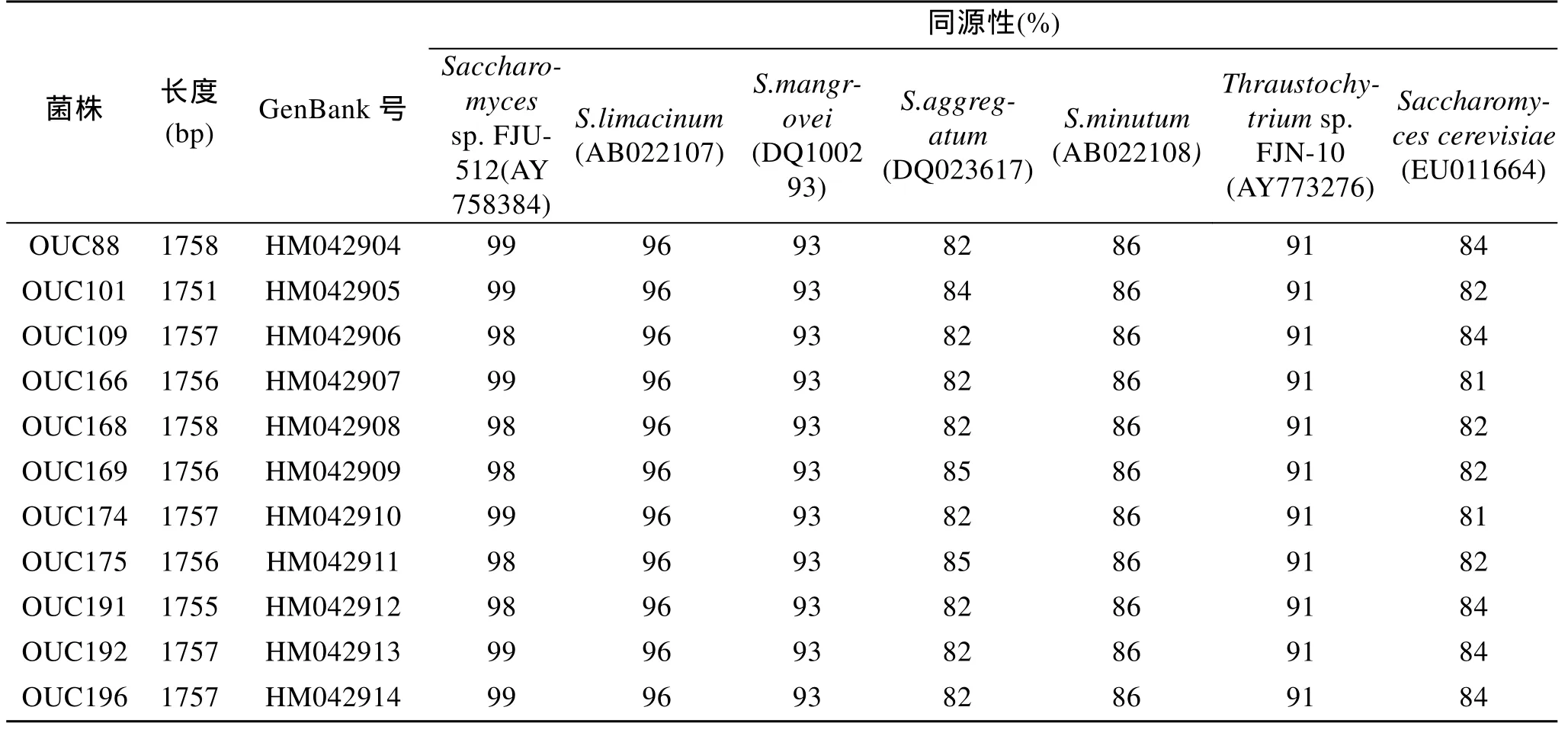
表2 11株裂殖壶菌的18S rDNA序列长度, GenBank号以及同源性比较Tab. 2 The length, GenBank No. and Max. identity of 18S rDNA gene from S .limacinum
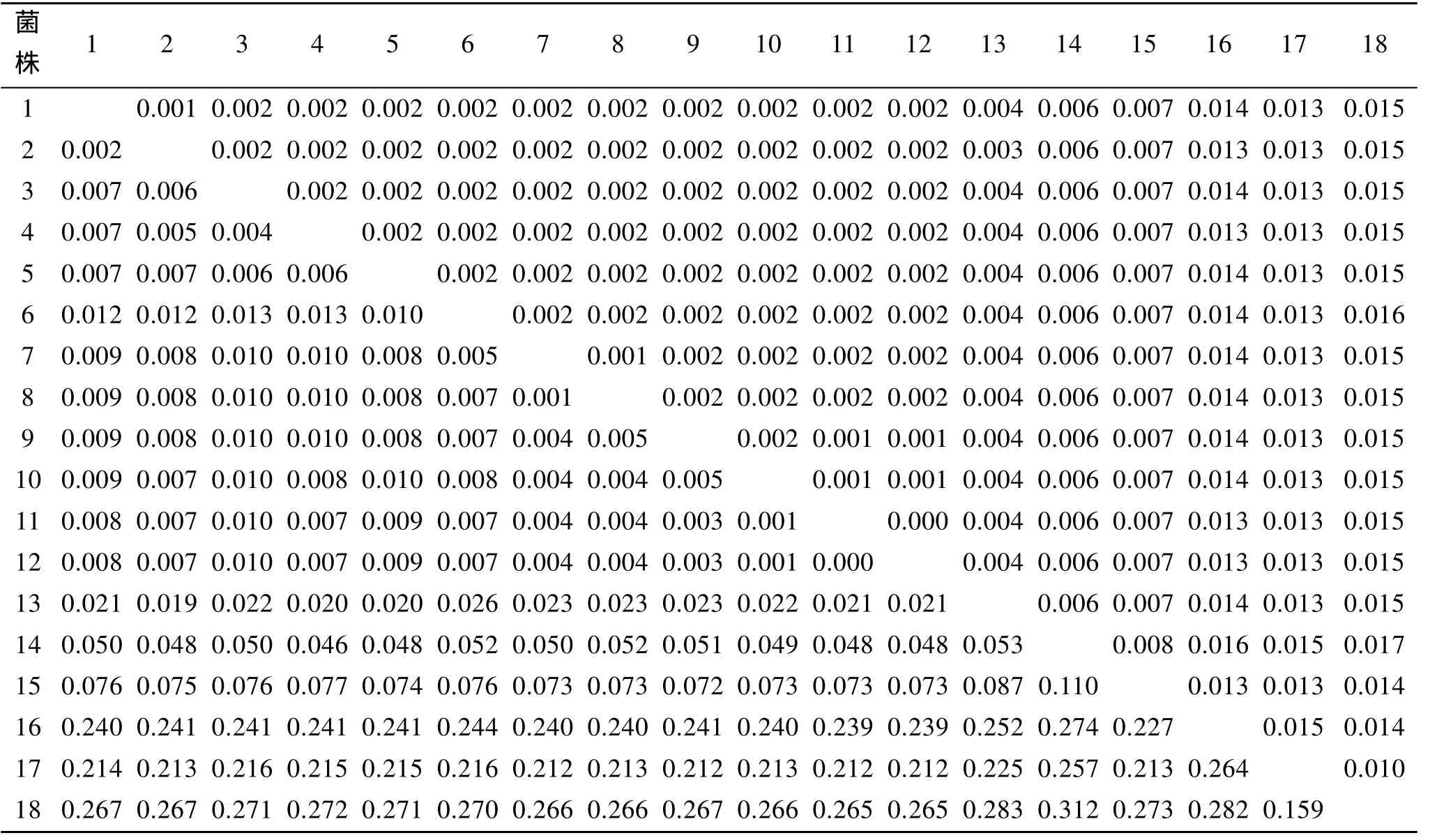
表3 18S rDNA遗传距离 (左下角为遗传距离, 右上角为标准误)Tab.3 Genetic distance (lower-left) and SE (upper-right) of 18S rDNA using Kimura-2-parameter
由图 2看出, 酿酒酵母菌Saccharomyces cerevisiaestrain NRRL Y-12632作为外类群位于进化树的底部,S. minutum与Saccharomyces cerevisiae聚为一支,S. limacinum与S. mangrovei聚为一支。S. limacinumOUC88及由其紫外诱变得到的10个派生菌株以 100%的置信度聚为一支。其中 OUC88与OUC174、OUC192、OUC101、OUC196、OUC166; OUC109、OUC191、OUC169、OUC175与 OUC168聚为一支。OUC101与Schizochytriumsp. FJU-512聚为一支。根据黄建忠[23]中结果Schizochytriumsp. FJU-512与S.limacinum具有大于98 %同源性, 在进化树中与S.limacinum距离最近,进化关系最密切。这一结果在本实验中也得到了进一步的证明, 可知Schizochytriumsp. FJU-512 应该属于S.limacinum。11株裂殖壶菌与Schizochytriumsp. FJU-512聚为一支, 与S. limacinum亲缘关系最近, 与S. mangrovei次之, 与S. aggregatum和S. minutum最远。

图2 18S rDNA的分子系统树(邻接树)Fig.2 Molecular phylogenetic tree of 18S rDNA (Neighbor-Joining tree)
3 分析与讨论
(1) 本实验测定了 11株裂殖壶菌完整的18SrDNA基因序列, 序列大小在1751bp到1758bp。提交到GenBank数据库中得到GenBank 登录号。这为裂殖壶菌属18S rDNA序列增添了新的信息内容,为其他菌株 18S rDNA序列比对和鉴定提供了更多的选择, 扩展了该属的分子系统学资料。
(2) 分析种内遗传差异, 并与种间差异进行量化比较
现代生物进化理论中, 突变、重组、自然选择及隔离是物种形成和生物进化的基础, 其中突变为生物进化提供大量的原材料,也是生物变异的根本来源。基因突变是指DNA分子结构的改变, 即基因内部脱氧核苷酸的排列顺序发生改变。根据突变发生的条件可分为自然突变和诱发突变两类。而紫外诱变就是一种应用广泛、效果明显的诱变方法。它的诱变频率高, 而且不易回复突变, 是微生物育种中最常用和有效的诱变方法之一。
在本实验中,S. limacinumOUC88是Schizochytrium limacinumSR21的紫外突变株, 其他10个菌株由OUC88紫外诱变分离单菌落得到, 它们经过紫外诱变后生物性状(生物量, 脂肪酸含量和 DHA含量等)都发生了很大改变。根据海洋真菌重要的分子标记18S rDNA序列, 分析由于突变形成的种内遗传差异, 并与种间差异进行了量化比较。
以S. limacinumOUC88为出发菌株, 经过紫外诱变得到 10个突变菌株, 比较出发菌株和突变菌株的18S rDNA序列, 显示它们之间的遗传距离为0.000到0.013, 远 小 于 与S. limacinum的 遗 传 距 离0.019~0.026。其中 OUC88与 OUC174、OUC192、OUC101、OUC196的遗传距离是0.007, 与OUC166的遗传距离是0.008, 它们以 91%置信度聚为了一支;而OUC88与OUC109、OUC191的遗传距离是0.012,与OUC169、OUC175的遗传距离是0.013, 与OUC168的遗传距离是 0.010, 这 5个菌株聚为一支。其中OUC174与OUC192遗传距离为0.000, 两个菌株亲缘关系最近; OUC101与Schizochytriumsp FJU-512遗传距离为0.002, 聚为一支。
从遗传距离上看, 11株裂殖壶菌的18S rDNA序列与S. limacinum的遗传距离为0.019~0.026, 与S. mangrovei的遗传距离为0.046~0.052, 小于S. limacinum、S. mangrove两种间距离0.053; 与S. aggregatum的遗传距离为 0.239~0.241, 小于种间距离0.252; 与S. minutum的遗传距离为0.212~0.216, 小于种间距离0.225。由分子系统树看, 11株裂殖壶菌与S. limacinum亲缘关系最近, 同源性比较也高达96%; 与S. aggregatum的亲缘关系比S. minutum近,这与同源性比较和遗传距离显示的结果矛盾, 分析原因可能是在GenBank数据库中S. aggregatum18S rDNA含有100 多个未知碱基, 实际上11株裂殖壶菌与S. aggregatum的遗传距离应小于0.244, 同源性比较应该高于82%~85%同源性。
S. minutum与Saccharomyces cerevisiae遗传距离为0.159, 在进化树中与Saccharomyces cerevisiae聚为一支, 但是它们分属于不同的物种, 形态、生理、代谢等方面差异大, 所以18S rDNA分子标记技术与传统的分类学方法相结合才能更好的判定亲缘关系。
利用 16S rDNA序列作为分子标记时, 其中99%~100%全序列相似的判定为一个种, 97%~99%全序列相似的判定为一个属[27-28]。18S rRN A序列作为划分科级阶元的工具时, 当位于同一分支上互成姐妹群的类群间的遗传距离超过 1% 时, 这几个类群属于不同的科[29]。虽然有文献报道, 在真菌类鉴定中,用 18S rDNA序列分析方法鉴定菌株一般只能鉴定到属[29-30], 但是本实验结果表明出发菌株与10个突变菌株 18S rDNA 序列遗传距离虽然有一定差距,但是变化没有超过S. limacinum种间的尺度, 仍然属于S. limacinum。本文结果表明18S rDNA序列不仅在分子分类上是一个重要的标志, 也可分析由突变引起的物种内细微的遗传变异。
(3) 诱变育种或转基因等基因工程育种可能带来生物基因的改变, 进而影响其形态和生物特性,创造人类需要的变异类型, 这种变化是否可以脱离物种的范围, 创造新物种还是一个有争议的问题。本研究结果表明裂殖壶菌经诱变选育获得的10个突变菌株, 虽然在形态和性状上与出发菌株有很大变化,但是经 18S rDNA序列分析发现他们的遗传距离变化很小, 仍没有超出物种的范围, 因此我们认为基因工程育种可能带来生物基因的改变, 但尚没有证据表明可以创造新物种, 因为新物种的形成, 需要这些进化材料的长期积累和隔离的过程[31]。本文结果进一步表明18S rDNA序列鉴定可作为诱变、转基因等基因工程育种后物种鉴定的有效方式。
[1] Nordoy A, Marchioli R, Arnesen H, et al. n-3 polyunsaturated fatty acid and cardiovascular disease [J] . Lipids, 2001, 36: 127-129.
[2] 曹景玉, 吴力群, 郭卫东, 等. ω-3多不饱和脂肪酸对肝癌细胞生长抑制的作用及机制[J]. 中国普通外科杂志, 2009, 18 (2): 150-155.
[3] 邹祖全, 张晓宏, 张莉娜, 等. 血清对 DHA 抑制肺癌 A549 细胞生长的影响及 DHA 的抗癌机制[J].营养学报, 2009, 31 (4) : 322-329.
[4] 朱丽娜, 张志国, 张敏, 等. DHA的生理功能及其在食品中的稳定性[J]. 中国乳品工业, 2009, 37(2): 45-48.
[5] 黄明发, 吴桂苹, 焦必宁. 二十二碳六烯酸和二十碳五烯酸的生理功能[J]. 食品与药品, 2007, 9(2): 69-71.
[6] Sijtsma L, de Swaaf M E. Biotechnological production and applications of the ω-3 polyunsaturated fatty acid docosahexaenoic acid[J]. Applied Microbiology and Biotechnology, 2004, 64(2): 146-153.
[7] Tzovenis I, de Panw N, Sorgeloos P. Optimization of T-ISO biomass production rich in essential acids, II. Effect of different light regimes on the production of fatty acids[J]. Aquaculture, 2003, 216(1-4): 223-242.
[8] 朱路英, 张学成. 环境条件对裂殖壶菌生长和脂肪酸组成的影响及鲨烯合酶基因和上游调控区序列的克隆与分析[D]. 青岛: 中国海洋大学, 2006.
[9] 朱路英, 张学成, 宋晓金, 等. 碳、氮源浓度和培养时间对裂殖壶菌生长和脂肪酸组成的影响[J]. 中国海洋大学学报, 2007, 37 (2) : 293-298.
[10] 宋晓金, 张学成. 富含 DHA的裂殖壶菌的工业化生产试验、脂肪酸提取及应用研究[D]. 青岛: 中国海洋大学, 2008.
[11] 江贤章, 秦丽娜, 田宝玉, 等. 海洋破囊壶菌Thraustochytriumsp. FJN-10 DHA生物合成途径相关延长酶的克隆与表达[J]. 微生物学报, 2008, 48(2): 176-183.
[12] Nakahara T, Yokochi T, Higashihara T, et al. Production of docosahexa-enoic and docosapentaenoic acids bySchizochytriumsp. Isolated from Yap Islands [J]. Journal of the American Oil Chemists′ Society, 1996, 73: 1421-1426.
[13] Yaguchi T, Tanaka S, Yokochi T, et al. Production of high yields of docosahexaenoic acids bySchizochytriumsp. strain SR21 [J]. Journal of the American Oil Chemists′ Society, 1997, 74: 1431-1434.
[14] Yamaguchi S, Endo K. Molecular phylogeny of Ostracoda(Crustacea) inferred from 18S ribosomal DNA sequences:implication for its origin and diversification[J]. Marine Biology, 2003, 143(1): 23-28.
[15] Iwamoto S, Tokumasu S, Suyama Y,et al. Molecular phylogeny of four selected species of the stric–tly amorphic genus Thysanophora using nuclear ribosomal DNA sequences[J]. Mycoscience, 2002,4(2): 169-180.
[16] 陈丽芬, 骆章群, 骆育敏, 等. 18S rDNA序列分析鉴定棕囊藻香港株 P2为球形棕囊藻[J]. 生态科学. 2003, 22(4): 349-350.
[17] 王宝庆, 葛菁萍, 平文祥. 18S rRNA在系统进化研究中的应用[J]. 黑龙江医药. 2005, 18(4): 251-252.
[18] 张耀东, 沈颂东,林阿朋, 等. 用18S rRNA序列探讨8种绿藻分子系统发育[J]. 大连水产学院学报. 2009, 28(1): 76-78.
[19] Mo C Q, Douck J, Rinkevich B. Development of a PCR strategy for thraustochytrid identification based on 18S rDNA sequence[J]. Mar Biol, 2002, 140(5): 883-889.
[20] Mo C Q, Baruch R A. Simple, Reliable, and Fast Protocol for Thraustochytrid DNA Extraction[J]. Marine Biotechnology, 2001, 3(2): 100-102.
[21] Silva-Pinhati A C O, Bacci M Jr, Hinkle G, et al. Low variation in ribosomal DNA and internal transcribed spacers of the symbiotic fungi of leaf-cuttingants.Brazilian[J]. Journal of Medical and Biological research. 2004, 37(10): 1463-1472.
[22] 黄建忠, 江贤章. DHA 高产菌Schizochytriumsp.FJU-512的分离及其18S rRNA基因序列比较分析[J]. 应用与环境生物学报, 2005, 11(2): 202-207.
[23] 黄建忠, 江贤章. DHA 高产菌 Schizochytrium sp.FJU-512脂肪酸组成与18S rRNA基因序列比较分析[J]. 工业微生物. 2004, 34(4): 6-12.
[24] Honda D, Yokochi T, Nakahara T, et al. Molecular phylogeny of labyrinthulids and thraustochytrids based on the sequencing of 18S ribosomal RNA gene[J]. Eukaryot Microbiol, 1999, 46 (6) : 637-647.
[25] 黄建忠, 刘丽霞, 吴松刚. 高产 DHA 海洋真菌Thraustochytriumsp. FJN-10脂肪酸的组型及其 18S rDNA序列分析[J]. 药物生物技术, 2005, 12(1): 1-5.
[26] Kurtzman C P, Robnett C J. Systematics of methanol assimilating yeasts and neighboring taxa from multigene sequence analysis and the proposal ofPeterozyma gen.nov., a new member of the Saccharomycetales [J]. FEMS Yeast Res. 2010, 10 (3), 353-361.
[27] Drancourt M, Bollet C, Carlioz R, et al. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifable bacterial isolates[J]. Journal of Clinical Microbiology, 2000, 38: 3623-3630.
[28] JandaJ M, Abbott S L. Bacterial identifcation for publication: When is enough? [J]. Journal of Clinical Microbiology, 2002 , 40: 1887-1891.
[29] 汪晓阳, 周志军, 黄原, 等. 基于 18S rRNA基因序列的直翅目主要类群系统发育关系研究[J]. 动物分类学报, 2011, 36 ( 3): 627- 638.
[30] 张 霞, 武志芳, 张胜潮, 等. 贵州浓香型白酒大曲中霉菌的18S rDNA系统发育分析[J]. 应用与环境生物学报, 2011, 17 ( 3 ): 334-337.
[31] 李难. 生物进化论[M]. 北京. 高等教育出版社, 1982.
(本文编辑: 张培新)
Cloning and analysis of 18S rDNA gene fromSchizochytrium limacinumOUC88 and 10 derived strains
LI Qing1, ZANG Xiao-nan1, ZHANG Xue-cheng1, SONG Xiao-jin2, YANG Qing3
(1. College of Marine Life Sciences, Ocean University of China, Qingdao 266003, China; 2. Qingdao Institute of Biomass Energy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao 266101, China; 3. Qingdao University of Science and Technology, Qingdao 266042, China)
Oct.,16,2012
SchizochytriumlimacinumOUC88; 18S rDNA; gene cloning; sequence analysis; phylogenetic
18S rDNA genes with length between 1751bp and 1758bp (GenBank : HM042904-HM042914) were cloned fromSchizochytrium limacinumOUC88 and its 10 derived strains by PCR. By sequencing and blasting with other 18S rDNA genes ofSchizochytriumfrom GenBank database, the genetic distances ofS. limacinumOUC88 and its derived strains were between zero and 0.013, and their identity was between 98% and 99% withSchizochytriumsp FJU-512 (GenBank No.AY758384), 96% withS. limacinum(GenBank No. AB022107) and 93% withS. mangrovei(GenBank No.DQ100293), respectively. By sequence analysis and the phylogenetic tree built up by MEGA4.0, the results showed that slight variation in mutation was less than in different species. These have provided molecular biology data for this important economic marine fungi. Furthmore, the results showed that the 18S rDNA sequence was not only an important sign in molecular classification, but also can be used to analyze slight genetic variation caused by mutation.
Q75
A
1000-3096(2014)01-0071-08
10.11759/hykx20130112003
2012-10-16;
2013-01-03
国家863计划项目(2008AA09Z410); 国家科技支撑计划项目(2006 BAD09A12)
李清(1986-), 女, 硕士研究生, 现从事裂殖壶菌脂肪酸合成分子调控研究, E-mail:liqing202020@126.com; 通信作者: 臧晓南, 女,副教授, 硕士生导师, E-mail: xnzang@ouc.edu.cn. 电话: 0532-82032789
