组蛋白去甲基化酶家族中组蛋白去甲基化酶4作用机制及其应用的研究进展
2014-02-16AndreanaHOLOWATYJRoselyneLABBEZengquanYANG
叶 覃,Andreana HOLOWATYJ,Roselyne M.LABBE,刘 辉,Zengquan YANG
组蛋白去甲基化酶家族中组蛋白去甲基化酶4作用机制及其应用的研究进展
叶 覃,Andreana HOLOWATYJ,Roselyne M.LABBE,刘 辉,Zengquan YANG
组蛋白甲基化是一种重要的表观遗传性修饰方式,是一个可逆的动态调节过程。组蛋白去甲基化酶家族中组蛋白去甲基化酶4能催化去除组蛋白赖氨酸残基甲基标记,调节染色质的结构,参与精细调控基因转录,维持染色质的活性和非活性平衡。组蛋白去甲基化酶4异常可能导致细胞增殖、分化、个体发育、能量代谢及肿瘤发生发展等多种生物进程异常。研究显示组蛋白去甲基化酶4可作为新的药物靶标。本文就组蛋白去甲基化酶4家族的结构、作用机制、在疾病发生发展进程中的生物学功能及特异性抑制剂开发的最新研究进展作一综述。
组蛋白去甲基化酶;组蛋白去甲基化酶4;肿瘤
核小体是染色质的基本组成单位,由核心组蛋白H2A、H2B、H3、H4各2分子构成的八聚体和缠绕1.75圈的146 bp DNA组成。组蛋白氨基末端可发生乙酰化、甲基化、磷酸化、泛素化等多种修饰,影响染色质结构,调控基因转录,其中组蛋白甲基化是组蛋白主要的修饰形式之一。甲基化组蛋白与不同效应蛋白协同参与调控异染色质形成、X染色体失活、特定基因转录调节、基因组完整性及细胞发育等多种生物学效应。组蛋白甲基修饰是甲基化酶和去甲基化酶动态相互作用的结果。最近研究表明组蛋白甲基化异常可导致基因错误表达,引起发育异常、代谢紊乱、疾病甚至肿瘤的发生[1-2]。本文就组蛋白去甲基化酶4(lysine-specific demethylase 4,KDM4)家族的结构、作用机制、在疾病发生发展进程中的生物学功能及特异性抑制剂开发的最新研究进展作一综述。
1 组蛋白去甲基化酶
组蛋白去甲基化酶作用位点主要为组蛋白H3上的赖氨酸(K)残基,包括H3上的K4、K9、K27和K36。通常H3K4、H3K36位点甲基化修饰介导基因转录活化,H3K9、H3K27的甲基化修饰则介导转录抑制。目前已知有两大类组蛋白赖氨酸去甲基化酶家族:一类属于氨基酸氧化酶家族,包括赖氨酸特异性组蛋白去甲基化酶1(lysine-specific demethylase 1,LSD1/KDM1A)和LSD2(KDM1B)[3];另一大类是含Jumonji C(JmjC)结构域的组蛋白去甲基化酶。这类去甲基化酶需要二价铁离子(Fe2+)和α-酮戊二酸为辅助因子,通过羟基化作用使组蛋白赖氨酸去甲基。在人类基因组中含有32个JmjC家族成员,其中24个证实有去甲基化酶活性,根据功能和不同的结构域组合又可细分为7个亚家族(KDM2-8)[2,4-6]。
2 KDM 4的结构和正常功能
KDM4是最大的JmjC去甲基化酶亚家族之一,在人类基因组中含有6个KDM4基因(KDM4A-F)。KDM4A-C基因编码的蛋白相对分子质量大约130× 103大小,分别含有1个JmjN和1个JmjC功能域,2个PHD和2个Tudor功能域;而KDM4D和KDM4E编码的蛋白缺少PHD和Tudor功能域,大小只有前3个成员的一半(图1)[7-8]。其中KDM4E最近才被证实是一个功能基因,和KDM4E结构类似的KDM4F是个假基因[2]。KDM4A-C能催化去除H3K9和H3K36三甲基和二甲基(me3/me2)标记,而KDM4D只能催化去除H3K9me3/me2标记,KDM4E则可催化H3K9me3和H3K56me3甲基基团的去除。KDM4各个成员在机体不同组织中表达水平不同:KDM4A-C在正常人体组织中广泛表达,在脾脏、卵巢及结肠有高表达;KDM4D和KDM4F在睾丸中高表达,在其他组织则较低或几乎无表达[2,9]。
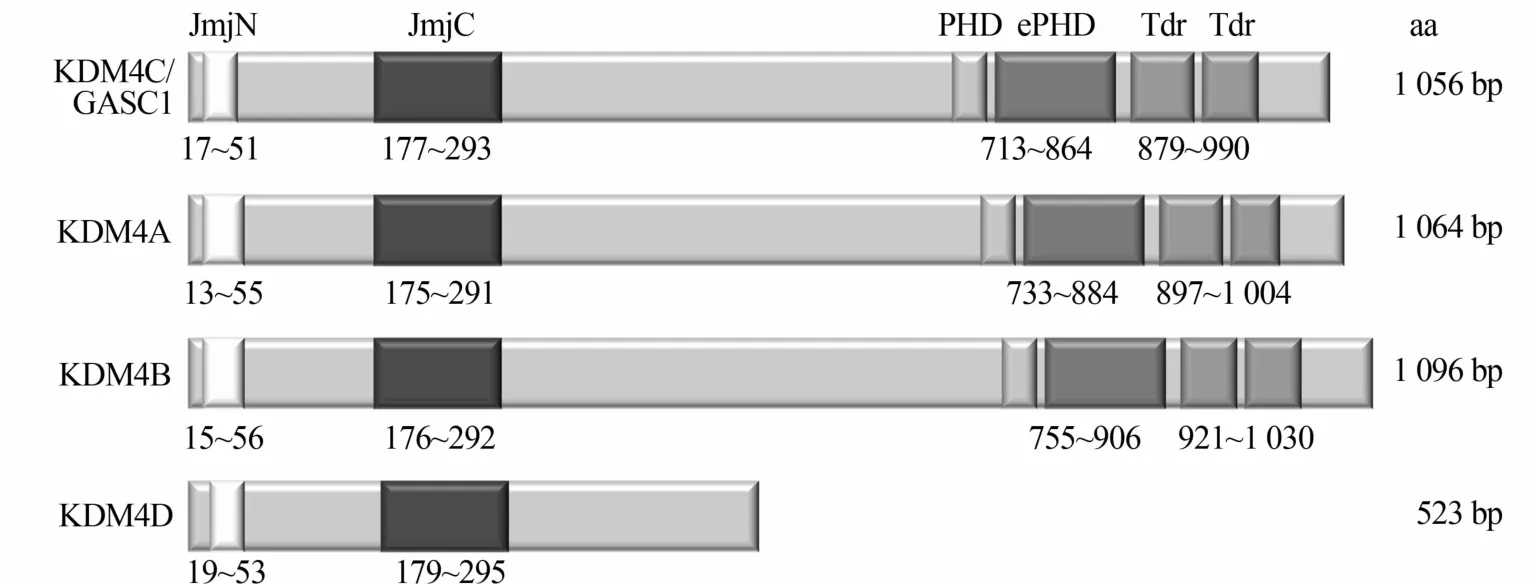
图1 KDM4A-D的蛋白结构
KDM4参与调控正常发育过程,KDM4的双纯合子敲除果蝇个体在二龄幼虫期就死亡[10];KDM4杂合子敲除果蝇DNA对紫外光照射则更加敏感[11];敲除KDM4基因的线虫出现生殖细胞凋亡以及DNA复制速度降低[12];条件敲除心脏特异KDM4A转基因小鼠会促进心肌肥厚性刺激的反应[13];乳腺上皮细胞敲除KDM4B会延迟乳腺发育[14]。KDM4还参与发育过程中维持胚胎干细胞中染色体结构开放状态;诱导Oct4(POU class 5 homeobox 1)、Sox2(sex determining region Y-box)2和Myc(v-myc avian myelocytomatosis viral oncogene homolog)等多能性因子的表达,调控胚胎发育过程中细胞分化的类型等[15-16]。另外,最新研究发现KDM4B和KDM4C在胚胎干细胞的自我更新和诱导多能干细胞的产生中具有各自独特的,但又相互配合的重要作用[17]。
3 KDM 4与疾病
自2006年KDM4被证明以来,研究人员对KDM4的活性进行了充分地鉴定,并发现KDM4在肿瘤及其他疾病的发生发展过程中可发挥重要的作用。下面就KDM4家族各成员与疾病特别是在肿瘤发生发展中的作用作一简述。
3.1 KDM4A 许多研究表明KDM4A在多种肿瘤中都有异常扩增或高表达,并可促进肿瘤细胞的侵袭性生长和转移[2]。分析1 770个8类肿瘤患者组织样本后,Black等[18]在18.7%的肿瘤中发现KDM4A基因拷贝的异常扩增;在卵巢癌,高达46%样本中有KDM4A的扩增;基因拷贝数变化直接决定KDM4A蛋白表达的增加或减少,而KDM4A过度表达会导致1q12、1q21以及Xq13.1特定位点基因拷贝数增加,但并不会引起全基因组染色体不稳定。Black的研究成果揭示组蛋白去甲基化酶异常表达可诱导包含原癌基因特定基因位点的拷贝数增加,为肿瘤细胞基因拷贝数变异提供一新的的分子机制。Ding等[19]发现KDM4A可改变转录因子激活蛋白(activator protein 1,AP-1)的分子激活过程,对鳞状细胞癌的侵袭性生长和转移起重要促进作用。机制研究表明,KDM4A的组蛋白去甲基化作用促进了AP-1复合物结合细胞内转录因子C-JUN和FOSL1(FOS like antigen 1)启动子,从而促进AP1激活的一个重要正反馈环。在人类鳞状细胞癌组织样本中,研究人员证实淋巴结转移灶中的KDM4A表达升高。在小鼠模型中,下调/敲除KDM4A的表达可显著抑制鳞状细胞癌侵袭和扩散。因此,KDM4A可能为抑制鳞状细胞癌侵袭性生长和转移提供了一个新的的治疗靶点。
3.2 KDM4B KDM4B结构、底物特异性和体外酶活性都与KDM4A非常相似,而且功能密切相关[2]。研究显示KDM4B异常表达不仅与肿瘤发生发展以及恶性程度密切相关,还参与肿瘤细胞缺氧应激反应[20-21]。KDM4B作为雌激素受体(estrogen receptor,ER)靶向基因,能与ER形成一个功能复合体,调控ER靶向相关基因的转录活性。因此,KDM4B表达下调会抑制ER阳性乳腺癌细胞系MCF7或T47D细胞增殖和体外成瘤体积,但是ER阴性细胞系MDA-MB-231细胞增殖不受KDM4B表达水平影响[14,22]。与此相对应,KDM4B异常高表达在ER阳性乳腺癌更为普遍[14]。另外,KDM4B可和H3K4甲基化酶MLL2形成复合体,协同调控ER靶向相关基因的转录活性[23]。最新研究发现,KDM4B能直接调控ER和ER信号级联通路中的关键转录增强子FOXA1(forkhead box A1)的表达。因此,KDM4B可通过ER信号级联通路在乳腺癌的形成和进展中发挥重要作用[22]。在前列腺癌中,雄激素受体(androgen receptor,AR)可调控KDM4B表达,而KDM4B又可和AR结合。不仅利用去甲基化作用增强AR靶向相关基因的转录,而且能通过泛素化修饰抑制AR蛋白降解。因此,KDM4B可与AR形成一个信号级联通路正反馈环,促进前列腺癌的形成和进展[24]。
3.3 KDM4C KDM4C,也称为鳞状细胞癌增强蛋白1抗体(gene amplified in squamous cell carcinoma 1,GASC1),最初是从食管鳞状细胞癌系中克隆出来的一个高度扩增的基因[25]。研究显示,KDM4C在乳腺癌、肺癌、前列腺癌、淋巴癌及髓母细胞瘤等多种肿瘤中都有扩增或异常高表达,并且与肿瘤的恶性程度密切相关[2,26-27]。我们实验室发现,KDM4C扩增在乳腺癌中约占15%,与KDM4B相反,其扩增或异常高表达在高度侵袭性的基底型乳腺癌更为普遍。我们实验室证明了KDM4C高表达可诱导正常乳腺上皮细胞转化和恶性增值。降低KDM4C表达可抑制乳腺癌生长和转移[27-28]。在机制上,KDM4C可通过H3K9去甲基化作用促进原癌基因c-MYC、MDM2(MDM2 oncogene,E3 ubiquitin protein ligase)、NOTCH1或多能性因子Oct4、NANOG的转录,从而诱导细胞转化和促进肿瘤干细胞增殖[16,27,29-30]。另外,KDM4C可与缺氧诱导因子(hypoxia inducible factor 1,HIF1)结合,是乳腺癌生长和转移所需的HIF1的辅激活因子。KDM4C可降低HIF1的靶基因位点上H3K9me3的水平,进而活化包括赖氨酰氧化酶样蛋白2和L1细胞黏附分子的HIF1靶基因,促进乳腺癌的转移[28]。KDM4C可以与雄激素受体AR形成复合体,共同刺激AR和依赖AR生长的前列腺肿瘤细胞增殖,参与前列腺癌肿瘤发生过程[31]。特别的是KDM4C也能够催化非组蛋白Pc2(polycomb 2 protein)去甲基化,激活细胞生长调控基因[32]。
3.4 KDM4D和KDM4E KDM4家族成员KDM4D,没有PHD和Tudor功能域,不能催化H3K36位点去甲基化,但是能催化H3K9me3/me2及H1.4K26me3的去甲基化作用[2]。KDM4D与AR共激活,参与前列腺癌细胞的扩增和杀伤逃逸[33];KDM4D调节肿瘤微环境和免疫细胞生长,如肿瘤坏死因子-α能诱导KDM4D在树突细胞和巨噬细胞中表达参与炎症通路[34];KDM4D能刺激p53依赖基因的表达[35]。有意思的是KDM4D刺激结肠癌细胞增殖和促进AR活性表现出原癌基因的特征,但是激活p53依赖基因的转录却起着抑癌基因作用,在肿瘤形成过程中KDM4D作用具有促癌和抑癌的双面性。KDM4E以前被认为是假基因,没有生物功能,最近才发现具有生物活性,但是尚需要更深入的实验证实[2]。
4 KDM 4抑制剂
KDM4对染色体的结构修饰和基因表达调控发挥着重要的作用,通过抑制KDM4活性,调控组蛋白异常甲基化状态可抑制或杀伤肿瘤细胞。因此,KDM4可以作为肿瘤治疗和药物筛选的新靶点,针对KDM4家族抑制剂的研究也成为药物化学的一个研究热点[36-37]。根据去甲基化酶JmjC结构域的三维结构和它们的催化机制,已有多个KDM4抑制剂的研究开发报道[2,37]。按照作用机制,已报道的抑制剂可分为辅助因子α-酮戊二酸类似物、金属辅因子干扰物及组蛋白底物类似物3类。
4.1 辅助因子α-酮戊二酸类似物 KDM4是α-酮戊二酸和铁离子依赖酶。目前绝大多数KDM4抑制剂是辅助因子α-酮戊二酸类似化合物,其可竞争性结合在铁离子与酶结合的催化位点,抑制JmjC去甲基化酶家族多个成员的去甲基化酶活性[2,37]。Hamada等[38]利用一种α-酮戊二酸类似物氨基化合物乙二酰氨基乙羧酸(N-Oxalyl glycine,NOG)作为药物骨架,设计了多种新型的α-酮戊二酸类似物KDM4抑制剂,如NCDM-32,并发现其对KDM4C抑制剂效率比其他NOG骨架抑制剂对KDM4C活性抑制效率高500倍、特异性高9 100倍。其他α-酮戊二酸类似物有植物生长调节剂diaminozide、2,4-pyridindicarboxylic酸(PDCA)、PDCA衍生物5-carboxy-8-hydroxyquinoline(5-Carboxy-8HQ)和2,2-联吡啶衍生物的4-羧酸盐(Bipyridine A)[2,39]。人体中有70~80个利用α-酮戊二酸的加氧酶,如缺氧诱导因子和脯氨酰羟化酶1-3。因此,α-酮戊二酸类似物靶向特异性差,限制了这类抑制剂在实验或治疗中的应用。
4.2 金属辅因子干扰物 非铁金属离子如镍,或者有机化合物,竞争结合到KDM4上二价铁离子结合位点,可抑制其催化活性[40]。另外,结构和生物信息学分析发现KDM4催化结构域有锌离子的结合位点,其他的组蛋白去甲基化酶家族或者是α-酮戊二酸加氧酶都没有这个位点,并且锌离子的结合位点与KDM4-组蛋白赖氨酸结合位点非常相近。因此,锌离子蛋白结合抑制化合物,如双硫仑(disulfiram)和依布硒(ebselen),可通过移除锌离子,选择性的抑制KDM4催化活性[41]。
4.3 组蛋白甲基赖氨酸类似物 组蛋白甲基赖氨酸类似物能与KDM4去甲基化酶组蛋白底物竞争结合,达到抑制酶活性的作用。底物模拟设计需要考虑到KDM4是多个功能域形成一个特殊的笼装结构,参与组蛋白识别。最近,筛选天然产物的文库中抑制KDM4C活性的研究发现儿茶酚(catechols)可能直接或间接的与去甲基化酶组蛋白底物竞争结合,抑制KDM4C活性[42]。Luo等[43]根据组蛋白去乙酰化酶(distone deacetylase,HDAC)抑制剂MS-275的分子框架,设计出同时包含组蛋白甲基赖氨酸类似物和α-酮戊二酸辅助因子类似物的双结构去甲基化酶抑制剂。这样的设计既能降低辅助因子类似物对非靶向性加氧酶抑制,提高抑制剂的特异性;同时,又能抑制去甲基化酶与底物结合,抑制酶活性。体外实验证明这种化合物能抑制KDM4A、KDM4C和KDM4E的活性。更为重要的是其药物前体Methylstat可抑制KDM4C扩增的食管鳞状细胞癌KYSE150细胞系的恶性增值[43]。
最近,Wang等[44]筛选抑制肿瘤细胞生长的化合物,发现了一种与上述抑制剂结构不同的去甲基化酶抑制剂JIB-04。生化分析,JIB-04可有效抑制KDM4成员,包括KDM4A、KDM4B、KDM4C和KDM4E的催化活性。JIB-04不是辅助因子α-酮戊二酸类似物,并且确切的分子机制尚不清楚。然而,JIB-04似乎不影响其他α-酮戊二酸依赖性酶的功能,也不影响正常细胞的生长;但可抑制肿瘤细胞去甲基化酶的功能,抑制肿瘤细胞生长。正因为如此,这种抑制剂可能代表一种新的表观遗传学药物类型,用于开发新的抗肿瘤药物。
5 展望
在组蛋白甲基转移酶和去甲基化酶的协同调控下,细胞中组蛋白甲基化和去甲基化的过程处于动态平衡。KDM4与多能性因子、核激素受体等相互结合精确调控基因表达及其他与染色质有关的功能,参与胚胎发育及肿瘤发生等多个生物进程。自2006年发现KDM4以来,已对其生物学功能及在疾病,特别是在肿瘤发生发展进程中的作用进行了多方面研究。很显然,我们对KDM4的分子作用机制仍然知之甚少。例如,KDM4A、KDM4B和KDM4C的结构和功能团非常相似,其均可催化H3K9和H3K36的去甲基化。越来越多的研究发现在不同的细胞类型中三者可能既有共同的,又具有各自独特的生物功能。然而,我们对KDM4A、KDM4B和KDM4C在不同的细胞类型及疾病中的生物学作用仍知之不多。再者,KDM4应与转录因子及其他的蛋白质形成复合体,然后被招募到特定基因位点,调控特定的下游基因,发挥其生物学作用。我们对KDM4复合体组成及在生理和病理状态的作用和分子机制仍不清楚。因此,深入了解KDM4各成员在生理和病理状态下的生物学作用和分子机制将是未来研究的一个重要组成部分。考虑到KDM4在多种肿瘤中都有异常扩增或高表达,并可促进肿瘤细胞的生长和转移,KDM4抑制剂有希望成为一种新型抗肿瘤药物。然而,需要注意的是目前报道的KDM4抑制剂特异性不高,因此可以预料KDM4研究的下一个十年将专注于开发具有高特异性KDM4抑制剂。这类抑制剂分子将为KDM4在肿瘤发生发展进程中的分子机制研究提供一个新的工具,并有希望成为能用于临床的抗肿瘤药物。
[1]Black JC,Van Rechem C,Whetstine JR.Histone lysine methylation dynamics:establishment,regulation,and biological impact[J].Mol Cell,2012,48(4):491-507.
[2]LabbéRM,Holowatyj A,Yang ZQ.Histone lysine demethylase(KDM)subfamily 4:structures,functions and therapeutic potential[J].Am JTransl Res,2013,6(1):1-15.
[3]Shi Y,Lan F,Matson C,etal.Histone demethylation mediated by the nuclear amine oxidase homolog LSD1[J]. Cell,2004,119(7):941-953.
[4]Klose RJ,Kallin EM,Zhang Y.JmjC-domain-containing proteins and histone demethylation[J].Nat Rev Genet,2006,7(9):715-727.
[5]Klose RJ,Zhang Y.Regulation of histone methylation by demethylimination and demethylation[J].Nat Rev Mol Cell Biol,2007,8(4):307-318.
[6]Cloos PA,Christensen J,Agger K,et al.The putative oncogene GASC1 demethylates tri-and dimethylated lysine 9 on histone H3[J].Nature,2006,442(7100):307-311.
[7]Whetstine JR,Nottke A,Lan F,et al.Reversal of histone lysine trimethylation by the JMJD2 family of histone demethylases[J].Cell,2006,125(3):467-481.
[8]Shin S,Janknecht R.Diversity within the JMJD2 histone demethylase family[J].Biochem Biophys Res Commun,2007,353(4):973-977.
[9]Hillringhaus L,Yue WW,Rose NR,et al.Structural and evolutionary basis for the dual substrate selectivity of human KDM4 histone demethylase family[J].JBiol Chem,2011,286(48):41616-41625.
[10]Tsurumi A,Dutta P,Yan SJ,et al.Drosophila Kdm4 demethylases in histone H3 lysine 9 demethylation and ecdysteroid signaling[J].Sci Rep,2013,3:2894.
[11]Palomera-Sanchez Z,Bucio-Mendez A,Valadez-Graham V,et al.Drosophila p53 is required to increase the levels of the dKDM4B demethylase after UV-induced DNA damage to demethylate histone H3 lysine 9[J].JBiol Chem,2010,285(41):31370-31379.
[12]Black JC,Allen A,Van Rechem C,et al.Conserved antagonism between JMJD2A/KDM4A and HP1γduring cell cycle progression[J].Mol Cell,2010,40(5):736-748.
[13]Zhang QJ,Chen HZ,Wang L,et al.The histone trimethyllysine demethylase JMJD2A promotes cardiac hypertrophy in response to hypertrophic stimuli in mice[J].J Clin Invest,2011,121(6):2447-2456.
[14]Kawazu M,Saso K,Tong KI,et al.Histone demethylase JMJD2B functions as a co-factor of estrogen receptor in breast cancer proliferation and mammary gland development[J].PLoSOne,2011,6(3):e17830.
[15]Katoh Y,Katoh M.Comparative integromics on JMJD2A,JMJD2B and JMJD2C:preferential expression of JMJD2C in undifferentiated ES cells[J].Int JMol Med,2007,20(2):269-273.
[16]Loh YH,ZhangW,Chen X,et al.Jm jd1a and Jmjd2c histone H3 Lys 9 demethylases regulate self-renewal in embryonic stem cells[J].Genes Dev,2007,21(20):2545-2557.
[17]Das PP,Shao Z,Beyaz S,et al.Distinct and combinatorial functions of Jmjd2b/Kdm4b and Jmjd2c/Kdm4c inmouse embryonic stem cell identity[J].Mol Cell,2014,53(1):32-48.
[18]Black JC,Manning AL,Van Rechem C,et al.KDM4A lysine demethylase induces site-specific copy gain and rereplication of regions amplified in tumors[J].Cell,2013,154(3):541-555.
[19]Ding X,Pan H,Li J,et al.Epigenetic activation of AP1 promotes squamous cell carcinoma metastasis[J].Sci Signal,2013,6(273):ra28.1-ra28.13.
[20]Kim JG,Yi JM,Park SJ,et al.Histone demethylase JMJD2B-mediated cell proliferation regulated by hypoxia and radiation in gastric cancer cell[J].Biochim Biophys Acta,2012,1819(11/12):1200-1207.
[21]Fu L,Chen L,Yang J,et al.HIF-1α-induced histone demethylase JMJD2B contributes to themalignant phenotype of colorectal cancer cellsvia an epigenetic mechanism[J]. Carcinogenesis,2012,33(9):1664-1673.
[22]Gaughan L,Stockley J,Coffey K,et al.KDM4B is amaster regulator of the estrogen receptor signalling cascade[J]. Nucleic Acids Res,2013,41(14):6892-6904.
[23]Shi L,Sun L,LiQ,et al.Histone demethylase JMJD2B coordinates H3K4/H3K9 methylation and promotes hormonally responsive breast carcinogenesis[J].Proc Natl Acad Sci USA,2011,108(18):7541-7546.
[24]Coffey K,Rogerson L,Ryan-Munden C,et al.The lysine demethylase,KDM4B,is a key molecule in androgen receptor signalling and turnover[J].Nucleic Acids Res,2013,41(8):4433-4446.
[25]Yang ZQ,Imoto I,Pimkhaokham A,et al.A novel amplicon at 9p23-24 in squamous cell carcinoma of the esophagus that lies proximal to GASC1 and harbors NFIB[J].Jpn JCancer Res,2001,92(4):423-428.
[26]Yang ZQ,Imoto I,Fukuda Y,et al.Identification of a novel gene,GASC1,within an amplicon at 9p23-24 frequently detected in esophageal cancer cell lines[J].Cancer Res,2000,60(17):4735-4739.
[27]Liu G,Bollig-Fischer A,Kreike B,et al.Genomic amplification and oncogenic properties of the GASC1 histone demethylase gene in breast cancer[J].Oncogene,2009,28(50):4491-4500.
[28]Luo W,Chang R,Zhong J,et al.Histone demethylase JMJD2C is a coactivator for hypoxia-inducible factor 1 that is required for breast cancer progression[J].Proc Natl Acad Sci USA,2012,109(49):E3367-E3376.
[29]Ishimura A,Terashima M,Kimura H,etal.Jm jd2c histone demethylase enhances the expression of Mdm2 oncogene[J].Biochem Biophys Res Commun,2009,389(2):366-371.
[30]Wang J,Zhang M,Zhang Y,et al.The histone demethylase JMJD2C is stage-specifically expressed in preimplantation mouse embryos and is required for embryonic development[J].Biol Reprod,2010,82(1):105-111.
[31]Wissmann M,Yin N,Müller JM,et al.Cooperative demethylation by JMJD2C and LSD1 promotes androgen receptor dependent gene expression[J].Nat Cell Biol,2007,9(3):347-353.
[32]Yang L,Lin C,Liu W,et al.ncRNA-and Pc2 methylation dependent gene relocation between nuclear structures mediates gene activation programs[J].Cell,2011,147(4):773-788.
[33]Shin S,Janknecht R.Activation of androgen receptor by histone demethylases JMJD2A and JMJD2D[J].Biochem Biophys Res Commun,2007,359(3):742-746.
[34]Zhu Y,van Essen D,Saccani S.Cell-type-specific control of enhancer activity by H3K9 trimethylation[J].Mol Cell,2012,46(4):408-423.
[35]Kim TD,Oh S,Shin S,et al.Regulation of tumor suppressor p53 and HCT116 cell physiology by histone demethylase JMJD2D/KDM4D[J].PloSOne,2012,7(4):e34618.
[36]Helin K,Dhanak D.Chromatin proteins and modifications as drug targets[J].Nature,2013,502(7472):480-488.
[37]Suzuki T,Miyata N.Lysine demethylases inhibitors[J].J Med Chem,2011,54(24):8236-8250.
[38]Hamada S,Suzuki T,Mino K,et al.Design,synthesis,enzyme-inhibitory activity,and effect on human cancer cells of a novel series of jumonji domain-containing protein 2 histone demethylase inhibitors[J].JMed Chem,2010,53(15):5629-5638.
[39]Wang Z,Patel DJ.Smallmolecule epigenetic inhibitors targeted to histone lysine methyltransferases and demethylases[J].Q Rev Biophys,2013,46(4):349-373.
[40]Giri NC,Passantino L,Sun H,et al.Structural investigations of the nickel-induced inhibition of truncated constructs of the JMJD2 family of histone demethylases using X-ray absorption spectroscopy[J].Biochemistry,2013,52(24):4168-4183.
[41]Sekirnik R,Rose NR,Thalhammer A,et al.Inhibition of the histone lysine demethylase JMJD2A by ejection of structural Zn(Ⅱ)[J].Chem Commun(Camb),2009,(42):6376-6378.
[42]Nielsen AL,Kristensen LH,Stephansen KB,et al.Identification of catechols as histone-lysine demethylase inhibitors[J].FEBS Lett,2012,586(8):1190-1194.
[43]Luo X,Liu Y,Kubicek S,et al.A selective inhibitor and probe of the cellular functions of JumonjiC domain-containing histone demethylases[J].JAm Chem Soc,2011,133(24):9451-9456.
[44]Wang L,Chang J,Varghese D,et al.A small molecule modulates Jumonjihistone demethylase activity and selectively inhibits cancer growth[J].Nat Commun,2013,4:2035.
The emerging functions and therapeutic application of the histone lysine demethylase lysine-specific demethylase 4 subfamily
YE Qin1,2,Andreana HOLOWATYJ1,Roselyne M.LABBE1,LIU Hui1,3,Zengquan YANG1
(1.Karmanos Cancer Institute,Department of Oncology,Wayne State University,Detroit,MI 48201,USA;2.College of Llife Science,Jiangsu Normal University,Jiangsu Xuzhou 221116,China;3.Key Laboratory of Pathology Biology,Ministry of Education,School of Basic Medical Sciences,Jilin University,Jilin Changchun 130021,China)
Histone lysine methylation,a covalent histone modification,plays a central role in epigenetic regulation networks of genome function.The histone lysine demethylase(KDM)4 subfamily catalyzes the removal of methyl marks from histone lysine residues to epigenetically regulate chromatin structure,cell cycle,genome integrity and gene expression.Dysregulation of the KDM4 demethylases is associated with abnormities of cell proliferation and differentiation,development and metabolism,aswell as tumorigenesis.Furthermore,KDM4 demethylases are promising druggable targets due to their enzymatic activity.In this review,we summarize recent findings regarding the structures and regulatory mechanisms of the KDM4 proteins,aswell as our current understanding of their alterations and roles in human disease.We also review the reported KDM4 inhibitors and discuss their potential as therapeutic agents.
Lysine-specific demethylase;Lysine-specific demethylase 4(KDM4);Tumour
Q55;R73
A
2095-3097(2014)01-0013-06
10.3969/j.issn.2095-3097.2014.01.004
2013-06-18 本文编辑:徐海琴)
Karmanos Cancer Institute-SRIG;江苏师范大学校基金(09XLA08、11XLR25)
MI 48201 Detroit USA,Karmanos Cancer Institute,Department of Oncology,Wayne State University(叶 覃,Andreana HOLOWATYJ,Roselyne M.LABBE,Zengquan YANG);221116江苏徐州,江苏师范大学生命科学学院(叶 覃);130021吉林长春,吉林大学白求恩医学院病理生物学教育部重点实验室(刘 辉)
