某院鲍曼不动杆菌流行克隆株的分子特征
2014-02-14刘超梅胡晓冬邢红英郭素芳李雪梅杨晓波
刘超梅,胡晓冬,邢红英,郭素芳,李雪梅,杨晓波,肖 丽
近年来,鲍曼不动杆菌(acinetobacter baumannii)引起的医院感染日益突出[1-4],其主要流行克隆株的监测及定义已成为分子流行病学学者关注的热点[5-6]。有研究依据AFLP分型方法将鲍曼不动杆菌的流行菌株分别定义为欧洲克隆谱系Ⅰ、Ⅱ和
Ⅲ[7];而英国学者 Turton等[8]依据脉冲场凝胶电泳(PFGE)分型方法发现了SE克隆系、OXA-23克隆系1和2。Turton等[9]基于序列分型的多重聚合酶链反应(PCR)方法,实现了准确而快速地对鲍曼不动杆菌的同源性进行分析,该研究将上述不同的克隆谱系分为3组(Group 1、Group 2和 Group 3),Group 1包括SE克隆系、欧洲克隆谱系Ⅱ及OXA-23克隆系1,Group 2包括欧洲克隆谱系Ⅰ、OXA-23克隆系2等,而Group 3主要包括欧洲克隆谱系Ⅲ。本文对不重复鲍曼不动杆菌进行同源性分析,了解其主要流行克隆株的分子特征,为控制医院感染提供理论依据。
1 材料与方法
1.1 菌株来源 将解放军253医院2010年6月—2012年12月临床分离的74株不重复鲍曼不动杆菌作为实验菌株,标本来源:痰32例,尿16例,创面或伤口分泌物13例,血液10例,脑脊液3例。
1.2 引物合成及PCR扩增 多种耐药基因、Group 1和Group 2多重PCR引物设计及PCR扩增方法参照文献[10-12],引物由上海生工生物工程技术服务有限公司合成。PCR产物在含0.5μg/ml溴化乙啶(EB)的1.5%琼脂糖凝胶上电泳,用凝胶成像系统记录电泳结果,每次PCR检测均设阴性对照。同时于阳性扩增产物中随机抽取3株,送上海英骏生物技术有限公司进行测序,测序结果通过GenBank(http://blast.ncbi.nlm.nih.gov/Blast.cgi)比对分析。
1.3 PFGE实验方法 按国家疾病控制中心传染病所PulseNet China网络实验室提供的鲍曼不动杆菌PFGE标准程序进行操作,具体实验方法及结果分析参照文献[13]。
1.4 克隆菌株聚类分析 基于序列分型的多重PCR方法鉴定鲍曼不动杆菌的主要流行克隆谱系,结果判定参照文献[12]。PFGE实验得到74株鲍曼不动杆菌的DNA指纹图谱,用BioNumerics软件对各菌株间进行聚类分析,将相似性系数87%作为不同型别的区分标准,相似度>87%为同一亚型,代表同一克隆株;<87%为不同的基因型,代表不同的克隆株。
1.5 多重耐药相关基因谱分析 应用PCR及测序方法[10-12]检测10种多重耐药相关基因(blaOXA-51-like、blaOXA-23-like、blaOXA-24-like、blaOXA-58-like、blaTEM、blaampC、armA、ISA ba1、intI 1 和 intI 2)的携带情况。
2 结果
2.1 Group PCR序列分型结果 74株鲍曼不动杆菌分为 6种 Group序列型(SG),其中 57株(77.0%)为 Group SG1,4 株(5.4%)为 Group SG4,8株(10.8%)为 Group SG5,2株(2.7%)为 Group SG9,1 株(1.4%)为 Group SG11,2 株(2.7%)为Group SG13。
2.2 克隆菌株聚类分析结果 74株鲍曼不动杆菌的PFGE带型表现出多样性,可大致分为13种图谱,其中 A型35株、B型17株、C型7株、D型5株,其余型别10株。74株鲍曼不动杆菌克隆菌株聚类分析树状图见图1。
2.3 鲍曼不动杆菌多重耐药相关基因谱的分布情况 blaOXA-51-like在所有菌株中均为阳性,除blaOXA-24-like、blaOXA-58-like和 intI 2基因为阴性外,其余多重耐药相关基因均有阳性扩增产物,通过阳性菌株数所占实验菌株数的比例获得耐药相关基因的阳性率,耐药相关基因 blaOXA-23-like、blaTEM、blaampC、armA、ISA ba1和intI 1的阳性率分别为54.1%(40/74)、86.5%(64/74)、66.2%(49/74)、89.2%(66/74)、90.5%(67/74)和85.1%(63/74),并且测序结果与GenBank库中的已知序列具有高度的一致性。多种耐药基因在不同PFGE型中的分布情况见表1。结果显示,多种耐药基因在A型和B型菌种中的分布比较集中,同一耐药基因在不同PFGE型中呈现出交叉分布现象。
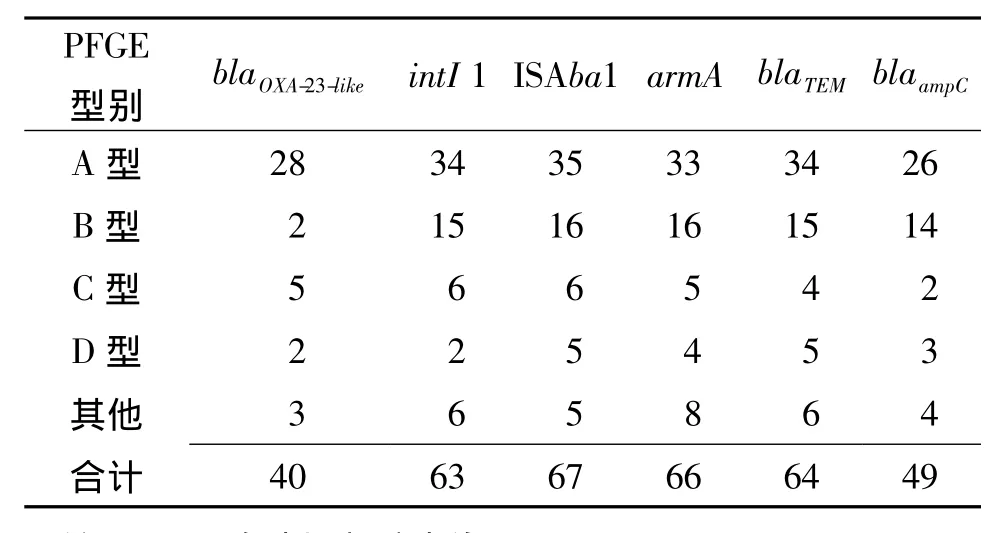
表1 74株鲍曼不动杆菌多重耐药相关基因在不同PFGE型别中的分布情况(株)

图1 74株鲍曼不动杆菌克隆菌株聚类分析树状图
3 讨论
在鲍曼不动杆菌的流行病学调查和预防控制研究方面,尤其是在医院感染暴发期间,为了证实是否存在其克隆株的传播流行,实现感染源的溯源,迫切需要选用一种行之有效的基因分型方法。基于SG的多重PCR方法操作简便,结果容易判读,无需进一步测序或应用其他分型技术,非常适合于临床实验室快速准确地鉴定鲍曼不动杆菌流行菌株的SG或克隆系,从而有助于实时掌控鲍曼不动杆菌院内感染发生的流行趋势。
本研究结果显示,本组鲍曼不动杆菌克隆谱系以Group GS1为主,占77.0%。根据Group GS1对应于欧洲克隆谱系Ⅱ的结果判断依据,解放军253医院临床分离的74株鲍曼不动杆菌的流行克隆谱系主要为欧洲克隆谱系Ⅱ,该谱系是鲍曼不动杆菌最大的流行克隆系,在全世界不同国家均有分布。
目前,多种基因分型技术在鲍曼不动杆菌菌株分型中得到了广泛应用[14-18],其中PFGE被誉为细菌分子流行病学分型技术的“金标准”[19]。本研究对74株鲍曼不动杆菌进行了PFGE分型,在87%的相似性水平上,共得到13种PFGE图谱,显示出高度多样性,其中A型和B型为主要型别,分别占总菌株数的 47.3%(35/74)和 23.0%(17/74),说明在解放军253医院住院患者中存在鲍曼不动杆菌主要克隆株的流行。因此,需要临床医生能快速、实时地监测、追踪鲍曼不动杆菌主要流行克隆株的变化趋势,并采取合理措施预防和控制其传播,尤其要防控医院感染的流行或暴发。
鲍曼不动杆菌的耐药呈逐年上升趋势[20-23],OXA类碳青霉烯酶、插入序列、整合子、ampC酶、超广谱β内酰胺酶(ESBLs)等在鲍曼不动杆菌的多重耐药机制方面发挥着重要的作用[24-25]。本研究结果显示,40株(54.1%)鲍曼不动杆菌携带有blaOXA-23-like基因,进一步证实耐碳青霉烯类的鲍曼不动杆菌以产 OXA-23型碳青霉烯酶为主[10];在66.2%的菌株中检出blaampC基因,86.5%的菌株中检出blaTEM基因,可见对于本组分离的鲍曼不动杆菌,blaTEM的检出率高于国内其他地区水平[12]。通过检测整合酶基因可以筛查和鉴定鲍曼不动杆菌的多重耐药性[26]。在本研究74株鲍曼不动杆菌中仅检测出intI 1,未发现intI 2,说明本组鲍曼不动杆菌主要携带Ⅰ类整合子,检出率高达85.1%。在鲍曼不动杆菌中,ISA ba1通过提供启动子序列来增强β内酰胺酶的表达[27],ADC型ampCβ内酰胺酶和OXA类碳青霉烯酶基因的上游经常发现存在有ISA ba1,本研究中插入序列ISA ba1的检出率高达90.5%。16SrRNA甲基化酶基因armA绝大多数位于整合子、质粒等可移性遗传元件上,易于在细菌间传播,与阿米卡星的耐药性密切相关,本研究74株实验菌株中89.2%的菌株携带此基因。
本研究对鲍曼不动杆菌多重耐药相关基因在不同PFGE型别中的分布特征进行了分析,发现在不同的PFGE型别中同一耐药基因呈现出交叉分布现象,进一步说明了在具有不同遗传背景的鲍曼不动杆菌中发生着耐药基因的水平转移。
[1] 侯万乐,田萍.98株鲍曼不动杆菌医院感染分布及耐药性分析[J].中国当代医药,2010,17(36):83-84.
[2] 喻茂文,肖严,周吕蒙,等.临床鲍曼不动杆菌分布和耐药性分析[J].中国现代医生,2012,50(28):62-64.
[3] 燕成岭,曹银芳,李凤娥.鲍曼不动杆菌感染分布及耐药性分析[J].中国医药,2011,6(7):863-864.
[4] 安宝清,王金侠,唱丽敏.320株鲍曼不动杆菌的临床分布及耐药性分析[J].临床荟萃,2011,26(17):1534-1536.
[5] Higgins P G,Janssen K,Fresen M M,et al.Molecular epidemiology of Acinetobacter baumannii bloodstream isolates obtained in the United States from 1995 to 2004 using rep-PCR and multilocus sequence typing[J].JClin Microbiol,2012,50(11):3493-3500.
[6] Hamouda A,Evans B A,Towner K J,et al.Characterization of epidemiologically unrelated Acinetobacter baumannii isolates from four continents by use of multilocus sequence typing,pulsed-field gel electrophoresis,and sequence-based typing of bla(OXA-51-like)genes[J].J Clin Microbiol,2010,48(7):2476-2483.
[7] van Dessel H,Dijkshoorn L,van der Reijden T,et al.Identification of a new geographically widespread multiresistant Acinetobacter baumanniiclone from European hospitals[J].Res Microbiol,2004,155(2):105-112.
[8] Turton J F,Kaufmann M E,Gill M J,et al.Comparison of Acinetobacter baumannii isolates from the United Kingdom and the United States that were associated with repatriated casualties of the Iraq conflict[J].JClin Microbiol,2006,44(7):2630-2634.
[9] Turton J F,Gabriel SN,Valderrey C,et al.Use of sequence-based typing and multiplex PCR to identify clonal lineages of outbreakstrains of Acinetobacter baumannii[J].Clin Microbiol Infect,2007,13(8):807-815.
[10]闫中强,沈定霞,罗燕萍,等.多重PCR方法检测多耐药鲍曼不动杆菌基因型[J].临床检验杂志,2008,26(6):422-424.
[11]董喆,徐雅萍,龚美亮.高龄住院患者鲍曼不动杆菌多重耐药相关基因的监测[J].解放军医学杂志,2012,37(3):234-237.
[12]董喆,丁志平,徐雅萍.部队医院多药耐药鲍氏不动杆菌主要流行克隆谱系的分析[J].中华医院感染学杂志,2011,21(24):5118-5121.
[13]崔晶花,杜小莉,崔志刚,等.PulseNet China网络实验室能力考核结果评价与分析[J].疾病监测,2011,26(10):766-769.
[14] Saeed S,Fakih M G,Riederer K,et al.Interinstitutional and intrainstitutional transmission of a strain of Acinetobacter baumanniidetected by molecular analysis:comparison of pulsed-field gel electrophoresis and repetitive sequence-based polymerase chain reaction[J].Infect Control Hosp Epidemiol,2006,27(9):981-983.
[15] Bartual SG,Seifert H,Hippler C,et al.Development of a multilocus sequence typing scheme for characterization of clinicalisolates of Acinetobacter baumannii[J].J Clin Microbiol,2005,43(9):4382-4390.
[16] Diancourt L,Passet V,Nemec A,et al.The population structure of Acinetobacter baumannii:expanding multiresistant clones from an ancestral susceptible genetic pool[J].PLoSOne,2010,5(4):e10034.
[17] Carretto E,Barbarini D,Farina C,et al.Use of the DiversiLab semiautomated repetitive-sequence-based polymerase chain reactionfor epidemiologic analysis on Acinetobacter baumannii isolates in different Italian hospitals[J].Diagn Microbiol Infect Dis,2008,60(1):1-7.
[18] Turton J F,Matos J,Kaufmann M E,et al.Variable number tandem repeat loci providing discrimination within widespread genotypesof Acinetobacter baumannii[J].Eur JClin Microbiol Infect Dis,2009,28(5):499-507.
[19]Peleg A Y,Seifert H,Paterson D L.Acinetobacter baumannii:emergence of a successful pathogen[J].Clin Microbiol Rev,2008,21(3):538-582.
[20]习慧明,徐英春,朱德妹,等.2010年中国CHINET鲍曼不动杆菌耐药性监测[J].中国感染与化疗杂志,2012,12(2):98-104.
[21]胡智玲.鲍曼不动杆菌感染的耐药性分析[J].中国煤炭工业医学杂志,2011,14(7):995-996.
[22]叶春幸,马真,卓宋明.鲍曼不动杆菌的院内感染及耐药性分析[J].中国医药导报,2013,10(14):116-117,120.
[23]董爱英,刘丽娜,郑永强,等.鲍曼不动杆菌耐药情况分析[J].中国煤炭工业医学杂志,2005,8(12):1277-1278.
[24]Karthikeyan K,Thirunarayan M A,Krishnan P.Coexistence of blaOXA-23 with blaNDM-1 and armA in clinical isolates of Acinetobacter baumannii from India[J].J Antimicrob Chemother,2010,65(10):2253-2254.
[25]Wang X,Zong Z,LüX.Tn2008 is a major vehicle carrying bla(OXA-23)in Acinetobacter baumannii from China[J].Diagn Microbiol Infect Dis,2011,69(2):218-222.
[26] Gaur A,Prakash P,Anupurba S,et al.Possible role of integrase gene polymerase chain reaction as an epidemiological marker:study of multidrug-resistant Acinetobacter baumannii isolated from nosocomial infections[J].Int J Antimicrob Agents,2007,29(4):446-450.
[27] Turton JF,Ward M E,Woodford N,et al.The role of ISAba1 in expression of OXA carbapenemase genes in Acinetobacter baumannii[J].FEMS Microbiol Lett,2006,258(1):72-77.
