伤口分泌物分离阴沟肠杆菌的耐药性分析
2014-02-08张丽琴肖九长吴天恩
张丽琴,肖九长,吴天恩
(赣州市人民医院/南昌大学附属赣州医院,江西赣州341000)
伤口分泌物分离阴沟肠杆菌的耐药性分析
张丽琴,肖九长,吴天恩
(赣州市人民医院/南昌大学附属赣州医院,江西赣州341000)
目的了解伤口分泌物分离的阴沟肠杆菌的耐药特点,为临床治疗提供参考.方法使用WHONET5.6统计软件回顾性分析2011年1月至2012年11月我院送检的伤口分泌物中分离的阴沟肠杆菌的耐药状况.结果106株阴沟肠杆菌中,阿米卡星的耐药率最低(8.5%),其次是亚胺培南(13.2%)和厄他培南(13.2%),头孢类抗生素的耐药率均超过30%.结论伤口分泌物中分离的阴沟肠杆菌多重耐药日趋严重,临床应根据其耐药特点选用合适的抗生素治疗,以防止多重耐药株和泛耐药株的传播.
伤口;阴沟肠杆菌;耐药性
阴沟肠杆菌是一种常见的条件致病菌,可导致呼吸道、泌尿道、伤口的严重感染,且多为医院内感染.为指导伤口感染阴沟肠杆菌的治疗,本文对我院2011年1月至2012年11月伤口分泌物中分离出的106株阴沟肠杆菌的耐药性进行分析,报道如下.
1 材料与方法
1.1 标本来源2011年1月至2012年11月我院骨科和创伤科住院患者送检的伤口分泌物,剔除同一患者多次分离的重复菌株.
1.2 仪器和试剂法国梅里埃公司生产的VITEK-2全自动微生物分析系统及配套使用的细菌鉴定GN卡和GN13药敏卡.
1.3 方法按《全国临床检验操作规程》第3版对送检标本进行培养,获得单个菌落后,上机鉴定和药敏.
1.4 质控菌株大肠埃希菌(ATCC25922)、嗜麦芽窄食单胞菌(ATCC17666).
1.5 统计学方法采用WHONET5.6软件进行数据统计分析.
2 结果
106株阴沟肠杆菌,多重耐药株17株,占16%,泛耐药株3株,占2.8%,详见表1.
3 讨论
近年来,随着广谱抗菌药物的广泛使用,阴沟肠杆菌的检出率及耐药性呈逐年升高趋势[1],特别是在伤口分泌物分离的肠杆菌科细菌中,阴沟肠杆菌的分离率仅次于大肠埃希菌[2].尽管,阴沟肠杆菌导致伤口化脓的机会是革兰阳性菌的1/4~1/5,但它们导致患者的死亡率却是后者的2倍[3].因而阴沟肠杆菌引起的伤口感染应引起临床重视.
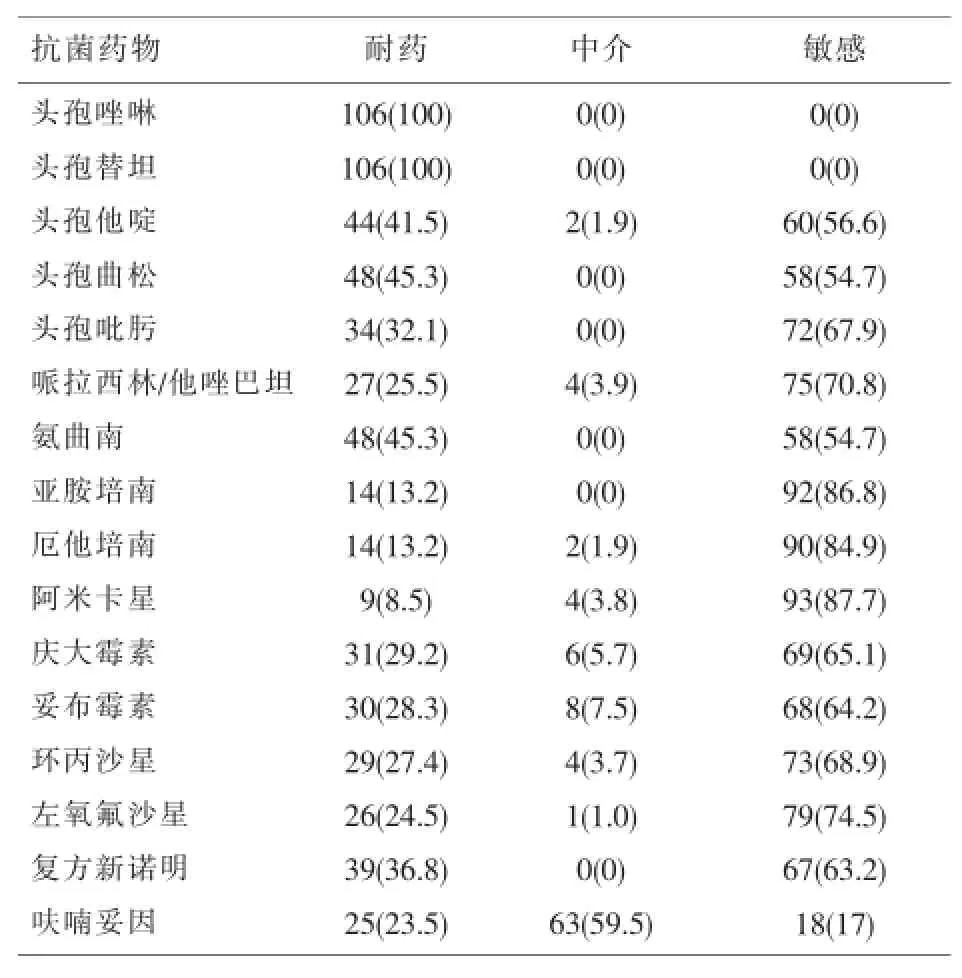
表1 106株阴沟肠杆菌对16种抗菌药物的耐药率[n(%)]
本结果显示,阴沟肠杆菌对三代头孢菌素和氨曲南的耐药率较高,都超过了40%.对四代头孢菌素的耐药率为32.1%,亚胺培南和厄他培南的耐药率为13.2%,仅阿米卡星的耐药率低于10%,它可作为治疗阴沟肠杆菌感染较好的选择药物,但其肾毒性和耳毒性等不良反应较大,临床使用时应慎重.亚胺培南等碳青霉烯类抗生素仍是治疗阴沟肠杆菌感染的最佳药物,因其抗菌活性强,对大多数β-内酰胺酶都有较高的稳定性,与青霉素结合蛋白亲和力强,能有效渗透细菌外膜进入周质间隙,达到快速杀菌的效果.
阴沟肠杆菌的耐药机制复杂,可表现为染色体介导的AmpC酶、质粒介导的ESBLs、去阻遏持续高产AmpC酶、膜蛋白丢失等机制共同作用,故呈现出耐药谱广等特点,给临床的治疗带来了困难.产AmpC酶是阴沟肠杆菌耐药的重要机制之一,这可能与阴沟肠杆菌染色体上AmpD基因有较高的突变率导致AmpC基因去阻遏持续高水平表达有关[4].三代头孢菌素对高产AmpC酶的选择能力是最强的,在用其治疗阴沟肠杆菌感染时易造成继发耐药.因此,有研究者建议在确认为阴沟肠杆菌感染后,应限制头孢菌素类抗菌剂的使用,以避免诱导高产AmpC酶的产生[5].
本次分析的106株阴沟肠杆菌中,多重耐药株所占的比例达到了16%,泛耐药株也占了2.8%,提示阴沟肠杆菌多重耐药现象日趋严重,临床应给予高度重视,采取相应措施,减少医源性感染.
[1]梁志科,刘朝晖,叶惠芬,等.阴沟肠杆菌临床感染分析及药敏监测[J].中华医院感染学杂志,2008,18(2):264-266.
[2]张丽琴,李世云,肖九长.伤口分泌物的细菌分离鉴定及耐药性分析[J].实验与检验医学,2010,28(4):411-412.
[3]Valles J,Leon C,Alvarez-lerma F.Nosocomial bacteremia in critically ill patients:a multicenter study evaluating epidemiology and prognosis[J].Clin Infect Dis,1997,24(3):387-395.
[4]隋秀丽,苏维奇.阴沟肠杆菌产AmpC酶和ESBLs的检测及其耐药机制研究[J].中国实验诊断学,2010,14(8):1255-1257.
[5]赵德军,田维涛,张碧霞,等.临床阴沟肠杆菌感染分布及其体外药敏分析[J].西南国防医药,2009,19(6):618-619.
R446.5,Q939.92,R446.19
A
1674-1129(2014)03-0289-02
10.3969/j.issn.1674-1129.2014.03.019
2013-12-17;
2013-12-23)
