猪Centaurin-α1的克隆分析与原核表达载体构建
2013-11-23马金友刘俊伟徐之勇朱岩昆陈金山
马金友,余 燕,刘俊伟,徐之勇,朱岩昆,陈金山
(河南科技学院动物科学学院,河南 新乡453003)
猪病毒病的暴发是长期制约生猪养殖和猪肉产品加工迅速发展的重要因素之一,尤其是2006年以来猪繁殖与呼吸综合征病毒(PRRSV)变异株引起的高热病的暴发使养猪业遭受了重大经济损失,因此,通过“预防为主”,提高猪只的自身免疫能力以增强其抗病力,是解决猪病及抗生素等药物滥用问题的一条非常有效的途径。目前,对于PRRSV免疫预防研究工作已经取得了一些进展[1-2],但对病毒入侵宿主细胞后,一些相关免疫因子对病毒感染过程中的囊泡运输系统是如何调控的还知之甚少,尤其在病毒的复制或参与病毒防御等方面。
Centaurin-α1是Centaurin蛋白家族中第一个被发现的成员,该基因于1996年首先在大鼠脑中被克隆得到,其可以特异性地通过PH结构域与3,4,5-三磷酸磷脂酰肌醇(phosphatidylinositol 3,4,5-trisphosphate,PtdIns(3,4,5)P(3))相结合,使 Centaurin-α1蛋白向细胞膜上迁移,通过 Ras-Raf-MEK-ERK1/2途径调控细胞功能,从而参与到信号转导通路中[3-4],对Centaurin-α1的研究主要是其作为ARF6的GAP(GTPase-activating protein)所行使的调控功能[5]。作为ARF的GAP,Centaurin-α1通过对ARF的调控而间接地参与到了病毒和抗病毒感染过程中[6],同时,作为细胞信号转导PI3-K途径下游的重要成员[4,7],我们仍可以预见,Centaurin-α1可能与猪只抗病毒免疫密切相关。
由于Centaurin-α1在PRRSV感染中的免疫防御作用和调节机制缺乏了解,本文拟通过猪只Centaurin-α1基因的克隆与分析,构建该基因的原核表达载体以获得抗血清,为进一步研究猪Centaurin-α1在PRRSV感染过程中的调控作用提供理论基础。
1 材料与方法
1.1 试剂与设备
1.1.1 常用试剂 限制性内切酶EcoRⅠ与HindⅢ、反转录酶 M-MLV、Taq DNA 聚合酶,pMD18-T载体、质粒提取试剂盒、DNA回收试剂盒、PrimeSTARTMHS DNA Polymerase,均购自宝生物工程(大连)有限公司;琼脂糖、蛋白胨和酵母浸出膏等,购自上海生工生物工程技术服务有限公司;枪头和离心管(Axygen),购自北京鼎国昌盛生物技术有限责任公司;其他常用试剂均为国产分析纯试剂;pET-His载体和大肠杆菌(Escherichia coli Top10)由本实验室保藏。
1.1.2 主要设备 PCR仪,凝胶成像系统,水平电泳槽,电泳仪,超净工作台,台式冷冻离心机,隔水式恒温培养箱,恒温摇床,电子精密天平,高压灭菌锅,制冰机等。
1.2 材料 从动物科学学院兽医院取疑似PRRSV感染的杜洛克猪肺、脑、淋巴结、心脏等少量组织,液氮冻存带回实验室处理。
1.3 引物设计 根据NCBI(National Center for Biotechnology Information)猪 Centaurin-α1(M _214226)序列设计扩增Centaurin-α1全长ORF引物(CentF:5′-ATGGCCAA-GGAGCGGCGG-3′ 和CentR:5′-CGCTAGGGTTTATGC-TTGAAGTG-3′,上海生工生物工程技术服务有限公司合成)和原核表达引物(Sus-CenF1:5′-GCGAATTCATCTGCCTGAGCTGCTTG-3′ 和 SusCenR1: 5′-CGAAGCTTCGCTAGGGTTTATG-CTTGAAG-3′,上海生工生物工程技术服务有限公司法合成,下划线为酶切位点)。
1.4 方法
1.4.1 猪Centaurin-α1基因扩增
1.4.1.1 组织总RNA提取 取适当组织(200mg以下)于研钵(180℃烘烤3h)中加入液氮充分研磨,然后加入1 000μL Trizol(Invitrogen)混匀,分装为2管,再分别加入Trizol补充至每管1 000μL,混匀(1管冻存,1管用于试验),室温放置10min;12 000r/min(4℃)离心10min;上清转移至一新管,加入0.2mL氯仿/每1 000μL Trizol,剧烈摇动15s左右,室温放置8min左右;12 000r/min(4℃)离心15min;上层水相转移至一新管中,加入0.5 mL异丙醇混匀,室温放置8min;12 000r/min(4℃)离心8min;弃上清,1mL 75%乙醇洗1min,7 500r/min(4℃)离心5min,重复洗1次;弃上清,空气干燥3min,加入60μL DEPC水溶解沉淀。
1.4.1.2 cDNA合成 首先配制以下成分:1μL 6 mer随机引物,4μg 总 RNA,1μL 10mmol/L dNTP混合物,DEPC水补充到14μL;PCR仪中65℃加热5min,然后冰浴至少1min;轻微离心一下,然后加入以下成分:4μL 5×first-strand Buffer,1μL Rnase inhibitor,1μL反转录酶 M-MLV;PCR仪中25℃温浴5min,42℃温浴60min,70℃加热15min以失活成分,4℃后保存冰箱中备用。
1.4.1.3 Centaurin-α1基因扩增 PCR 反应管中分别加入以下成分:5×Prime-STARTMBuffer 5 μL,2.5mmol/L dNTP混合物2μL,上下游引物各1.0μL,10倍稀释的反转录产物2.0μL,PrimeSTARTMHS DNA Polymerase 0.25μL,补充灭菌三蒸水至25μL。PCR反应程序如下:95℃,3min;98℃10s,55℃15s,72℃70s,30个循环;72℃再延伸5 min,4℃终止。
1.4.2 基因扩增产物的回收、连接和测序 检测扩增的目的片段,根据DNA回收试剂盒说明回收目的片段;0.5mL离心管中依次加入:载体(pMD18-T)0.5L,目的片段6.0L,10×Liga-tion Buffer 1.0L,T4Ligase 0.6L,灭菌三蒸水补充至10.0 L,16℃连接6h;连接产物转化Top 10细菌和转化细菌的检测及测序,正确测序产物质粒的提取、保存。
1.4.3 序列分析 分别运用 Clustal X[8]和 Blast程序 (http://www.ncbi.nlm.nih.gov/blast/)及Signal P 4.0 (http://www.cbs.dtu.dk/services/SignalP/)分析核酸序列和预测蛋白的保守结构域及信号肽。通过 MEGA 4.0[9]程序的邻接法(Neighbor-Joining,NJ,bootstrap1000)构建了相关物种Centaurin-α1的系统发生树。
1.4.4 猪 Centaurin-α1融合蛋白原核表达质粒构建
1.4.4.1 Centaurin-α1基因表达序列的扩增 除扩增引物为SusCenF1和SusCenR1及模板为提取含Centaurin-α1序列质粒外,其他成分和程序参照1.4.1.3的成分和步骤。
1.4.4.2 扩增的 Centaurin-α1序列和连接载体酶切及回收 1.5mL Eppendorf管中分别依次加入:内切酶缓冲液(10xK)5.0L;DNA片段和pET-His分别为43.0L;EcoRⅠ1.0L,HindⅢ1.0L,37℃酶切2.5h。酶切后的片段回收按照DNA回收试剂盒说明进行。
1.4.4.3 载体和目的片段连接、转化及检测 0.5 mL离心管中依次加入:酶切载体(pET-His)1.5 L,待连接的酶切 Centaurin-α1片段6.5L,10×Ligation Buffer 1.0L,T4Ligase 1.0μL,总体积10.0L,16℃连接6h。连接产物转化Top 10细菌,转化细菌的菌落检测和质粒双酶切检测验证,符合要求的菌落测序,正确测序产物质粒的提取、保存。
2 结果
2.1 猪Centaurin-α1基因ORF的克隆与分析
2.1.1 猪Centaurin-α1基因ORF的克隆 通过猪脑和肺脏组织总RNA的提取、反转录和基因的扩增,得到了和目的基因一致的条带(图1),测序后确认为猪Centaurin-α1基因,大小为1 100bp左右。
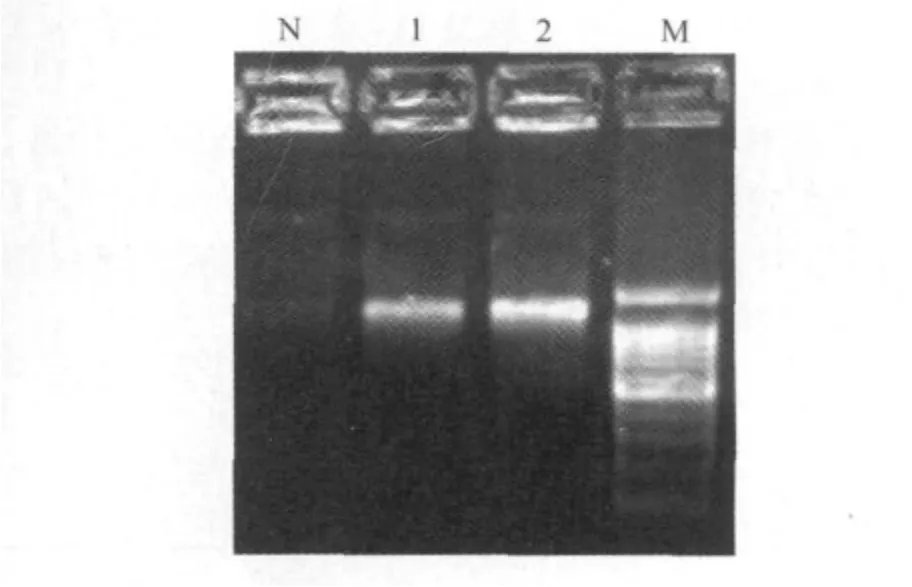
图1 克隆的猪Centaurin-α1电泳图

图2 猪Centaurin-α1基因全长序列和蛋白序列中的保守结构域
2.1.2 猪Centaurin-α1基因ORF及结构特征 猪Centaurin-α1基因包含一个长度为1 122bp开放阅读框,预测编码374个氨基酸,理论分子质量约为43.50kDa,pI为8.95(图2,A)。
通过NCBI Blast寻找其序列中的保守结构域,我们发现,猪Centaurin-α1蛋白包含一个Arf GAP和两个PH结构域(图2,B)。信号肽预测该蛋白前22个氨基酸为信号肽序列。
2.1.3 猪Centaurin-α1分子系统分析 根据 Centaurin-α1氨基酸序列构建的 NJ树(Neighbor-Joining tree)显示:该蛋白首先与牛 Centaurin-α1聚为一类,说明其与牛Centaurin-α1亲缘关系较近,这从它们同为偶蹄目动物能够得到进一步的证实,与哺乳动物中人和啮齿目动物也有较高的同源性,而和鱼纲中的斑马鱼和两栖类的非洲爪蛙亲缘关系稍远(图3)。

图3 构建的猪Centaurin-α1NJ树
2.2 猪Centaurin-α1融合蛋白原核表达载体构建由于Centaurin-α1是细胞中调控囊泡运输蛋白的重要因子,对病毒的入侵起着重要的调控作用,为了获得抗血清进一步研究它的功能,对其原核表达载体进行构建。通过设计的原核表达引物经PCR从质粒pMD18-T-Centaurin-α1中克隆表达序列,双酶切后连接到pET-His载体上,通过菌落PCR获得了和目的片段大小一致的条带,挑选的菌落培养后提取的质粒双酶切图谱(图4)进一步证实构建融合蛋白表达载体的正确性。根据克隆的Centaurin-α1基因,我们成功构建了猪Centaurin-α1原核表达载体用于融合蛋白的表达。

图4 构建的猪Centaurin-α1融合蛋白表达载体双酶切
3 讨论
PRRSV是影响世界猪产业发展的重要流行性传染病之一,具有导致怀孕母猪发生流产、早产和死胎等严重病症特点,尤其2006年后该病的暴发使养猪业遭受了重大损失。在研究中发现PRRSV感染早期猪Centaurin-α1基因表达明显上调,说明猪Centaurin-α1因子可能参与了早期病毒感染的防御作用。目前,国内外关于Centaurin蛋白家族的报道还非常少,作为这个家族中最早被发现的成员,Centaurin-α1在人们的研究中大多数都还仅限于它与ARF6的关系。然而,从Centaurin-α1蛋白所具有的保守结构域上,我们不难看到该蛋白可能具有的功能和作用是不容忽视的。因为Centaurin-α1不仅可以作为ARF6的GAP,从而间接参与到细胞吞噬、细胞运动和物质运输等活动中,它拥有的两个PH结构域及可与第二信使3,4,5-三磷酸磷脂酰肌醇特异性结合的能力也暗示了它可能在细胞信号转导中具有重要地位。而且,根据文献报道,Centaurin-α1可能是重要细胞信号转导途径PI3-K途径下游的成员[7],尽管目前在这方面还没有具体的研究,但是我们仍是可以预见Centaurin-α1可能与猪的相关病毒入侵有着密切的相关性。
[1]Luo R,Fang L R,Jin H,et al.Antiviral activity of type I and type III interferons against porcine reproductive and respiratory syndrome virus(PRRSV)[J].Antiviral Res,2011,91(2):99-101.
[2]Cruz J L G,Zúñiga S,Bécares M,et al.Vectored vaccines to protect against PRRSV[J].Virus Res,2010,154(1-2):150-160.
[3]Hammonds-Odie L P,Jackson T R,Profit A A,et al.Identification and cloning of centaurin-αa novel phosphatidylinositol-3,4,5-trisphosphate-binding protein from rat brain[J].J Biol Chem,1996,271(31):18859-18868.
[4]Hayashi H,Matsuzaki O,Muramatsu S,et al.Centaurin-α1 Is a Phosphatidylinositol 3-Kinase-dependent activator of ERK1/2mitogen-activated protein kinases[J].J Biol Chem,2006,281(3):1332-1337.
[5]Lawrence J,Mundell S J,Yun H R,et al.Centaurin-α1,an ADP-ribosylation factor 6GTPase activating protein,inhibits β2-adrenoceptor internalization[J].Mol Pharmacol,2005,67(6):1822-1828.
[6]Wang H F,Ma J Y,Ruan L W,et al.Cloning of a centaurina1like gene MjCent involved in WSSV infection from shrimp Marsupeneaus japonicus [J].Fish Shellfish Immunol,2009,26(2):279-284.
[7]Ehrhardt C,Marjuki H,Wolff T,et al.Bivalent role of the phosphatidylinositol-3-kinase(PI3K)during influenza virus infection and host cell defence[J].Cell Microbiol,2006,8(8):1336-1348.
[8]Jeanmougin F,Thompson J D,Gouy M,et al.Multiple sequence alignment with Clustal X [J].Trends Biochem Sci,1998,23(10):403-405.
[9]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
