易错PCR提高华根霉脂肪酶的热稳定性
2013-10-31王睿喻晓蔚徐岩
王睿,喻晓蔚,徐岩
1 江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122
2 江南大学 药学院,江苏 无锡 214122
脂肪酶 (EC 3.1.1.3)不仅能催化油脂水解,也能在非水相中催化酯合成、转酯化、酸解等反应,被广泛地应用于化学、制药、生物能源工业[1-2]及食品工业[3-4]中。根霉脂肪酶在脂肪酶体系中有重要地位。根霉属主要包括微孢根霉Rhizopus microsporus、匍枝根霉 Rhizopus stolonifer和米根霉Rhizopus oryzae 3类,米根霉中又包括德式根霉Rhizopus delemar、爪哇根霉Rhizopus javanicus、雪白根霉Rhizopus niveus和少根根霉Rhizopus arrhizus。不同来源的根霉脂肪酶序列有所差异,但是它们都具有以下共同结构特征:具有 а/β水解酶折叠结构特征;拥有Gly-X-Ser-X-Gly模体;催化三联体由Ser-His-Asp/Glu被一个具有两亲性的肽环“盖子”所覆盖。当脂肪酶在油一水界面时,肽环“盖子”构型发生改变,催化部位被暴露,底物与催化中心接触而进行反应;稳定性佳、转化效率高等。目前已经有超过30种商品化的根霉脂肪酶应用于芳香酯、生物柴油、手性化合物的生产中[5-6]。
本研究室在前期研究中从酿造浓香型大曲酒的酒曲中筛选到一株华根霉[5],从中克隆得到脂肪酶基因 proRCL[7],并成功实现了脂肪酶r27RCL在毕赤酵母中的高效表达以及在发酵罐中的高密度表达[7-8]。但是,由于华根霉脂肪酶是中温酶,热稳定性差[9],高温下的半衰期非常短[10-12]。这不仅限制了其应用范围,还增加了工业应用成本,为实际应用带来困难。因此,提高r27RCL的热稳定性至关重要。
定向进化是提高酶的热稳定性最快捷而有效的方法。近 10年来,定向进化技术已在脂肪酶热稳定性改造领域取得巨大成功。如:Kohno等利用易错PCR,将来自雪白根霉R. niveus的脂肪酶RNL的Tm值提高了10 ℃[13]。又如谭天伟等经过易错PCR、DNA shuffling和筛选,使来自少根根霉R. arrhizus的脂肪酶RAL的最适温度提高10 ℃,50 ℃下的半衰期提高为原来的12倍[14]。本研究利用易错PCR的方法向该酶中随机引入突变,利用 PCR依赖型方法构建华根霉脂肪酶基因突变文库[15],再结合fast-blue RR顶层琼脂筛选方法,获得了热稳定性提高的脂肪酶突变株。
1 材料与方法
1.1 材料
1.1.1 酶与试剂
限制性内切酶、T4 DNA连接酶、Taq DNA聚合酶、PCR试剂 (TaKaRa宝生物公司),引物(上海生工生物工程技术服务有限公司)合成,DL2 000 DNA Ladder marker、E10 000 DNA Ladder marker、琼脂糖凝胶DNA回收试剂盒、PCR产物纯化试剂盒 (天根生化科技 (北京)有限 公 司),Plasmid Mini KitⅠ (OMEGA BIO-TEK),Stirred Ultrafiltration Cell 8050 Ultrafiltration Membrane (Millipore Corporation,Bedford, U.S.A), SP-Sepharose FF、Phenyl-Sepharose 6 FF (Pharmacia 公司),其余试剂均为国产或进口分析纯。
1.1.2 菌株与质粒
大肠杆菌Escherichia coli JM109、巴斯德毕赤酵母Pichia pastoris GS115和华根霉Rhizopus chinensis CCTCCM201021由本实验室保存,携带华根霉脂肪酶基因 proRCL的重组质粒pPIC9K-proRCL (本实验室构建),.载体 pPIC9K购自Invitrogen公司。
1.1.3 培养基
YPD:1%酵母膏,2%蛋白胨,2%葡萄糖;
MD:1.34% YNB,4×10-5%生物素,2%葡萄糖;
BMMY:1%酵母膏,2%蛋白胨,1%甲醇,0.1 mmol/L pH 6.0 磷酸缓冲液,1.34% YNB,4×10-5生物素;
BMMYA’平板:1%酵母膏,2%蛋白胨,2%甲醇,0.1 mmol/L pH 6.0磷酸缓冲液,1.34%YNB,4×10-5生物素,2%琼脂;
Fast-blue RR顶层琼脂:A:80 mg Fast-blue RR溶于1 mL二甲亚砜,B:20 mg萘酯溶于1 mL二甲基甲酰胺,取80 µL A液,180 µL B液和0.2 g琼脂于15 mL蒸馏水中,加热溶解备用。
1.2 方法
1.2.1 proRCL的易错PCR扩增
以重组质粒 pPIC9k-proRCL为模板,设计上、下游引物F和R (表1)。50 µL易错PCR体系如下:5 µL 10×易错PCR缓冲液(500 mmol/L KCl,70 mmol/L MgCl2,100 mmol/L Tris-HCl pH 8.3,0.1%(W/V,明胶));0.5 mmol/L dATP/dGTP,2.5 mmol/L dCTP和dTTP;引物F/R各40 pmol;MgCl27 mmol/L;MnCl20.3 mmol/L;Taq 2.5 U,超纯水补足50 µL。PCR扩增条件:94℃预变性3 min;94 ℃ 1 min,59 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃延伸10 min。
易错PCR扩增产物命名为promRCL,经DNA纯化试剂盒纯化备用。
1.2.2 表达载体pPIK9K的扩增
以pPIC9K为模板,以9K-short F和9K-short R (表1)为引物,扩增得到9K-short (2 000 bp)的短片段;同样以引物 9K-long F和 9K-long R(表1)扩增得到9K-long (7 226 bp)的长片段。将扩增片段9K-short和9K-long利用纯化试剂盒纯化后备用。
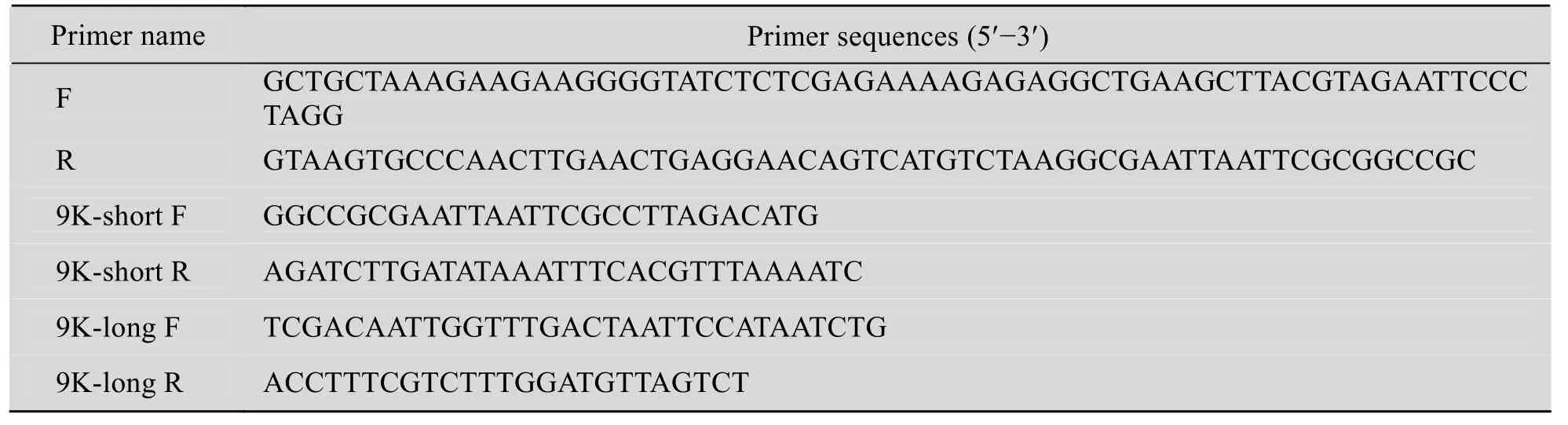
表1 本研究中用到的引物及其序列Table 1 The primers used in this study
1.2.3 基因突变文库的构建
利用 PCR依赖型方法构建高质量酵母基因突变文库[15]。将9K-short和9K-long片段等摩尔混合,再与易错 PCR扩增得到的突变基因promRCL片段按照摩尔比1∶10混合,于干燥箱中浓缩至体积<1 µL。将混合物电转化毕赤酵母感受态,转化产物涂布于MD平板上,30 ℃培养2 d,得到突变文库。以第一轮筛选得到的目的菌株的基因组等摩尔混合物作为第二轮易错PCR时的模板。
1.2.4 热稳定性突变菌株的初步筛选
用灭菌牙签将MD平板上生长出的His+转化子复制到YPD和BMMYA’平板的相同位置,同时将对照菌GS115 pPIC9k-pro’接种至BMMYA’平板上。30 ℃培养5 d。
保存生长完毕的YPD平板。将BMMYA’平板正置于60 ℃热处理60 min,冰浴15 min,室温平衡30 min。将15 mL新鲜配制的Fast-blue RR顶层琼脂均匀倾倒于处理后的各板上。2 min内颜色深于阳性对照的菌株为初筛目的菌株。
1.2.5 初筛目的菌株的复筛与鉴定
向 1.8 mL/孔 (平底)的 96孔板中加入300 µL BMGY 培养基,121 ℃灭菌 20 min。向其中接入保藏于 YPD平板上的初筛目的菌株 (同时接入 GS115 pPIC9k-pro’作为对照),30 ℃250 r/min振荡培养至OD600为2~6 (约16~18 h)。离心,弃上清,用900 µL BMMY培养基重悬菌体,并加入1% (V/V)甲醇诱导脂肪酶表达。此后每 24 h补加 100 µL BMMY 培养基和 1%(V/V)甲醇,诱导3.5 d。
将诱导表达 84 h的 96孔板发酵液3 000 r/min离心10 min,收集上清,于60 ℃热处理20 min,冰上放置15 min,室温放置30 min平衡。取1 µL上清液稀释500倍后,取5 µL于另一 96孔板中,用排枪加入底物,振荡混匀。2 min内迅速显示明显黄色的菌株为复筛目的菌株。相同条件下,对照菌 GS115 pPIC9k-pro’的发酵上清液不能显示明显黄色。
复筛目的菌株经过基因的酶切和PCR验证后,送往上海生工生物工程技术服务有限公司测序。
1.2.6 脂肪酶的诱导表达和分离纯化
将含有氨基酸突变的菌株,接种至 25 mL BMGY培养基中,30 ℃振荡培养 16~20 h至OD600为2~6,离心收集菌体,用BMMY培养基稀释至OD600为1,每隔24 h添加0.5%的甲醇诱导表达,培养3~4 d后,收集发酵上清液。
将突变菌株的发酵上清液经过10 kDa超滤膜浓缩,SP-Sepharose FF强阳离子交换层析和Phenyl-Sepharose 6 FF疏水色谱柱层析后得到突变脂肪酶活性组分proRCL。
1.2.7 突变脂肪酶酶学性质研究
利用pNPP法对纯化后的突变脂肪酶进行酶活测定[16]。在40 ℃,pH 8.5条件下,每分钟产生 1 μmol对硝基苯酚的酶量定义为一个脂肪酶水解酶活国际单位 (U)。
利用pNPP法测定纯化后的突变脂肪酶的最适pH及pH稳定性,最适温度和温度稳定性及60 ℃下的半衰期。60 ℃下的半衰期测定方法为:10 U纯化酶于60 ℃下,经不同的时间间隔取出,冰浴20 min,室温平衡5 min,测定酶活。取出后酶活为未热处理时的 50%的时间,即为酶在60 ℃下的半衰期t1/2。T50值是在某温度下放置一定时间后,使酶活降为原来的 50%的温度[17]。测定方法为:10 U纯化酶于40 ~80 ℃℃的范围中各温度下热处理30 min,冰浴20 min,室温平衡5 min后于40 ℃下测定酶活。当酶活为原来的50%时,该热处理温度即为酶的T50值。
以 pNPP作为底物测定纯化后的突变脂肪酶的Km值和Kcat值,Km值的测定采用双倒数作图法。测定过程中,底物浓度范围是25~1 000 mmol/L。
1.2.8 突变脂肪酶三级结构模拟和热稳定性机理研究
利用同源建模法 (SWISS-MODEL)对出发脂肪酶进行结构建模,同源搜索比对发现其三级结构与来自 Rhizopus niveus的脂肪酶(Protein Data Bank PDB登录号1LGY)结构同源性最高,为80.76%,以1LGY为模板酶进行同源建模。利用蛋白质模型对热稳定性机理进行分析。
2 结果与分析
2.1 易错PCR条件优化
在易错PCR中,较高浓度的Mg2+可以稳定非互补的碱基对,有利于突变。Mn2+可以降低聚合酶对模板的特异性。为了得到合适的突变率和较高的突变库容量,将Mg2+浓度和Mn2+浓度进行组合优化 (图1)。将各梯度得到的突变文库进行测序,结果表明,梯度1 (Mg2+7 mmol/L、Mn2+0.1 mmol/L)的平均突变率为0.4%,即每个基因序列平均有 4个碱基突变;梯度 2 (Mg2+9 mmol/L、Mn2+0.2 mmol/L)的平均突变率为0.6%,即每个基因序列平均有6个碱基突变。梯度3和梯度4的突变率也较高,弃用。同时,将各梯度得到的突变子接种至BMMYA’平板上,甲醇诱导4 d,用fast-blue RR染色法染色,确定各梯度平板重组毕赤酵母的表达情况。Fast-blue RR染色结果显示,梯度1中,黑色的菌落占总菌落数的80%;梯度2中,黑色的菌落占总数的65%;梯度 3和 4中,黑色的菌落占总数的<30%,弃用。
综上,梯度 1,即 Mg2+7 mmol/L、Mn2+0.1 mmol/L时突变率 (0.25%)较为可行。
2.2 表达载体pPIK9K的PCR扩增
以质粒 pPIC9K为模板,9K-longF/R和9K-shortF/R为引物,分别扩增得到两段大小分别为7 226 bp和2 000 bp的片段 (图1)。
2.3 突变文库中突变子热稳定性和酶活的关系
本研究对突变文库中突变子热稳定性和酶活的关系进行研究。在突变文库中随机抽取2 000个突变子检测其酶活和热稳定性,结果如图2。进化后,2.5%的突变子的酶活高于出发菌株,97%的突变子保留出发菌株 70%以上的酶活,只有少于0.25%的突变子在突变后酶活丧失严重。对以上随机抽取的2 000株突变体进行热稳定性初步筛选。利用96孔板发酵,得到2 000株突变体的粗酶。用pNPP法对粗酶进行热稳定性筛选。结果如图2B:约0.3%的突变酶在热处理后仍可以保持95%以上酶活,约1%的突变酶热处理后能保持90%以上酶活,约2.5%的突变酶热处理后能保持80%以上酶活。考察热处理后酶活保持在80%以上的46株突变株,0.05%的突变子的酶活和热稳定性都得到了显著提高,6.25%的突变子热稳定性提高,酶活保留出发菌株的80%以上。剩余的菌株都是负突变。以上结果表明,热稳定性的提高对酶活影响不大。
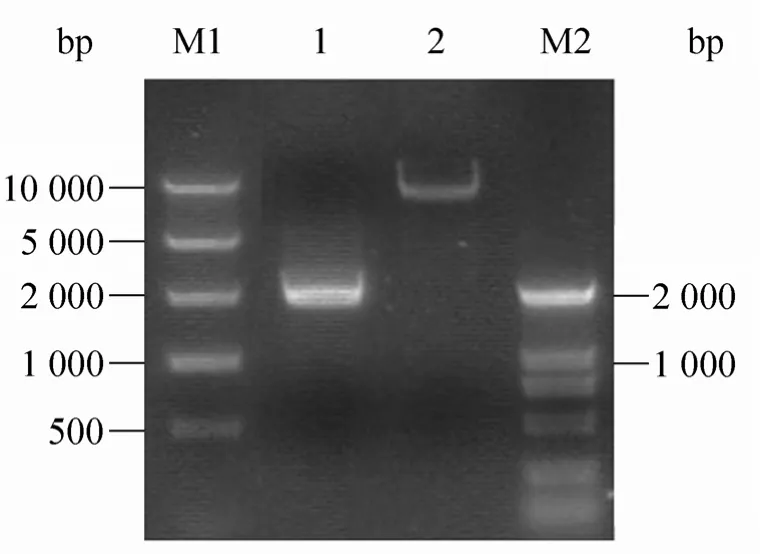
图1 表达载体pPIC9K长短片段的扩增图Fig. 1 Amplification of pPIC9K-long and pPIC9K-short. M1: E10 000 DNA Ladder marker; M2: 2 000 bp DNA Ladder marker; 1, 2 : pPIC9K-Short (2 000 bp)and pPIC9K-long (7 226 bp).
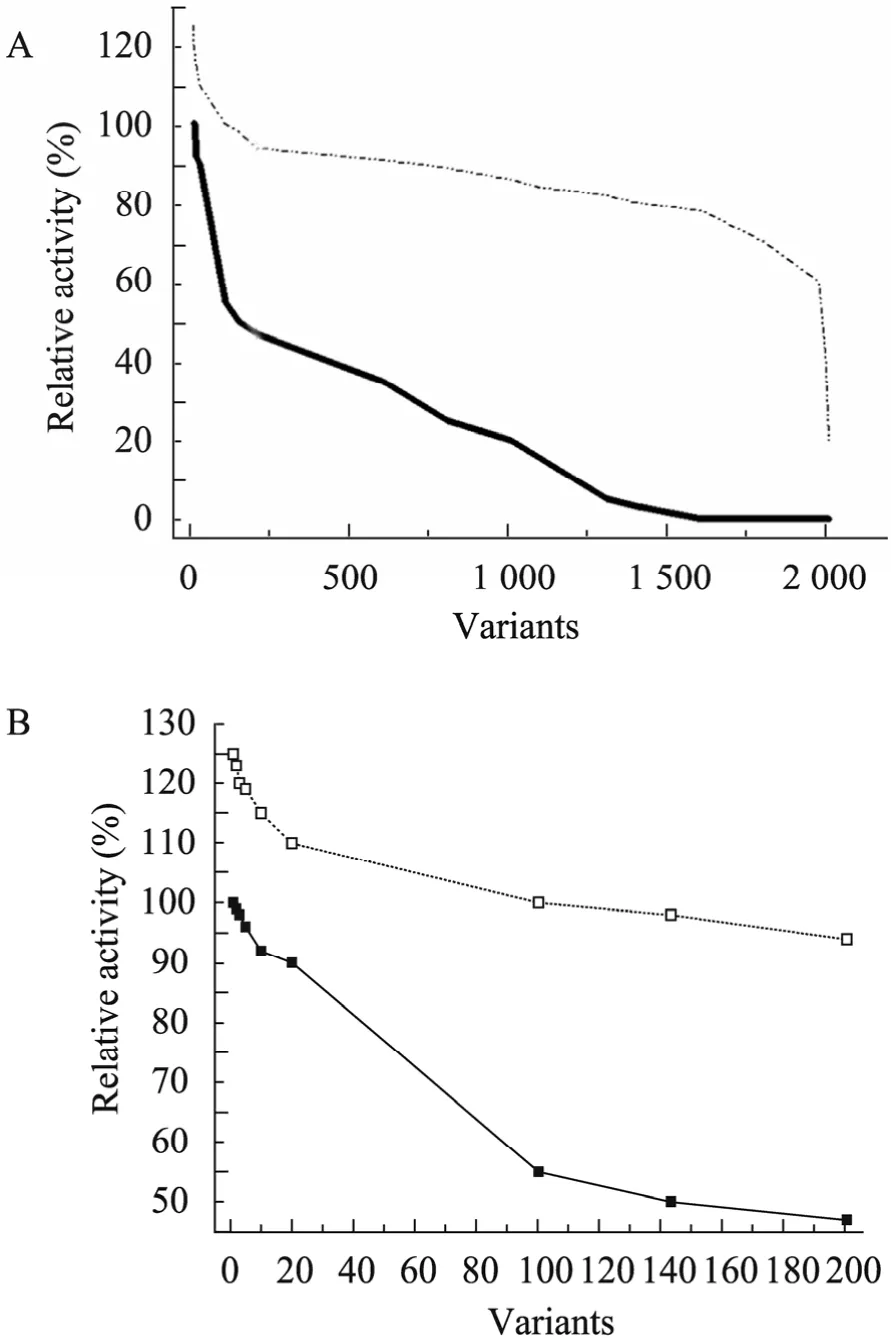
图2 定向进化构建的突变文库总览图Fig. 2 The landscape of mutant library. Dash dot line:enzymatic activity of clones in mutant library, plotted in descending order. The solid line: thermostability of clones in mutant library, plotted in descending order.Details in dotted line box in (A)were magnified in (B).For Figure (A), 2 000 clones were randomly selected from the mutant library of first generation, and their enzymatic activities were determined by 96 MTPs screening using pNPP. The enzymatic activity value of each mutant was compared to that of r27RCL, and the ratio was remarked as Y axis of dash dot line. Then, these mutant enzymes were heat-treated at 60 ℃ for 20 min,cooled in ice-bath for 20 min and in room temperature for 5 min. Their residual enzymatic activities were also measured by 96 MTPs screening using pNPP. Then,residual activity value of each mutant was compared to that of its initial activity, and the ratio was remarked as Y axis of solid line.
2.4 热稳定性突变株的初筛与复筛结果
实验共经过2轮易错PCR,突变文库容量均约为5 000株突变株左右。每轮筛选得到的突变株及其包含的氨基酸突变位点如下表。
以Fast-blue RR顶层琼脂显色进行初筛,第一轮得到目的突变体2个,共包含3个突变位点(S234F,P168L,V329A);第二轮得到目的突变体4个,共包含8个突变位点,其中有6个由第二轮易错PCR新引入(D182Y,A230F,A129S,K219D,N366D,L180H)。
2.5 突变脂肪酶的酶学性质研究
2.5.1 突变酶的分离纯化结果
对各突变酶进行纯化,纯化结果如图 3 (以ep2-4的纯化蛋白质电泳图为例)。纯酶的蛋白质分子量大小为37 kDa。
2.5.2 突变酶的最适pH、pH稳定性和最适温度
利用pNPP法对突变酶的最适pH、pH稳定性和最适温度进行测定。结果表明,相对原始酶r27RCL,各突变酶的最适pH、pH稳定性和最适温度几乎都未发生变化。
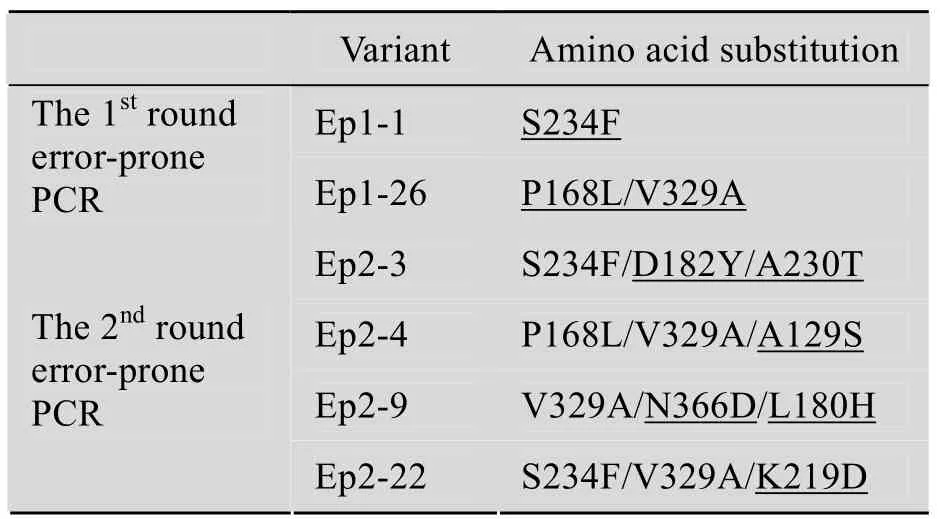
表2 易错PCR得到的突变株及其氨基酸突变位点Table 2 Lineage of variants and amino acid substitutions by error-prone PCR
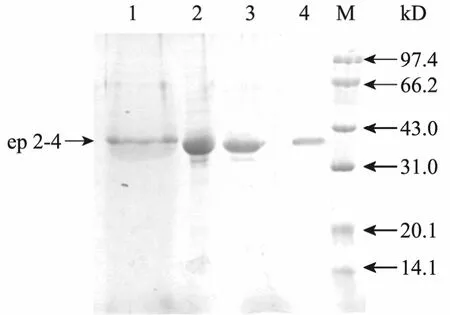
图3 突变酶ep2-4的纯化结果Fig. 3 SDS-PAGE analysis of purified mutant ep2-4. 1:culture supernatants; 2: treated with 10 kDa membrane;3: treated with SP-Sepharose chromatography; 4: treated with Phenyl-Sepharose FF chromatography; M: protein ladder.
2.5.3 突变酶的热稳定性
测定以上各轮突变酶在60 ℃下的半衰期t1/2和各突变酶的T50值。
2.5.4 突变酶ep2-4在60 ℃下的热稳定性及温度对Ep2-4的影响
考察原始酶r27RCL和最优突变酶ep2-4的热稳定性及温度对Ep2-4的影响。
如上图所示,将纯化后的原始酶r27RCL和最优突变酶 ep2-4于 20~80 ℃温度范围内处理30 min后,测定其残余酶活。r27RCL在45 ℃下放置 30 min后,酶活丧失 50%;50 ℃下放置30 min后,酶活仅存 20%。而突变酶 ep2-4于55 ℃下放置30 min后,酶活可保存53%;60 ℃下放置30 min后,还残存30%的酶活。
将纯化后的原始酶 r27RCL和最优突变酶ep2-4于 60 ℃下处理不同时间,如上图所示,r27RCL在4 min内丧失50%的酶活,10 min后丧失全部酶活。而突变酶Ep2-4在60 ℃下处理21 min还保留50%的酶活,处理50 min后才丧失全部酶活。在高温下,Ep2-4的稳定性有了显著提高。
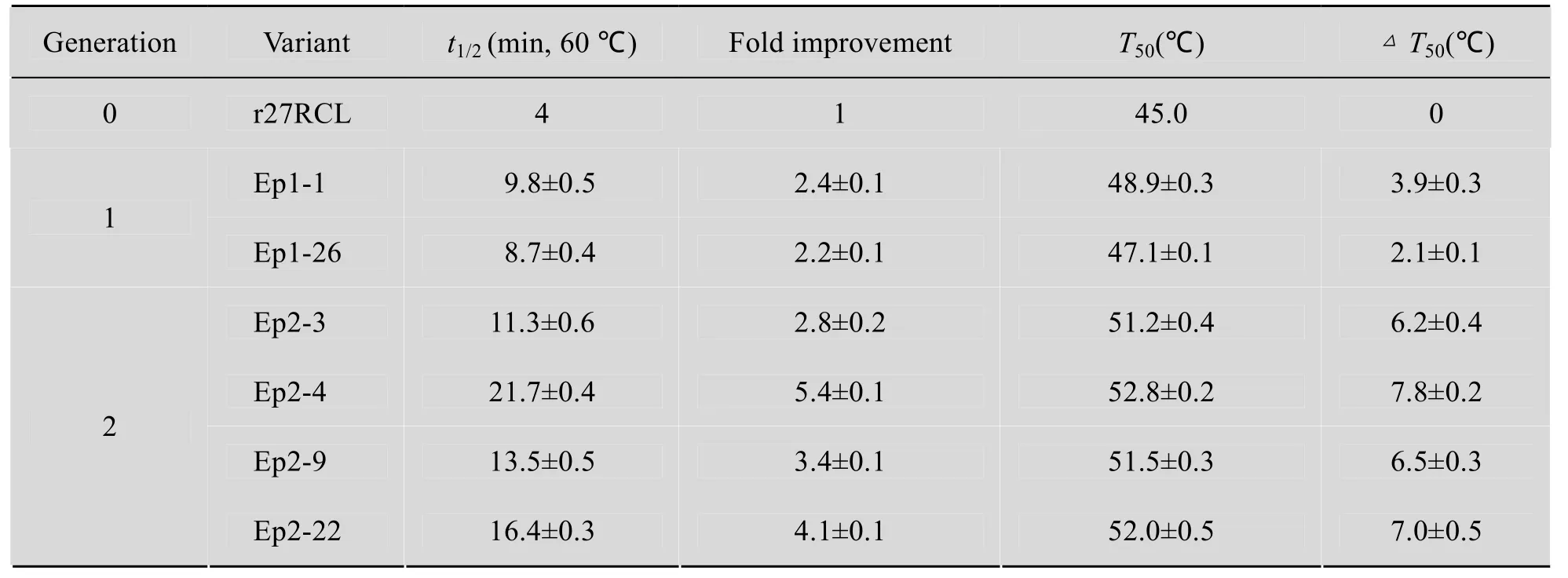
表3 原始酶和各轮突变酶在60 ℃下的半衰期及T50Table 3 Half-lives (60 ℃)and T50 of variants of each generation compared with that of r27RCL
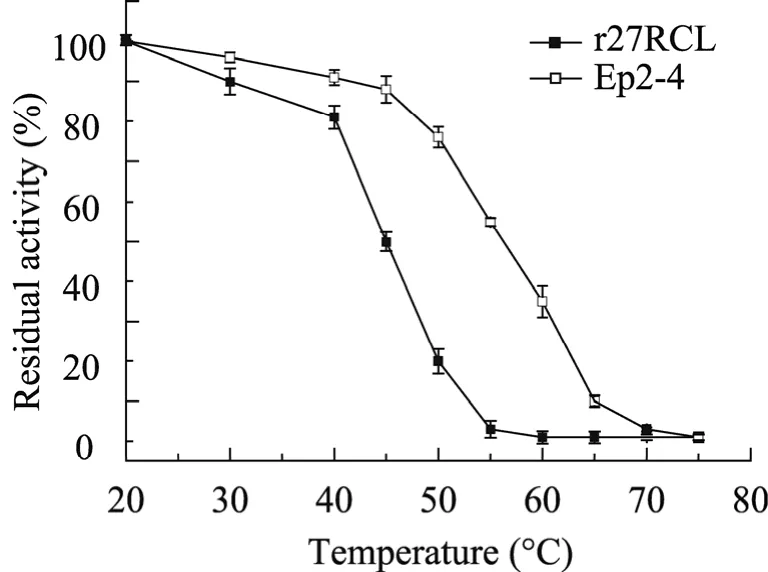
图4 温度对r27RCL和Ep2-4稳定性的影响Fig. 4 Effect of temperature on the stability of r27RCL and Ep2-4. Enzymes were incubated for 30 min at the increasing temperatures, after which residual activities were determined taking pNPP as substrate. The error bars show standard deviations calculated for three replicate experiments.
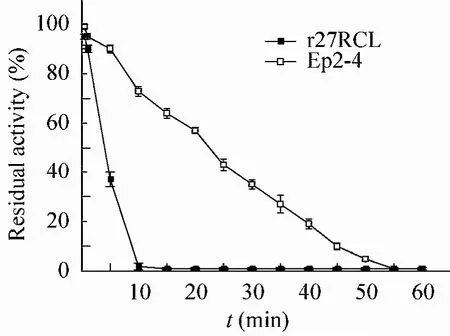
图5 r27RCL和ep2-4在60 ℃下的热稳定性Fig. 5 Thermostability of the r27RCL and Ep2-4 at 60 ℃. Enzymes were heated at 60 ℃ for increasing times, after which residual activites were determined taking pNPP as substrate. The error bars show standard deviations calculated for three replicate experiments.
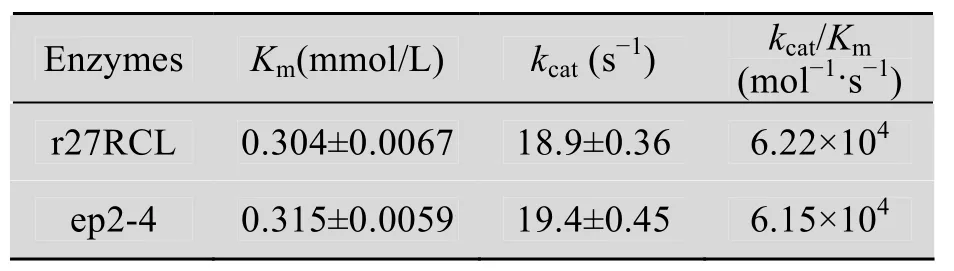
表4 原始酶r27RCL和突变酶ep2-4的动力学参数Table 4 Kinetic parameters of the parent and ep2-4
2.5.5 酶动力学分析
考察原始酶r27RCL和突变酶ep2-4的动力学参数。
以 pNPP为底物检测测定 r27RCL和 ep2-4的酶动力学参数。如上表,突变酶Ep2-4对于底物的亲和性比r27RCL下降了3.6%,转化数上升了 2.6%,总体的转化效率几乎保持不变,即突变酶的酶活没有受到影响。
2.6 突变脂肪酶的结构模拟与突变位点分析
众所周知,单个突变位点对酶热稳定性提高的作用难以精确解释,因为可以同时影响酶热稳定性的结构因素较多,单个因素的影响又很微小,而它们的综合影响却很巨大。目前,r27RCL的晶体结构还没有报道,以与其具有较高同源性的 R. niveus的脂肪酶的结构为模板,对原始酶r27RCL的结构进行同源建模,研究突变位点对酶热稳定性可能产生的影响。
对突变脂肪酶Ep2-4的三维结构进行模拟,如图6,3个氨基酸突变位点 (A129S、P168L和V329A)显示为红色,其中A129S和P168L位于蛋白质结构表面,远离酶的催化中心(S265-D324-H377)。众所周知,蛋白质热变性最初都是从蛋白质表面的部分开始的[18-19]。V329A在蛋白质内部,与活性中心接近。
突变 A129S位于蛋白质表面一个 α螺旋的尾端,该突变可能是通过增加蛋白质的亲水性和极性来提高酶的热稳定性[20]。另一个推测是突变后的 Ser129可能会与 Gln136中的氨基形成氢键,从而增强蛋白质表面的稳定性[21-22]。突变前,Ala129和Gln133在空间上没有重叠区域;突变后,Ser129和Gln133在空间上出现重叠区域,且Ser129带有的羟基中的氧与Gln133带有的酰胺基中的氢原子间距离为3.08 Å (图7),可以形成强度中等的氢键。
突变P168L位于蛋白质表面连接α螺旋和β转角的 loop上,由 Pro突变成了疏水性更强的Leu (图8)。突变前,Pro168和邻近的α螺旋上Leu164在空间上没有重叠区域 (图8A),而突变后,Leu168和Leu164在空间上出现重叠区域,推测Leu168和Leu164形成疏水键,从而增加了脂肪酶表面的疏水性,提高了酶的热稳定性[23-24]。
突变 V329A位于蛋白质内部,与活性中心接近。Val突变为Ala,会使蛋白质疏水性降低,可能会影响酶活。但是酶动力学参数表明,该突变对酶活几乎没有产生影响。目前尚不了解该突变位点对热稳定性的影响,还需要进一步的研究才能得出结论。
以上的位点对热稳定性所起的作用均为推测分析,需要后续的研究来确定分析的准确性。
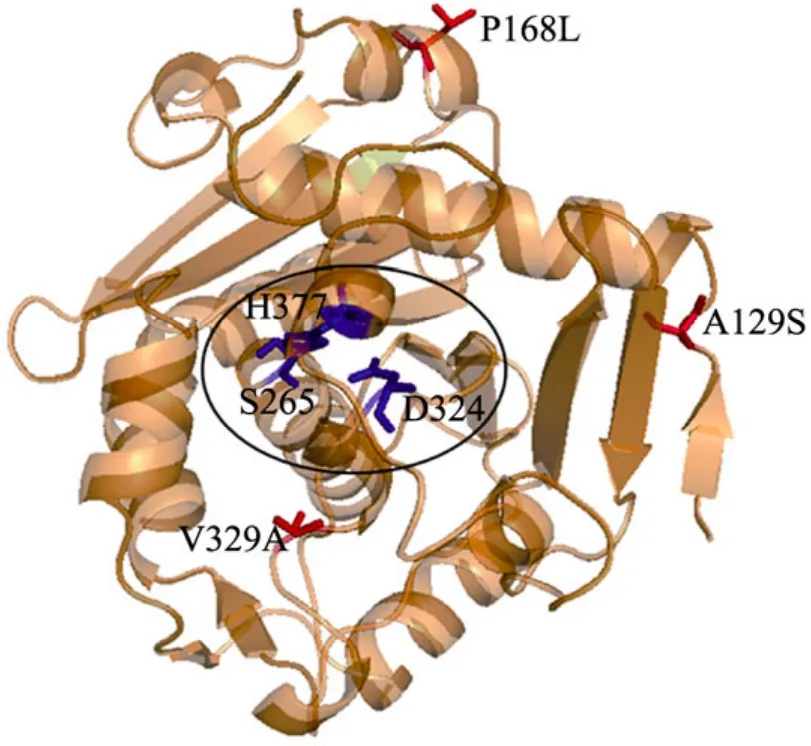
图6 突变华根霉脂肪酶Ep2-4的三维结构模拟Fig. 6 Simulted three-dimensional structure of mutant Rhizopus chinensis lipase Ep2-4. The three amino acid substitutions (A129S, P168L, V329A)in Ep2-4 were shown in red sticks and the catalytic residues were shown in blue sticks (in the oval).
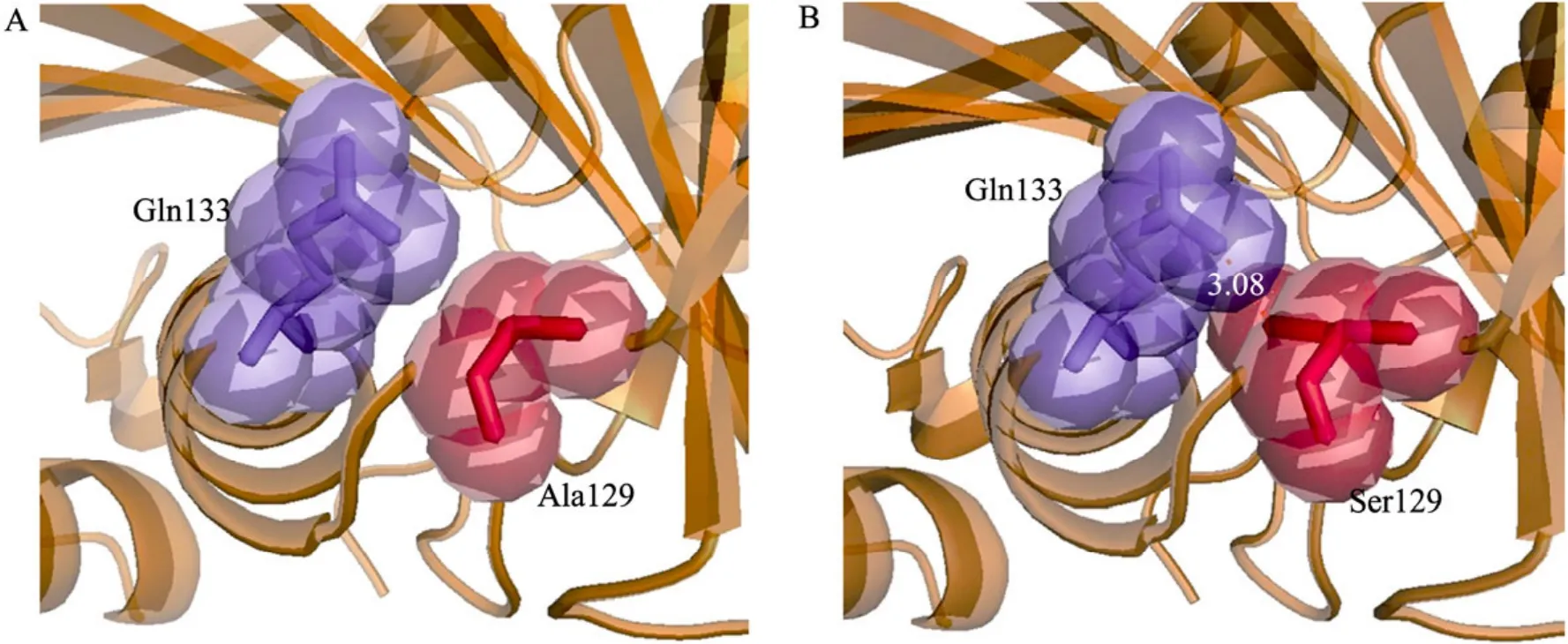
图7 位点129突变前 (A)后 (B)三维结构模拟Fig. 7 Simulated three-dimensional structure prior (A)and after (B)residue 129 muation. The red part was residue Ala129 (A)and Ser129 (B), the blue part was the nearby Gln133, formng hydrogen bond with Ser129.

图8 位点168突变前 (A)后 (B)三维结构模拟Fig. 8 Simulated three-dimensional structure prior (A)and after (B)residue 168 muation. The red part was residue Pro168 (A)and Leu168 (B), the blue part was the nearby Leu164, formng hydrophobic bond with Leu168.
3 讨论
易错PCR通过控制Mg2+、Mn2+和dNTP浓度来控制突变率。一般来说,控制合适的突变率为 0.5%~2%[25]之间。这样可以保证在获得有效突变子的同时,保证酶其他方面的能力。有报道表明,酶热稳定性提高通常要以损失酶活为代价[26-27]。但是本文通过对整个突变库中突变子酶活和热稳定性关系的研究发现,97%的突变子可以保持70%以上的酶活,可以筛选到热稳定性大幅提高、仍保持原有催化水平的突变株。
本文经过2轮易错PCR,获得了60 ℃下半衰期提高2.5倍的突变株,此突变株包含3个氨基酸突变位点。为了考察这3个突变位点单独作用时对热稳定的影响,及其组合后是加和还是协同作用,需构建这3个突变位点的单突变体和双突变体,才能精确分析各突变位点的作用。在此基础上,再利用DNA shuffling或理性设计来进一步提高酶的热稳定性。
另外,可以将突变酶进行纯化结晶,利用X-ray来进行酶结构分析和模拟。由此可以较为准确地分析出各突变位点对热稳定性的影响,为进一步改造酶的性质提供理论依据。
[1]Takahashi S, Ueda M, Atomi H, et al. Extracellular production of active Rhizopus oryzae lipase by Saccharomyces cerevisiae. J Ferment Bioeng,1998, 86(2): 164−168.
[2]Patel RN. Enzymatic synthesis of chiral intermediates for Omapatrilat, an antihypertensive drug. Biomol Eng, 2001, 17(6): 167−182.
[3]Mukherjee KD. Lipase-catalyzed reactions for modification of fats and other lipids. Biocatalysis Biotransformation, 1990, 3(4): 277−293.
[4]Gandhi NN. Applications of lipase. J Am Oil Chem Soc, 1997, 74(6): 621−634.
[5]Xu Y, Wang D, Mu XQ, et al. Biosynthesis of ethyl esters of short-chain fatty acids using whole-cell lipase from Rhizopus chinesis CCTCC M201021 in non-aqueous phase. J Mol Catal B: Enzym, 18(1):29−37.
[6]Yasohara Y, Kizaki N, Miyamoto K, et al.Enzymatically enantioselective hydrolysis of prochiral 1,3-diacyloxyglycerol derivatives. Biosci Biotechnol Biochem, 2001, 65(9): 2044−2049.
[7]Yu XW, Wang LL, Xu Y. Phizopus chinensis lipase: gene cloning, expression in Pachia pastoris and properties. J Mol Catal B: Enzym, 2009,57(1/4): 304−311.
[8]Wang TC, Yu XW, Xu Y. Expression of Rhizopus chinensis lipase induced by different temperatures and stability of recombinant protein. Ind Microbiol,2010, 40(4): 34−39 (in Chinese).王同春, 喻晓蔚, 徐岩. 诱导温度调控华根霉(Rhizopus chinensis)前导肽脂肪酶的表达及稳定性研究. 工业微生物, 2010, 40(4): 34−39.
[9]Kamiya N, Ogawa T, Nagamune T. Enhancement of apparent thermostability of lipase from Rhizopus sp. by the treatment with a microbial transglutaminase. Biotechnol Lett, 2001, 23(19):1629−1632.
[10]Niu W, Li Z, Tan T. Secretion of pro-and mature Rhizopus arrhizus lipases by Pichia pastoris and properties of the proteins. Mol Biotechnol, 2006,32(1): 73−81.
[11]Hiol A, Jonzo MD, Rugani N, et al. Purification and characterization of an extracellular lipase from a thermophilic Rhizopus oryzae strain isolated from palm fruit. Enzyme Microb Technol, 2000,26(5−6): 421−430.
[12]Beer HD, McCarthy JEG, Bornscheuer UT, et al.Cloning, expression, characterization and role of the leader sequence of a lipase from Rhizopus oryzae. Biochim Biophys Acta, 1998, 1399(2):173−180.
[13]Kohno M, Enatsu M, Funatsu J, et al. Improvement of the optimum temperature of lipase activity for Rhizopus niveus by random mutagenesis and its structural interpretation. J Biotechnol, 2001, 87(3): 203−210.
[14]Niu WN, Li ZP, Zhang DW, et al. Improved thermostability and the optimum temperature of Rhizopus arrhizus lipase by directed evolution. J Mol Catal B: Enzym, 2006, 43(1/4): 33−39.
[15]Wang R, Yu XW, Xu Y, et al. Construction of high-quality gene mutant pool in Pichia pastoris by a PCR dependent method. Chin J Biotech, 2011,27(9): 1326−1336 (in Chinese).王睿, 喻晓蔚, 徐岩, 等. PCR 依赖型方法构建高质量酵母基因突变文库. 生物工程学报, 2011,27(9): 1326−1336.
[16]Kordel M, Hofmann B, Schomburg D, et al.Extracellular lipase of Pseudomonas sp. strain ATCC 21808: purification, characterization,crystallization, and preliminary X-ray diffraction data. J Bacteriol, 1991, 173(15): 4836−4841.
[17]Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability.Angew Chem Int Ed, 2006, 45: 7745−7751.
[18]Eijsink VGH, Bjork A, Gaseidnes S, et al. Rational engineering of enzyme stability. J Biotechnol,2004, 113(1−3): 105−120.
[19]Johannes TW, Woodyer RD, Zhao H. Directed evolution of a thermostable phosphite dehydrogenase for NAD(P)H regeneration. Appl Environ Microbiol, 2005, 71(10): 5728−5734.
[20]Li W, Zhou X, Lu P. Structural features of thermozymes. Biotechnol Adv, 2005, 23(4):271−281.
[21]Eijsink VGH, Dijkstra BW, Vriend G, et al. The effect of cavity-filling mutations on the thermostability of Bacillus stearothermophilus neutral protease. Protein Eng, 1992, 5(5): 421−426.
[22]Cadwell RC, Joyce GF. Randomization of genes by PCR mutagenesis. Genome Res, 1992, 2(1): 28−33.
[23]Martin A, Kather I, Schmid FX. In-vitro selection of high stabilized protein variants with optimized surface. J Mol Biol, 2001, 309: 717−726.
[24]Martin A, Kather I, Schmid FX. Origins of the high stability of an in vitro-selected cold-shock protein.J Mol Biol, 2002, 318(5):1341−1349.
[25]Zumárraga M, Bulter T, Shleev S, et al. Alcalde M:in vitro evolution of a fungal laccase in high concentrations of organic cosolvents. Chem Biol,2007, 14(9): 1052−1064.
[26]Fernández L, Jiao N, Soni P, et al. An efficient method for mutant library creation in Pichia pastoris useful in directed evolution. Biocataly Biotransfor, 2010, 28(2): 122−129.
[27]Fromant M, Blanquet S, Plateau P. Direct random mutagenesis of gene-sized DNA fragments using polymerase chain reaction. Anal Biochem, 1995,224(1): 347−353.