全球B型流感病毒耐药相关突变株NA和HA序列特征分析
2013-09-26邢晓会李希妍成艳辉谭敏菊郭俊峰黄维娟隗合江曾晓旭王大燕舒跃龙
邢晓会,赵 翔,李希妍,成艳辉,谭敏菊,郭俊峰,黄维娟,隗合江,曾晓旭,王大燕,舒跃龙
近年来,全球每年都有B型流感病毒流行,并在部分地区引起小规模暴发,且有研究表明B型流感也能导致严重的疾病负担[1-2]。B型流感病毒有两种表面糖蛋白:血凝素(Hemagglutinin,HA)和神经氨酸酶(Neuraminidase,NA)。HA的功能是识别并结合靶细胞表面的唾液酸受体,NA的功能是催化裂解唾液酸使子代病毒释放并防止病毒形成聚集体,在这两种蛋白貌似相反的功能机制下病毒得以入侵细胞并随后释放子代病毒,HA和NA在功能上相互影响,并会发生协同突变,以利于病毒的复制和传播[3]。
NA抑制剂是目前上市的唯一对B型流感病毒有效的一类药物,包括奥斯他韦,扎那米韦等,而B型流感病毒NA基因的变异会导致毒株对该类药物的敏感性下降。随着各国耐药性监测体系的建立和完善,到目前为止,全球已经积累了相当的耐药相关突变株信息,因此有必要探究B型流感病毒耐药相关突变株的特点和规律,以获得科学依据,提高流感防控能力。
1 材料与方法
1.1 毒株 中国国家流感中心采用血凝抑制实验对省/市级疾控中心自2008年1月至2012年12月寄送的B型流感病毒进行复核鉴定,若为阳性,则选取部分毒株进行序列测定。血凝抑制实验方法参见参考文献[4]。
1.2 病毒RNA提取 使用德国QIAGEN公司Rneasy®Mini Kit,在QIACUBE全自动工作站提取病毒 RNA,100μL Rnase Free Water溶解后于-70℃保存备用。操作按试剂盒和仪器使用手册进行。
1.3 一步法逆转录聚合酶链反应(RT-PCR) 采用德国 QIAGEN 公司的one-step RT-PCR Kit,按使用手册进行操作。反应体系共25μL:Rnase Free Water 16μL,5×buffer 5μL,10mmol/L dNTP 1 μL,RNAsin 0.13μL,Enzymix 1μL,上下游引物各0.5μL,RNA模板1μL。反应条件:逆转录60℃l min,42℃20min,50℃20min,95℃15min,PCR扩增94℃30s,55℃30s,72℃1min,循环35次后,72℃10min。使用美国Affymetrix公司的USB®HT ExoSAP-IT®High-Throughput PCR Product Cleanup对PCR产物进行纯化后,用于下一步测序反应。
1.4 测序 使用美国ABI公司的BigDye®Terminator v3.1Kit。反应体系共 10μL:Rnase Free Water 5.5μL,5×Sequencing Buffer 1.5μL,引物1μL,RR-2500 1μL,PCR纯化产物1μL。测序反应条件为先96℃1min,然后循环扩增:96℃10 s,50℃5s,60℃4min,循环25次。使用ABI公司的BigDye®XTerminatorTM Purification Kit即SAMTMSolution对测序反应产物进行磁珠纯化,然后在ABI公司的3730xl全自动DNA测序仪读取序列。
1.5 序列分析和进化树分析 利用Lasergene 7.1中的SeqMan软件对DNA测序仪测得的序列进行拼接。此外,从 NCBI的Influenza Virus Database下载所需B型流感病毒的HA和NA基因序列。将我国2008-2012年测得的B型流感病毒序列、全球其他国家的B型耐药相关突变株、近期疫苗株以及国内国际同期流行株的NA核苷酸序列和对应的HA核苷酸序列分别利用MEGA5.0软件进行序列比对和分析,找出所有NA耐药相关突变位点,并通过与同期敏感株相比,找出伴随这些NA耐药相关突变的其它NA突变和HA突变。进一步利用MEGA5.0软件,采用邻位相邻法(Neighbor-Joining)分别构建HA和NA基因进化树,设置bootstrap值为2000。
1.6 突变位点在蛋白质分子模型中的标记 在蛋白质数据库(Protein Data Bank,PDB)中下载已有的B/Perth/211/2001病毒的NA蛋白晶体结构模型(PDB ID:3K36)和 B/Brisbane/60/2008病毒的HA蛋白晶体结构模型(PDB ID:4FQM),在此基础上获得B型流感病毒的NA单体和HA单体晶体结构模型,利用RasWin软件展示和标记耐药相关突变位点和伴随突变位点。
2 结 果
2.1 NA耐药相关突变位点与伴随突变位点 通过序列分析,在我国2008-2012年682株B型流感病毒中,共发现6株B型流感病毒的NA基因携带耐药相关突变。全球其他地区发现的B型耐药相关突变株共有22株,其中有4株病毒的NA突变仅为文献报道,但在NCBI中未收录其NA基因序列,本文未做分析。故本文共分析了24株病毒的NA序列和其相应的HA序列,NA耐药相关突变以及伴随的NA其它突变和HA突变位点信息总结于表1。由表1可知,我国2008年没有发现耐药相关突变株;2009年发现1株,携带H273Y突变;2010年发现3株,均携带I221T突变;2011年发现2株,分别携带I221T突变和D197N突变;2012年我国所有B型流感病毒的NA均未发生与耐药相关的位点突变。上述6株病毒中除携带H273Y突变的毒株属于B-Victoria系之外,其他5株病毒均属于B-Yamagata系。
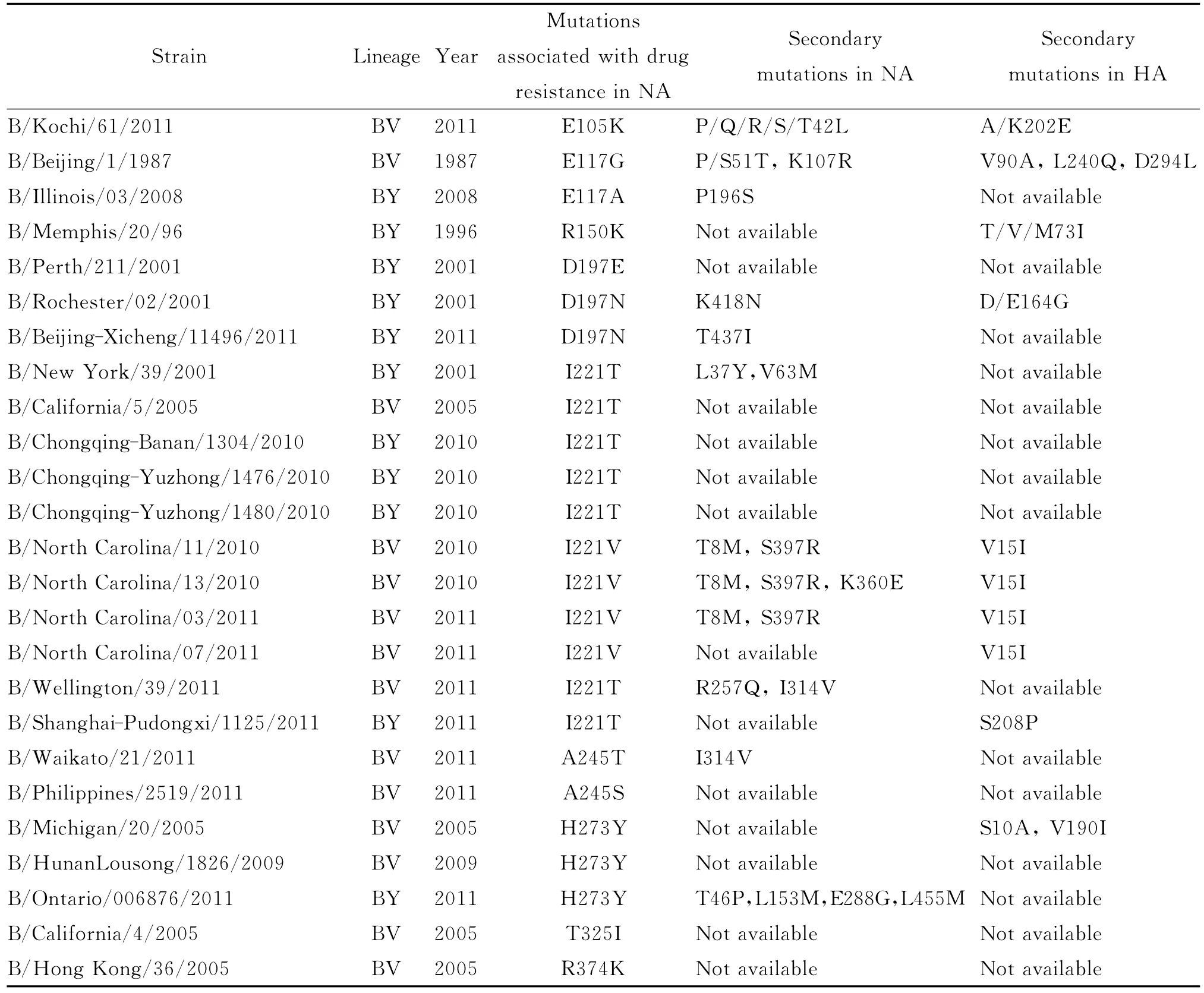
表1 B型流感病毒NA耐药相关突变位点以及NA、HA伴随突变位点Tab.1 Mutations associated with drug resistance in NA and the secondary mutations in NA or HA gene of influenza B virus
全球其他地区的耐药相关突变株最早发现于1987年,而2010-2011年发现的耐药相关突变株最多,2012年没有发现。这些突变株大多携带I221V/T、D197N、H273Y和E117G/A等常见位点突变,也有少量毒株携带 A245T/S、T325I和R374K等罕见位点突变。上述24株耐药相关突变株中有13株属于B-Victoria系、11株属于BYamagata系。
在我国重庆和上海发现的、携带NA I221T突变的4株B-Yamagata毒株,只有1株(即B/Shanghai-Pudongxi/1125/201)携 带 HA S208P 伴 随 突变,其他3株不携带任何NA或HA伴随突变;而在美国北卡罗来纳州聚集性病例中发现的、携带NA I221V突变的3株B-Victoria病毒株均伴随NA T8M、NA S397R和 HA V15I突变,其中有1株(即B/North Carolina/13/2010)还伴随 NA K360E 突变,或许正是由于这些伴随突变的存在才使得这些携带NA I221V突变的B型病毒当时能够在社区中引起暴发流行。
携带NA D197N突变的B-Yamagata系毒株B/Beijing-Xicheng/11496/2011携带 NA T437I伴随突变;同样发生NA D197N突变的B/Rochester/02/2001病毒则携带 NA K418N 和 HA D/E164G伴随突变。发生NA H273Y突变的B-Victoria系毒株B/HunanLousong/1826/2009不携带任何 NA或HA伴随突变,美国H273Y突变株B/Michigan/20/2005也为B-Victoria系,则携带2个 HA伴随突 变 S10A、V190I,而 加 拿 大 B-Yamagata 系H273Y 突变株 B/Ontario/006876/2011携带4个NA伴随突变T46P、L153M、E288G、L455M。
总体而言,我国2008-2012年发现的6株耐药相关突变株携带的伴随突变位点较少(仅有2个),而国外发现的耐药相关突变株携带的伴随突变较多,并且我国的耐药相关突变株携带的耐药相关突变大多相同,但伴随突变均不相同,提示这些伴随突变是随机发生的,也可能与地域差异有关。
2.2 NA和HA基因序列进化分析 为分析耐药相关突变株NA和HA基因的进化特征,利用MEGA5.0软件对全球(包括我国)所有B型耐药相关突变株、近期疫苗株以及国内外同期未发生耐药相关突变的流行株序列构建进化树,共计包括101株病毒的NA核苷酸序列和相应的HA核苷酸序列,结果如图1和图2所示,可见我国的耐药相关突变株大多属于B-Yamagata系,国外的大多属于BVictoria系。总体而言,我国耐药相关突变株的NA、HA序列与国外的NA、HA序列处于不同的进化分支。

图1 B型流感病毒的NA耐药相关突变位点及其NA、HA伴随突变位点Fig.1 Mutations associated with drug resistance in NA and the secondary mutations in NA or HA of influenza B virusThe mutations associated with drug resistance in NA were showed in purple font and the secondary mutations in NA or HA in black font.Mutations associated with drug resistance were showed in the same color with the corresponding secondary mutations.A:The crystal structure of NA;B:The crystal structure of HA.
2.3 耐药相关突变株的NA和HA蛋白晶体结构特点 利用RasWin软件分别展示耐药相关突变株NA和HA蛋白的表面和内部位点。图3为利用RasWin软件标记的B型流感病毒NA耐药相关突变以及NA、HA伴随突变,由该图可以看出,有些突变位点位于NA酶活性中心或HA受体结合部位,也有些位于NA、HA的结构框架位点(framework)上,还有个别突变位于NA、HA的非功能区和非结构区。在整个空间构型上,有些耐药相关突变位点与其伴随突变位点相距较远,有些则相距较近。

图2 B型流感病毒耐药相关突变株NA基因进化分析Fig.2 Phylogenetic analysis of NA gene of influenza B viruses associated with drug resistanceBlack circle--Strains associated with drug resistance isolated from China;Black triangle--Strains associated with drug resistance isolated from other countries in the world;Black square--Recent vaccine candidate strains.
3 讨 论
B型流感病毒的两种表面糖蛋白HA和NA,是影响流感病毒在体内生存的重要因素,并且其功能相互影响。Fumihiro[5]等的研究表明,发生 HA E190K突变的A(H1N1)病毒在表达NA的MDCK细胞(即NA-MDCK)中的生长情况优于其在不表达NA的MDCK细胞(即正常MDCK细胞)中的生长情况,体现了HA和NA在功能上的协同补偿机制。

图3 B型流感病毒耐药相关突变株HA基因进化分析Fig.3 Phylogenetic analysis of HA gene of influenza B viruses associated with drug resistanceBlack circle-Strains associated with drug resistance isolated from China;Black triangle-Strains associated with drug resistance isolated from other countries in the world;Black square-Recent vaccine candidate strains.
NA耐药相关突变均位于NA的酶活性功能区或结构区,这些突变的产生,会导致NA的功能受到影响,可能会降低病毒的复制和传播能力。NA耐药相关突变对病毒适应性和传播力造成的影响可依下述因素不同而不同[6]:突变位置(催化位点或者结构位点),NA型别/亚型,毒株的遗传背景,NA补偿突变的存在,NA功能的损失程度,以及适当的HA-NA功能平衡。另外,宿主免疫反应的不同以及遗传背景的差异也会对其产生影响。既往有研究表明,携带NA R150K耐药相关突变的B型流感病毒在雪貂中的感染性和传播性有所降低[7],而与此相反,携带NA D197N突变的B型流感病毒在雪貂中的生长特性与野生型相似[8],这些差异,可能是伴随突变的影响造成的。伴随突变对NA耐药相关突变株的毒株特性的影响,在A型流感病毒的相关研究较多,Jesse[9]等和 Teridah[10]等分别指出,A 型流感病毒NA发生的部分突变(如:R194G,R222Q,V234M等)以及HA发生的部分突变(如:T82K,K141E,R189K等)可对耐药相关NA突变起到一定的补偿作用,因此我们推测B型流感病毒也存在耐药相关突变位点之外的起补偿作用的位点,因此,我们对全球B型流感病毒耐药相关突变株的序列特点进行了分析,并发现了多个位于NA和HA基因的伴随突变位点。本文发现的伴随突变位点为进一步通过反向遗传学的方法验证这些位点对B型流感病毒的适应性和传播力的补偿功能打下了基础。
2011年初,美国从北卡罗来纳州的聚集性病例中,分离到14株对奥司他韦和帕拉米韦敏感性降低的B型流感病毒[11]。这提示我们,出现生物学适应性未降低的流感病毒耐药株是很有可能的,况且目前发现的耐药相关突变株其突变位点多种多样,因此应进一步在全球范围内加强对流感病毒的耐药性监测,及时发现新的耐药相关突变株,并做好流感大流行出现耐药株的应对措施。我们的研究表明,我国的B型流感病毒耐药情况与国际情况并不十分一致,故应更加密切监测我国流感病毒的耐药情况,这对于我国流感防控策略的制定和临床用药具有现实指导意义。
[1]Paul Glezen W,Schmier JK,Kuehn CM,etal.The burden of influenza B:a structured literature review[J].Am J Public Health,2013,103(3):e43-51.DOI:10.2105/AJPH.2012.301137
[2]Feng L,Shay DK,Jiang Y,etal.Influenza-associated mortality in temperate and subtropical Chinese cities,2003-2008[J].Bull World Health Organ,2012,90(4):279-288.DOI:10.2471/BLT.11.096958
[3]Myers JL,Hensley SE.Oseltamivir-resistant influenza viruses get by with a little help from permissive mutations[J].Expert Rev Anti Infect Ther,2011,9(4):385-388.DOI:10.1586/eri.11.2
[4]Kendal AP,Pereia MS,Shekel J.Concepts and procedures for laboratory-based influenza surveillance[M].Geneva: World Health Organization,1982.
[5]Gen F,Yamada S,Kato K,etal.Attenuation of an influenza A virus due to alteration of its hemagglutinin-neuraminidase functional balance in mice[J].Arch Virol,2013,158(5):1003-1011.DOI:10.1007/s00705-012-1577-3
[6]Govorkova EA.Consequences of resistance:in vitro fitness,in vivoinfectivity,and transmissibility of oseltamivir-resistant influenza A viruses[J].Influenza Other Respi Viruses,2013,Suppl 1:50-57.DOI:10.1111/irv.12044
[7]Gubareva LV,Matrosovich MN,Brenner MK,etal.Evidence for zanamivir resistance in an immunocompromised child infected with influenza B virus[J].J Infect Dis,1998,178(5):1257-1262.
[8]Mishin VP,Hayden FG,Gubareva LV.Susceptibilities of antiviral-resistant influenza viruses to novel neuraminidase inhibitors[J].Antimicrob Agents Chemother,2005,49(11):4515-4520.
[9]Bloom JD,Gong LI,Baltimore D.Permissive secondary mutations enable the evolution of influenza oseltamivir resistance[J].Science,2010,328(5983):1272-1275.DOI:10.1126/science.1187816
[10]Ginting TE,Shinya K,Kyan Y,etal.Amino acid changes in hemagglutinin contribute to the replication of oseltamivir-resistant H1N1influenza viruses[J].J Virol,2012,86(1):121-127.DOI:10.1128/JVI.06085-11
[11]Sleeman K,Sheu TG,Moore Z,etal.Influenza B viruses with mutation in the neuraminidase active site,North Carolina,USA,2010-11[J].Emerg Infect Dis,2011,17(11):2043-2046.DOI:10.3201/eid1711.110787
