山葡萄转录因子CBF1基因的克隆及序列分析
2013-09-14刘洋孙佳帝
刘洋 孙佳帝
低温是经常发生且危害严重的逆境因素之一,许多果树在经受低温胁迫后都发生不同程度的伤害,严重时甚至导致整个植株死亡。近年来,在模式植物拟南芥中发现的CBF(C- repeat/ dehydration responsive element binding factor)低温反应途径,使当前果树抗寒基因工程研究取得了重大突破。CBF转录因子能够特异结合启动子中含有CRT/DRE 的顺式元件,激活COR等基因的表达,从而提高植物的抗冻力[1,2]。本研究采用生物信息学方法对来自不同种属的植物的低温诱导基因CBF1的同源性进行分析,以此为基础克隆山葡萄低温诱导基因CBF1,并对该基因的核苷酸序列及氨基酸序列进行分析,旨在为下一步表达载体的构建及基因转化奠定基础。
1 材料与方法
1.1 材料
植物材料为吉林农业大学园艺学院选育的两性花山葡萄(Vitis amurensisRupr.)栽培品种“双优”。E.coli DH5α、表达质粒pBI121由吉林农业大学生命科学院提供。克隆载体pMD18-T vector、T4 DNA连接酶、限制性内切酶、Taq DNA聚合酶及其他工具酶购自宝生物工程(大连)有限公司;DEPC、Tris等RNA提取试剂、植物基因组DNA小量提取试剂盒、切胶回收试剂盒、DNA清洁试剂盒、DNA Marker DL2000购自杭州维特洁生化技术公司。引物合成和测序由上海生工生物工程有限公司完成,序列测定用测序仪ABIPRISMTM377DNA Sequence进行。
1.2 方法
1.2.1 山葡萄叶片组织中总RNA的提取 以水培法培养山葡萄(Vitis amurensisRupr.)枝条,待枝条长出3-5片真叶时进行4℃低温处理4h,采用CTAB法(操作方法参见《分子克隆实验指南》第3版)提取山葡萄叶片组织总RNA。
1.2.2 山葡萄转录因子CBF1基因中间片段的分离
取 5μL(≤2μg)mRNA,2μL 反转录酶,4μL buffer(5×),2μL dNTP mix(10mmol/L),0.5μL RNase Ribonuclease Inhibitor,3.5μL DEPC 水,70℃ 水浴变性5min,冰浴5min稍离心,慢慢混匀后,低速离心,收集液滴,42℃温浴60min,85℃加热10min灭活反转录酶,得到cDNA。以cDNA为模板进行PCR扩增,20μL的体系包括0.5μL cDNA,2μL buffer(10×),2μL Mg2+(25mmol/L),0.25μL dNTP(10mmol/L),0.5μL P1,0.5μL P2,0.25μL Taq 酶(5U/μL),14μL ddH2O,扩增引物见表 1。扩增程序如下:95℃温育15min后,进入热循环,第 1 个循环参数为 94℃ 45s,68℃ 30s,72℃ 30s,此后每个循环的退火温度下降1℃,共计9 个循环,然后以94℃ 45s,56℃→ 61℃ 30s,72℃30 s梯度PCR 方式运行35个循环,72℃延伸8min 结束。PCR产物经1%琼脂糖凝胶电泳回收后连接到pMD18-T载体上,转化大肠杆菌DH5α,在Amp平板上挑取重组质粒,PCR鉴定后送至大连宝生物工程公司进行测序。
1.2.3 反向PCR方法分离山葡萄转录因子CBF1基因侧翼序列 根据1.2.2分离出来的CBF1基因中间片段,设计两对特异引物(ViCBF1-F1、 ViCBF1-R1及ViCBF1-F2、ViCBF1-R2),见表1。用植物基因组DNA小量提取试剂盒(见说明书)提取山葡萄基因组DNA,应用一种在已知序列及侧翼的未知区域中没有酶切位点的限制性内切酶HindⅢ消化,消化体系 40μL,包括HindⅢ(30U)2μL,Buffer(10×)4μL,BSA(0.1%)4μL,DNA 20μL(1.5μg),dd-H2O 10μL,65℃处理18-20min,灭活内在的限制性内切酶。纯化回收DNA样品(用DNA清洁试剂盒),具体操作方法按照说明书进行,利用回收的DNA样品作为反向PCR的模板,进行PCR扩增,扩增产物在1%琼脂糖凝胶中电泳分离,回收目的片段进行克隆测序。
1.2.4 山葡萄转录因子CBF1基因全长DNA及cDNA的克隆 根据方法2.3得到的山葡萄转录因子CBF1基因的侧翼序列,在其侧翼序列设计一对特异引物(ViCBF1-F3 、ViCBF1-R3),见表 1。以山葡萄基因组DNA为模板进行PCR扩增,扩增产物在1%琼脂糖凝胶中电泳分离,回收目的片段进行克隆测序。
在ViCBF1基因的ORF设计一对特异引物(ViCBF1-F4 、ViCBF1-R4)(表 1)。以反转录 cDNA为模板进行RT-PCR扩增,获得山葡萄CBF1基因的全长cDNA 序列。

表1 克隆CBF1基因所用引物
1.2.5 山葡萄转录因子CBF1基因的序列分析 山葡萄转录因子CBF1基因序列相似性比较在http://ncbi.nlm.nih.gov网站上用BLAST进行,根据目的基因核苷酸序列用Primer 5.0软件推导出目的基因的氨基酸序列,并进行序列分析。
2 结果
2.1 山葡萄总RNA的提取与mRNA的分离
山葡萄植物细胞中含酚类化合物,并且多糖含量比较高,为此在本试验在CTAB原方法的基础上增加PVP(聚乙烯吡咯烷酮)含量,因为PVP中的CO-N=基有很强的结合多酚化合物的能力,能有效地去除多酚对RNA提取的干扰,从而获得纯度较高的山葡萄RNA,如图1所示,23S 核糖体RNA 与18S核糖体RNA 的带型清晰锐利,它们的亮度比基本上呈2∶1 关系,不存在蛋白质、多糖和酚类物质的明显干扰。
2.2 山葡萄转录因子CBF1基因中间片段及侧翼序列的获得
以mRNA反转录产物cDNA为模板进行电泳,通过PCR获得cDNA的扩增片段,将扩增产物重组到pMD18-T克隆载体上进行测序,测序结果为758bp。根据序列信息,设计两对侧翼序列嵌套引物,反向PCR获得一长度约为750bp的片段(图2);测序结果为753bp(图3)所示,其中有546bp是与上述已经分离出来目的基因中间片段的重叠区域(图3中的阴影部分即是)。图3中的方框部分是新扩增出来的片段,长度为206bp,双下划线部分的6bp是HindⅢ酶切位点,HindⅢ的实际酶切位点是AAGCTT,而在本试验中由于此酶产生了星号活性,此位点变为ATGCTT(双下划线),剩余的200bp新扩增出来的片段,其中有94bp是目的基因的5'端片段(即方框中双下划线右侧序列,106bp目的基因的3'端片段(即方框中双下划线左侧序列)。

图1 4℃低温处理4h山葡萄的RNA的提取
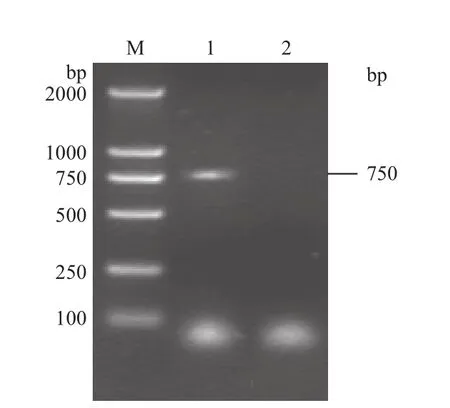
图2 RT-PCR产物的琼脂糖凝胶电泳

图3 反向PCR测序结果
2.3 山葡萄转录因子CBF1基因全长DNA及 cDNA的克隆
以山葡萄基因组DNA为模板,引物ViCBF1-F3、ViCBF1-R3(表1)。进行PCR扩增,得到的PCR产物比较特异,无杂带,无降解现象(图4)。以山葡萄mRNA反转录产物cDNA为模板进行电泳,引物ViCBF1-F4、ViCBF1-R4(表1),PCR获得cDNA的扩增片段,如图5所示。测序结果所得到的序列与我们用基因组扩增所得到的序列在编码区内完全一致,这说明山葡萄转录因子CBF1基因在编码区内没有内含子。与所报道的其他植物中CBF1基因在编码区内没有内含子的结论完全一致。过程受此区域的控制。位于C末端的转录激活区酸性氨基酸含量较高,位于第237-251个氨基酸之间,其pI在3.6-3.9之间,这一区域在转录激活中起主要作用。
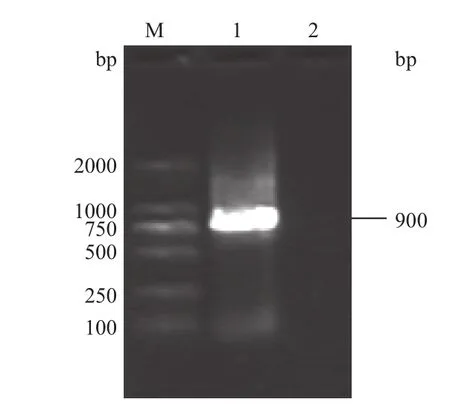
图4 目的基因全长的PCR分析
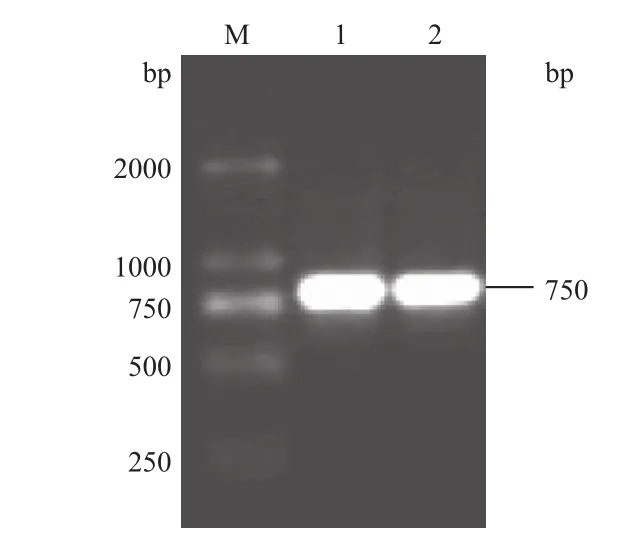
图5 目的基因的cDNA克隆的PCR产物
2.4 山葡萄转录因子CBF1基因核苷酸序列及推导的氨基酸序列分析
山葡萄转录因子CBF1基因序列全长为910bp,编码区长为753bp,无内含子。山葡萄转录因子CBF1基因的核苷酸序列及推测的氨基酸序列,如图6所示。CBF1转录因子属于AP2/EREBP类转录因子,其一级结构中含有AP2 DNA结合域(图6中的阴影部分),它由79个氨基酸残基组成,在不同的植物中非常保守,山葡萄转录因子CBF1基因与其他几种植物中的CBF1基因的同源性主要归因于它们的AP2 DNA结合域的高度同源。预测的碱性核定位信号位于N末端区域,位于第36-75个氨基酸之间,富含精氨酸和赖氨酸,CBF1转录因子进入细胞核的

图6 山葡萄转录因子CBF1的核苷酸序列及其相应的氨基酸序列
2.5 山葡萄转录因子CBF1基因的氨基酸序列分析
根据开放阅读框的密码子翻译,推测该基因编码一个含有251个氨基酸的多肽,分子量约为27.503kD,等电点偏低,pI=3.11,属酸性蛋白。
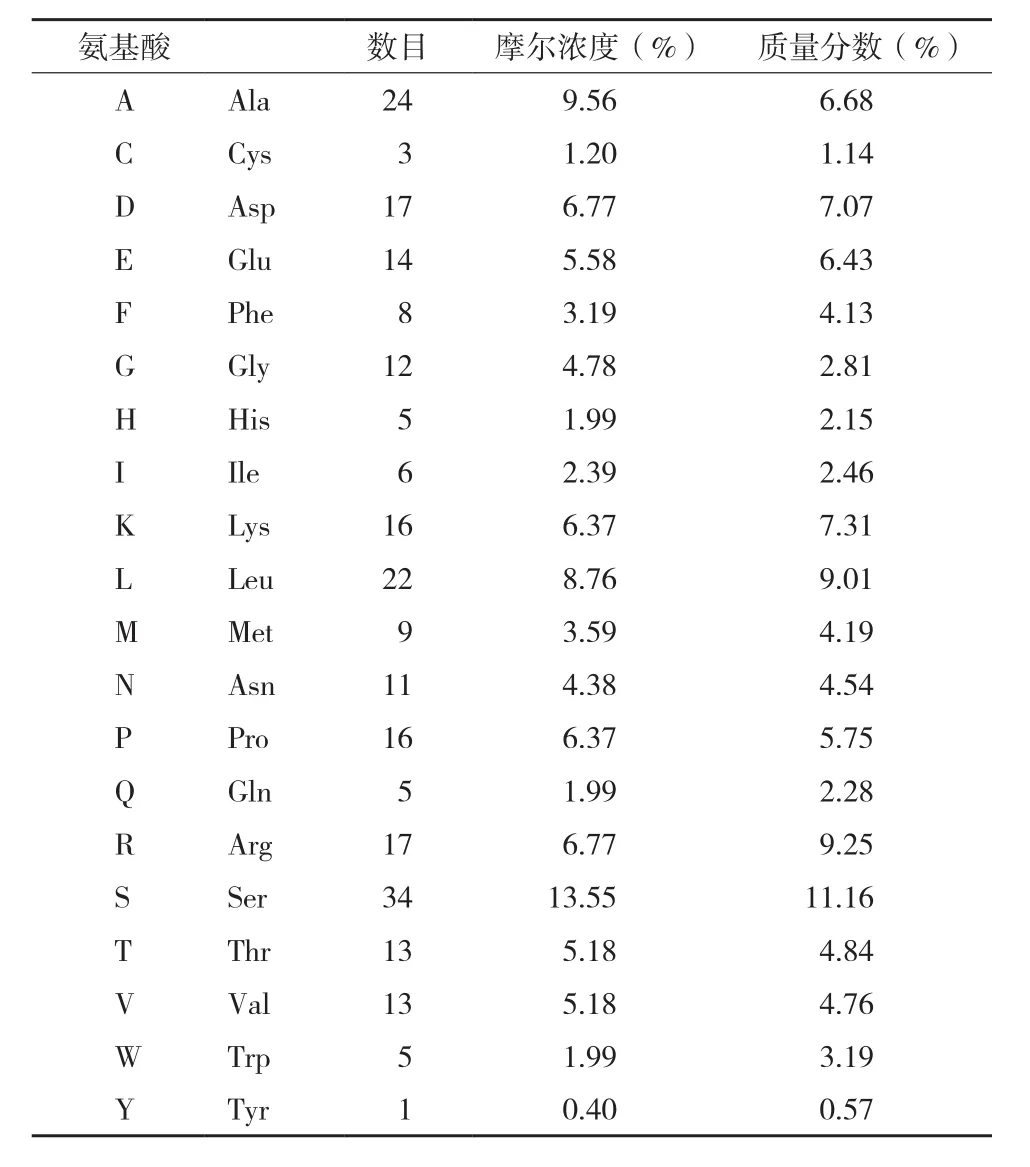
表2 氨基酸成分组成成分
通过氨基酸组成分析得知,山葡萄转录因子CBF1基因中丝氨酸的浓度较高,达到13.55%,而碱性氨基酸赖氨酸和精氨酸的浓度分别为6.37%和6.77%,酸性氨基酸谷氨酸和天冬氨酸的浓度分别为5.58%和6.77%(表2)。
2.6 山葡萄转录因子CBF1基因氨基酸序列比较
Blast软件分析表明,山葡萄转录因子CBF1基因与其他几种植物的CBF1有着很高的相似性,其中与茄科辣椒属中的簇生椒(Capsicum annuum)、盐芥(Thellungiella salsuginea)、橡胶树(Heavea brasiliensis)、葡萄(Vitis vinifera)、拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)苹果(Malus×domestica)、大麦(Hordeum vulquare)、马蔺(Iris lactea var. chinensis)等植物的CBF1基因的氨基酸同源性达到80.7%。如图7所示,在不同植物中AP2/EREBP区域内是高度保守的,它含有1个由约60-70个氨基酸残基组成的非常保守的DNA结合域(DNA-binding domain),主要功能是调节低温、干旱、高盐等胁迫条件下的应答反应。而且在AP2/EREBP结构域的上游和下游分别有两段保守序列,即PKK/RPAGRxKFxETRHP和DSAWR,在预测由拟南芥编码产生的140多种 AP2/EREBP域蛋白中,只有CBF1、CBF2和CBF3才具有这两段短多肽序列,因而称其为CBF蛋白的特征序列,而在AP2/EREBP区域的两侧氨基酸序列总体上看没有明显的同源性。

图7 十种植物低温诱导转录因子CBF1基因的氨基酸序列比较
3 讨论
近 3年来,在拟南芥[3]、油菜[3]、水稻[4]和番茄[5]等植物中发现了CBF1转录激活因子,本研究采用生物信息学方法对所能获得的低温诱导植物的CBF1基因序列进行了系统分析,进而获得了不同低温诱导植物的CBF1基因的共同结构域,藉此合成兼并引物,并以山葡萄叶片组织为材料,采用RT-PCR和反向PCR技术成功获得了山葡萄转录因子CBF1基因,在GenBank上的登录号为DQ517296,并采用Blast软件分析对大麦(Hordeum vulquare)、水稻(Oryza sativa)等10种低温诱导植物的CBF1基因的氨基端序列进行了比对,其同源性达到80.7%,这也说明基因进化的保守性。该研究的核心技术在于:兼并引物的设计及兼并引物条件下PCR条件的设置。该方法与惯常使用的同源序列法的不同点在于以大量低温诱导植物的CBF1基因的相似性为基础,采用梯度PCR方法,无须要求引物序列的完全匹配,对同源性的要求较低。此外,本研究采用反向PCR的方法一次性获得目的基因的侧翼序列即非编码区部分,该方法只需要提取基因组DNA进行单酶切和自身环化,从而简化了分离未知基因的步骤。
比较分析现有的低温诱导的植物CBF1基因序列发现,CBF1基因中含有高度保守的AP2/EREBP结 构 域[6,7],AP2/EREBP 家 族 的 转 录 因 子 是 与环境胁迫应答反应等有关的一系列转录因子。在山葡萄AP2/EREBP域的上游和下游存在PKK/RPAGRxKFxETRH和DSAWR特征序列,在拟南芥[8]编码产生的140多种AP2/EREBP域蛋白中,只有CBF1、CBF2、CBF3才具有这两段短多肽序列,两序列在进化程度不同的物种[9,10]中保守存在。因此,推测这两段序列为CBF蛋白的特征序列,在CBF基因抗寒性功能研究中发挥非常重要的作用。结合本研究的结果及CBF1同源基因研究方面的文献,可以推测这样一种可能:即存在一种和CBF 蛋白相似的蛋白质与编码去饱和酶的基因的启动子中某一元件结合,促进编码去饱和酶的基因的表达,去饱和酶使膜脂的不饱和度增加,从而最终增加膜脂在低温条件下的流动性和稳定性,增强植物的抗寒力。
4 结论
本研究设计了一对兼并引物,采用RT-PCR及反向PCR技术克隆了山葡萄转录因子CBF1基因全长,其ORF长为753bp,无内含子。经序列比较分析,该基因属于AP2/EREBP类转录因子,其一级结构中含有AP2DNA结合域,它由79个氨基酸残基组成,在不同的植物中较保守。
[1]Thomashow MF, Gilmour SJ, St ockinger EJ, et al. Role of theArabidopsisCBF transcriptional activators in cold acclimation[J]. Physiol Plant, 2001, 112:171-175.
[2]Gilmour SJ, Zarka DG, Stockinger EJ, et al. Low temperature regulation of theArabidopsisCBF family of AP2transcriptional activator as an early step in cold-induced COR gene expression[J]. Plant J,1998, 16(4):433-442.
[3]Jaglo KR, Kleff S, Amundsen KL, et al. Components of theArabidopsisC-repeat/dehydration element binding factor cold-response pathway are conserved inBrassica napusand other plant species[J].Plant Physiol, 2001, 127:910-917.
[4]Dubouzet JG, Sakuma Y, Ito Y, et al. OsDREB genes in rice, Oryza sativaL.encode transcription activators that function in drought-,high-salt-and cold-responsive gene expression[J]. Plant J, 2003,13(5):356-359.
[5]Hsieh TH, Lee JT, Charng YY, et al. Tomato plants ectopically expressingArabidopsisCBF 1 show enhanced resistance to water deficit stress[J]. Plant Physiology, 2002, 130:618-626.
[6]Riechmann JL, Meyerowitz EM. The AP2/EREBP family of plant transcription factors[J]. Biol Chem, 1998, 379:633-646.
[7]Okamuro JK, Caster B, Villarroel R, et al. The AP2domain of APETALA2defines a large new family of DNA binding proteins in Arabidopsis[J]. Proc Natl Acad SciUSA, 1997, 94:7076-7081.
[8]Kanaya E, Nakajima N, Morikawa K, et al. Characterization of the transcriptional activator CBF1 fromArabidopsis thaliana:evidence for cold denaturation in regions outside of the DNA binding domain[J]. Biol Chem, 1999, 274(23):16068-16076.
[9]Novillo F, Alonso JM, Ecker JR, et al. CBF2/DREB1C is a negative regulator of CBF1/DREB1B and CBF3/DREB1A expression and plays a central role in stress tolerance inArabidopsis[J]. Proc Natil Acad SciUSA, 2004, 101(11):2985-2990.
[10]Gilmour SJ, Sebolt AM, Salazar MP, et al. Overexpression of theArabidopsisCBF3transcriptional activator mimics multiple biochemical changes associated with cold acclimation[J]. Plant Physiology, 2000, 124:1854-1865.
