基于复合EST-SSR标记的大白菜种子纯度鉴定及SNP位点获取
2013-09-03兰青阔贺长征李欧静娜朱郭永泽
赵 新 王 永* 兰青阔 贺长征 陈 锐 李欧静 刘 娜朱 珠 郭永泽
(1天津市农业质量标准与检测技术研究所,天津 300381;2天津市种子管理站,天津 300061)
大白菜〔Brassica campestrisL. ssp.pekinensis(Lour)Olsson〕为十字花科芸薹属蔬菜,作为我国主要的蔬菜作物在农业生产中占有重要位置。大白菜的种植区域分布广泛,种植数量较多,近几年对大白菜种子纯度的监督抽查工作较为重视,种子质量的好坏直接影响到种子生产者、经营者和使用者的合法权益。因此,加强大白菜种子纯度的控制和检查管理尤为重要。传统的田间形态鉴定是种子纯度鉴定的仲裁方法,但时间长、易受环境条件的影响,因此为适应种子纯度快速鉴定的需求,室内分子检测技术成为近年来研究的热点。
随着分子生物学标记技术的发展,人们开始利用分子标记技术进行种子纯度的研究,ESTSSR技术是其中的一种。EST-SSR技术又叫基于表达序列标签(expressed sequence tags,EST)的简单重复序列(si mple sequence repeats,SSR)标记技术,与其他分子标记技术相比具有技术操作简单、特异性较强、重复性稳定、多态信息含量高、共显性遗传等特点(Powell et al.,1996;忻雅 等,2006)。同时GenBank数据库中积累了大量重要物种的EST序列,且获取方便,无需任何费用,已经成为发展SSR标记的重要来源,因此EST-SSR技术不仅保留了SSR标记技术的优点,而且凸显了其便捷、花费低、效率高的优势(Wim et al.,2009);但SSR技术也存在着相对的不足,即一次试验得到的位点较少,针对这一问题,Tommasini等(2002)发展了多重SSR分析技术,并有效地鉴定了油菜品种。
EST-SNP技术也叫基于表达序列标签的单核苷酸多态性(Single Nucleotide Polymorphism,SNP)标记,是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性,是继SSR之后的新一代分子标记技术,具有密度高、稳定性好、分析易自动化等特性,近年来被广泛应用于甜瓜、菜豆、甜椒等蔬菜分子辅助育种及品种鉴定研究(Carlos et al.,2009;Wim et al.,2009;Jung et al.,2010;兰青阔 等,2012)。
本试验以10份大白菜杂交种及其亲本为试材,利用新型复合EST-SSR分子标记进行大白菜杂交种纯度鉴定的研究,筛选杂交种特异谱带为亲本互补带型的引物,建立一次试验可获得多位点的标记方法,快速、高通量、低成本,确保了种子纯度鉴定结果的可靠性。同时基于EST序列信息,结合HRM和Pyrosequencing技术,初步探索了大白菜SNP位点的筛选、确认和检测技术路线,旨在为建立精确、快速、高通量和高度自动化的大白菜种子纯度鉴定方法提供技术参考。
1 材料与方法
1.1 材料
试验于2011年12月至2012年11月在天津市农业质量标准与检测技术研究所遗传育种分析实验室进行,以10份市售大白菜杂交种(表1)为试验材料,种子由天津科润蔬菜研究所提供。
1.2 方法
1.2.1 单粒种子育苗条件 本试验通过1台简单的恒温培养箱来模拟种子萌发外界条件,达到在最短时间内成功育苗的效果,以提高试验效率。具体方法如下:采用玻璃培养皿,以棉花、纱布垫底,用自来水将其湿润后均匀撒入单粒种子,为持久保持种子湿润度,在种子表面再覆盖一层面巾纸,夜间在28 ℃恒温培养箱中过夜,白天采用室温日光灯补光,48 h种子破壳率可达100%,7 d后发芽率可达100%。
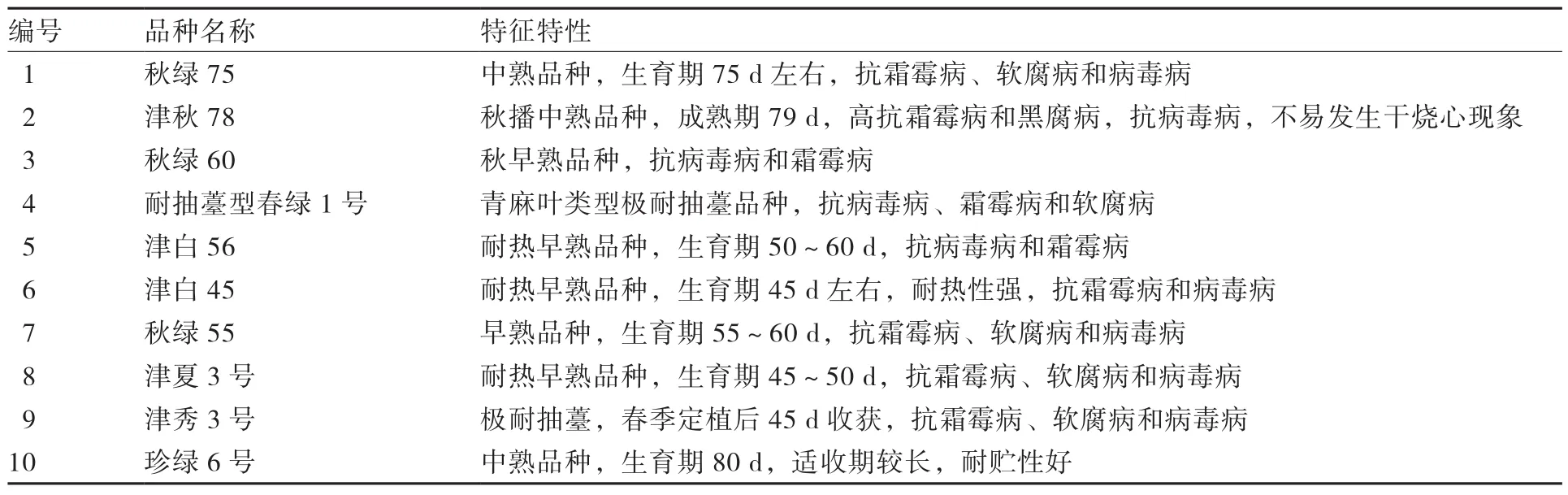
表1 供试品种名称及特征特性
1.2.2 DNA提取 选取经48 h刚刚破壳的大白菜种子和经7 d达到苗期的单株叶片,同时应用改良CTAB法进行DNA提取,经1%凝胶电泳检测,比较DNA提取质量。具体改良CTAB法如下:取单粒破壳种子或单株叶片粉碎,加入CTAB-A液(裂解缓冲液)550 μL,65 ℃水浴30 min;加入氯仿异戊醇500 μL,颠倒混匀,13 200 r·min-1离心10 min;吸取上清液500 μL,加入2倍体积CTAB-B液(沉淀缓冲液),室温静置30 min;13 200 r·min-1离心10 min;弃上清液,沉淀中加入350 μL CTAB-C液,回溶;加入350 μL氯仿异戊醇,颠倒混匀,13 200 r·min-1离心10 min;吸取上清液340 μL,加入0.7倍体积预冷的异戊醇,颠倒混匀,13 200 r·min-1离心10 min;弃上清液,沉淀中加入500 μL 70%乙醇清洗,13 200 r·min-1离心10 min;弃上清液,沉淀风干,加入30~40 μL TE缓冲液充分溶解后即为DNA。
1.2.3 高通量快速DNA提取方法 为适应大批量种子纯度鉴定工作的需要,对种子高通量快速DN A提取方法进行比对,包括chexe-100法、碱煮法、CTAB-A液直接裂解法。3种方法都分别设置20、40、60、120 min 4个时间梯度,具体方法如下:① chexe-100法。使用电动组织研磨器将单粒种子或单株叶片破壁打碎至粉浆状,加入50 μL chexe-100,65℃水浴,设置时间梯度;-20℃冷冻10 min,13 200 r·min-1离心3~4 min,上清液即为DNA。② 碱煮法。使用电动组织研磨器将单粒种子或单株叶片破壁打碎至粉浆状,加入50 μL NaOH(1 mol·L-1),100 ℃煮沸,设置时间梯度;加入 25 μL Tris-HCl(1 mol·L-1,pH 8.0),100 ℃煮沸 5 min;-20℃冷冻10 min,13 200 r·min-1离心3~4 min,上清液即为DNA。③ CTAB-A液直接裂解法。使用电动组织研磨器将单粒种子或单株叶片破壁打碎至粉浆状,加入50 μL CTAB-A液,65 ℃水浴,设置时间梯度;-20 ℃冷冻10 min,13 200 r·min-1离心3~4 min,上清液即为DNA。
1.2.4 EST-SSR引物设计与合成 利用GenBank(http://www.ncbi.nlm.nih.gov/sites/entrez)上公布的170 148条大白菜EST序列,通过CMD SSR Server(http://www.cottonmarker.org/cgibin/cmd_ssr)在线设计30对核心引物,命名为BC1~BC30,引物由上海生工生物工程技术服务有限公司合成。
1.2.5 复合EST-SSR引物筛选 以大白菜杂交种及其亲本的基因组DNA为模板,分别用30对EST-SSR引物进行扩增,产物用8%聚丙烯酰胺凝胶电泳检测。 筛选出杂交种特异谱带为亲本互补带型的引物,同时结合单一引物扩增结果及特异谱带的相互位置差异,优化组合多个引物对,筛选复合EST-SSR标记。
1.2.6 PCR扩增体系及程序 PCR扩增反应的总体积为10 μL:2×GoTaq®Master Mixes 5 μL,上游和下游引物(10 μmol·L-1)各0.25 μL,大白菜基因组DNA模板(50 ng·μL-1)1 μL,双蒸水补足10 μL。PCR反应程序为:94 ℃预变性5 min; 94 ℃变性1 min,53.5 ℃退火45 s,72℃延伸45 s,35个循环;72 ℃延伸7 min,4 ℃保存。
1.2.7 EST-SNP位点获取 基于两种技术路线探索EST-SNP获取方法。方法一:分析ESTSSR引物聚丙烯凝胶电泳结果,回收亲本互补条带并测序,经序列比对寻找SNP候选位点,设计引物,通过焦磷酸验证,确定Baicai-9 SNP位点。方法二:通过NCBI数据库查询芸薹属SNP位点,设计PCR引物,对大白菜杂交种及其亲本进行扩增,通过高分辨率溶解曲线分析候选SNP位点,扩增产物送上海生工生物工程技术服务有限公司测序,应用生物学软件对序列进行Blast比对,对候选13 799 SNP位点设计焦磷酸引物进行验证。
2 结果与分析
2.1 单粒种子育苗条件及提取状态确定
从图1和图2可以看出,从48 h破壳的大白菜种子和经7 d达苗期状态的大白菜单株提取的基因组DNA均表现主带清晰,整齐一致,没有明显降解,可用于下一步PCR扩增。
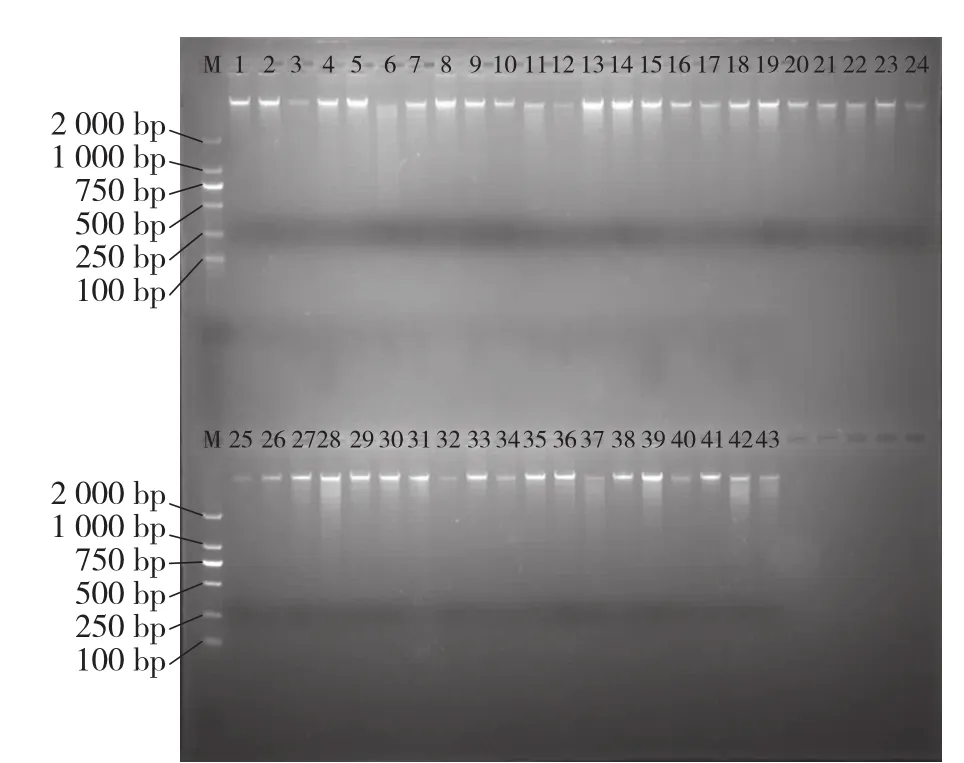
图1 48 h破壳的大白菜基因组DNA电泳结果
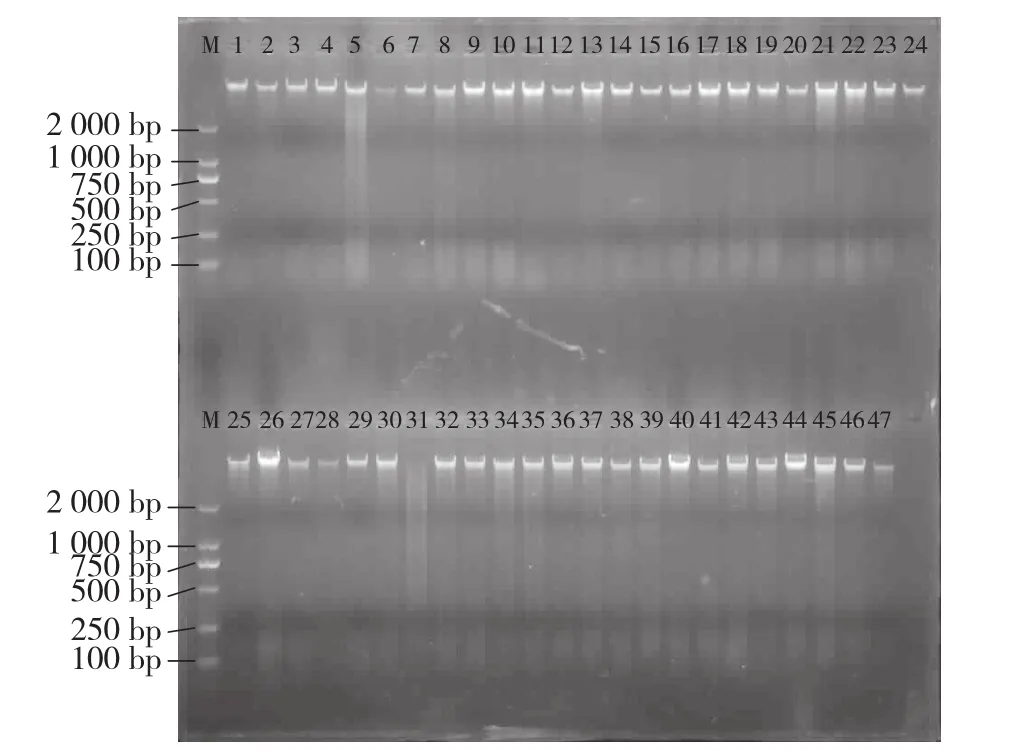
图2 经7 d达苗期的大白菜基因组DNA电泳结果
2.2 引物及复合EST-SSR引物筛选结果
以10份大白菜杂交种及其亲本为模板,对30对EST-SSR引物进行筛选,其中7对引物的杂交种带型为双亲的互补带型,可用于大白菜杂交种种子纯度的鉴定。选取其中条带清晰、特异性主带位置差异明显的3对引物,用于鉴定种子纯度。对这3对引物进行优化组合,确定了一套复合EST-SSR引物,可同时对10份大白菜杂交种进行种子纯度鉴定。引物序列及可鉴定品种见表2。应用这套复合EST-SSR引物对10份大白菜杂交种进行纯度鉴定的聚丙烯酰胺凝胶电泳图谱如图3所示。由表2和图3均可得出杂交种1(秋绿75)、3(秋绿60)、5(津白56)可同时应用3对引物对其纯度进行鉴定,既提高了鉴定效率,也增强了纯度鉴定结果的可靠性。

表2 引物序列及可鉴定品种名称
2.3 高通量快速DNA提取方法比较结果
3种高通量快速DNA提取方法提取大白菜津白56经48 h破壳状态的单粒种子和经7 d达到苗期状态的单株叶片基因组DNA,经1%琼脂糖电泳均未见DNA条带,但以chexe-100法提取经7 d达到苗期状态的单株叶片的DNA为模板,经PCR扩增可达到EST-SSR理想的鉴定效果,其他2种方法提取的DNA均未扩增出相应条带,结果如图4所示。且3种方法所需单株状态均为苗期,可见种子破壳初期的状态尚难以满足高通量快速DNA提取的需要。
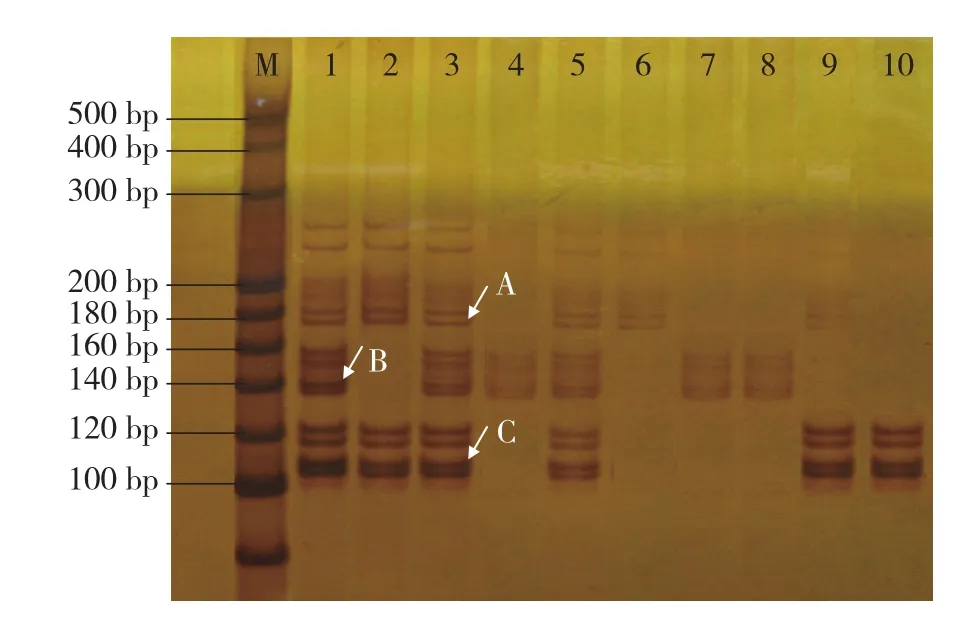
图3 10份大白菜杂交种复合EST-SSR引物纯度鉴定结果
2.4 Baicai-9 SNP位点获取
2.4.1 SNP候选位点的获得 应用引物BC-9扩增双亲,以回收的特征谱带DNA为模板进行PCR扩增,产物送上海生工生物工程技术服务有限公司测序,拼接测序结果,对双亲特异性条带的序列进行比对,通过碱基差异寻找SNP候选位点,结果如图5所示。
2.4.2 SNP引物设计与合成 应用焦磷酸引物设计软件,对SNP候选位点进行引物设计,引物由上海生工生物工程技术服务有限公司合成。引物序列见表3。

图4 不同提取方法提取津白56 DNA的复合EST-SSR引物扩增结果
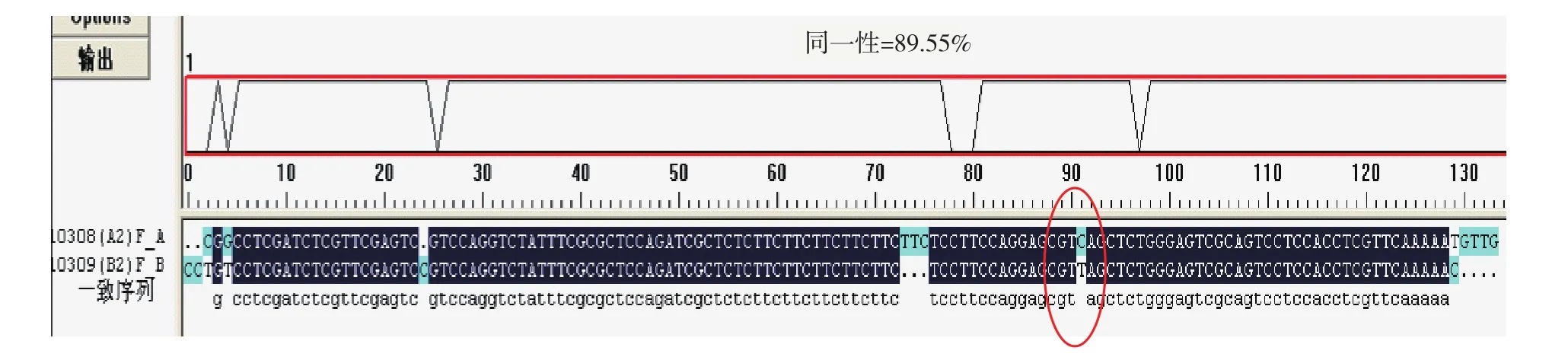
图5 引物BC-9扩增大白菜亲本序列同源性比对结果
2.4.3 SNP位点的验证 通过焦磷酸测序引物(正向和反向引物)对秋绿75、秋绿60、耐抽薹型春绿1号、津白56、秋绿55、津夏3号的亲本及杂交种进行前期PCR扩增。PCR扩增反应的总体积为50 μL:2×GoTaq®Master Mixes 25 μL,上游和下游引物(10 μmol·L-1)各1.25 μL,基因组DNA模板(50 ng·μL-1)3 μL,双蒸水补足 50 μL。PCR反应程序为:94℃预变性5 min;94 ℃变性1 min,53.5 ℃退火 45 s,72 ℃延伸 45 s,40个循环;72 ℃延伸7 min,4 ℃保存。
测序单链的制备:将3 μL链亲和素包被的磁珠和47 μL binding buffer混匀,然后加入到50 μL的PCR产物中,1 300~1 400 r·min-1室温下振荡混匀10~15 min;预先在PSQ Low反应板中加入40 μL含有0.3 μmol·L-1测序引物的Annealing Buffer;在Vacuum prep workstation中进行单链制备,备用。再根据软件提示,在试剂舱相对应位置加入所需的底物,酶和A、C、G、T。焦磷酸测序反应碱基喷入次序(dispensation order)为C/TAGCTCTGGGA。由C-T碱基峰高与后续AGCTC碱基峰高比较可以看出,SNP分型为C/T,筛选得到的SNP位点可对6份大白菜杂交种进行纯度鉴定,测序结果如图6所示。
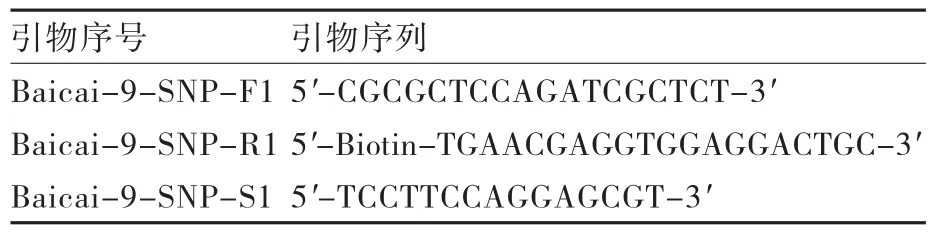
表3 SNP引物序列
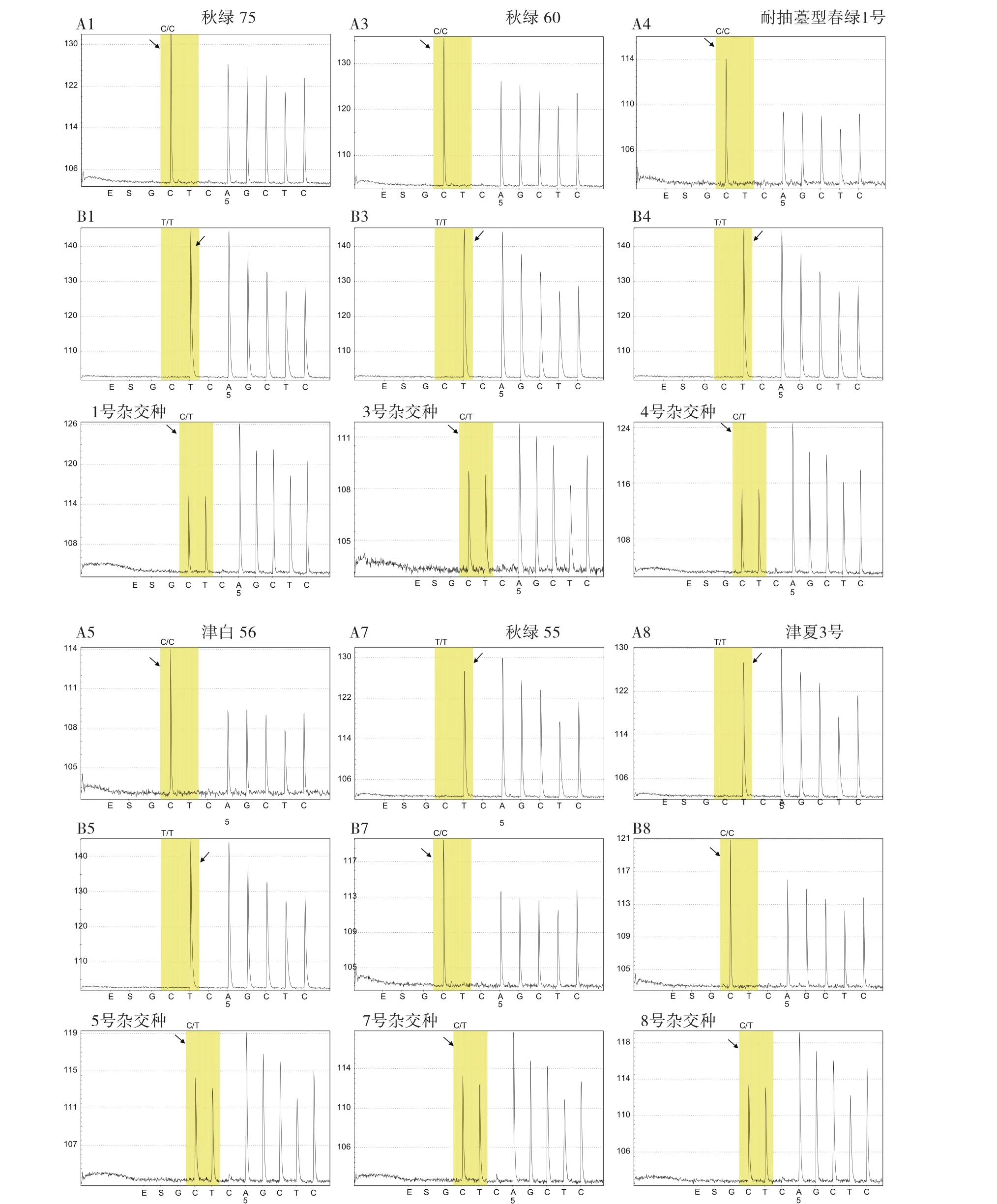
图6 6种大白菜杂交种SNP鉴定结果
2.5 13 799 SNP位点获取
通过NCBI数据库查询芸薹属SNP位点,设计PCR引物,对大白菜杂交种及其亲本进行扩增,扩增产物送上海生工生物工程技术服务有限公司测序,应用生物学软件对序列进行Blast比对,对候选位点设计SNP焦磷酸引物,进行验证,通过津夏3号验证13 799是否为SNP位点,能否用于杂交种纯度鉴定。
2.5.1 依据芸薹属SNP位点设计PCR引物 通过查询CuGenDB数据库,获得Contig 13 799序列信息,该序列在94 bp 处具有C/T/A多态性的候选SNP位点、在96 bp 处具有C/T/G多态性的候选SNP位点、在107 处具有C/T/G多态性的候选SNP位点。使用LightScanner Primer Design software设计用于高分辨率熔解曲线技术扫描分析(HRM)的PCR引物,上游引物序列为 H13799-F:5′-TTACTTTCGCCGACACAAAC -3′,下游引物序列为 H13799-R:5′- ATGTGCTCCTATGTAACCCA-3′。
2.5.2 测序及SNP引物设计 依据HRM扫描分析结果,将候选SNP位点送上海生工生物工程技术服务有限公司测序,依据Contig 13 799序列信息,使用PSQ Assay Design软件设计一套焦磷酸测序引物,包括PCR上游引物P13799-F:Biotin-5′-CAACCATTGCTGATTAGAGTAACA-3′、下 游 引 物 P13799-R:5′-TTGATTTAGCGTTAGGGAGTCTG -3 ′ 及 测 序 引 物 P13799-Seq:5′-GCTAGTGAGACCGTTAAGAT-3′。
2.5.3 焦磷酸测序结果 依据HRM扫描分析结果,对候选SNP位点进行PCR扩增和焦磷酸测序确认。焦磷酸测序反应碱基喷入次序(dispensation order)为GC/TCTAGC,其中第1位碱基G和第5位碱基A为阴性对照。图7为焦磷酸测序结果,由C、T碱基峰高与后续CTAGC碱基峰高比较可以看出,SNP分型为C/T,命名该SNP位点为13 799。该位点与采用2.4中回收双亲特征谱带测序的技术路线,所获取的津夏3号SNP位点实为大白菜品种的同一SNP分子特异性标记位点。
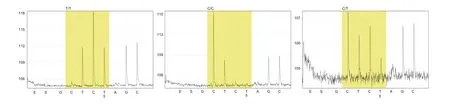
图7 津夏3号SNP鉴定结果
3 结论与讨论
随着基因组数据库的不断完善以及分子辅助育种技术的突破性进展,使得种子真实性鉴定及纯度检测的田间形态学标记正逐步被分子标记技术所替代。与传统形态学性状鉴定相比,基于DNA水平差异的EST-SSR标记技术,具有稳定性高、共显性、重复性好等特点,而且开发成本相对低廉(赵新 等,2013),更重要的是其检测不受基因表达和环境条件的影响,可以在植株生长的任何阶段进行,大大提高了检测效率,可以在育种初期及时有效地保证育种的质量,避免造成不必要的经济损失。
近年来,通过不断研究大白菜基因组序列信息和SSR位点特异性分子标记,丰富了相关数据信息,但SNP位点特异性分子标记长期以来未见研究报道,在一定程度上滞后了分子标记等现代育种手段在大白菜育种和种子纯度鉴定上的应用(鲁忠富 等,2012)。本试验基于EST序列信息,通过两种技术路线,成功获取到大白菜品种同一SNP分子特异性标记位点,更加确保了位点的可靠性,同时应用获取EST-SNP位点的方法和思路,结合分子生物学基础信息研究,通过进一步的测序、筛选,可以获取更多的EST-SNP特异性位点,对更多的大白菜品种或其他物种进行标记和鉴定,为分子辅助遗传育种课题组下一步工作奠定了基础。
然而,对于种子纯度鉴定,无论是田间形态学还是分子标记技术,其纯度结果与检样量基数密切相关,为确保检测结果的准确性,必须保证充足且科学的检样量基数,一般一个品种的检测数量不得少于60株单株。因此,为适应高通量种子纯度检测的需要,本试验对EST-SSR标记复合体系进行了筛选,根据单一引物的相互位置差异进行优化组合,使单一引物的扩增片段处在不同片段大小的区域,避免复合标记结果的重叠性,同时对提高检测效率的相关环节也进行了优化。复合EST-SSR标记对10份大白菜杂交种纯度鉴定具有很好的区分效果,通过一次反应最多可同时检测到3个不同的多态性标记位点,既有效地降低了试验成本,同时又加大了试验结果的可靠性。通过对提高检测效率的相关环节的摸索,确定了最佳育苗条件及DNA提取方法,对于高通量苗期单株DNA的提取可在40 min内完成,在一定程度上有效地节省了检测时间,降低了人力物力的消耗。
兰青阔,张桂华,王永,赵新,朱珠,崔兴华,郭永泽,程奕.2012.基于SNP标记的黄瓜杂交种纯度鉴定方法.中国蔬菜,(6):58-63.
鲁忠富,徐沛,吴晓花,汪宝根,王莎,刘永华,李国景.2012.SSR分子标记技术在瓠瓜种子纯度快速鉴定中的应用.浙江农业学报,24(4):578-581.
忻雅,崔海瑞,卢美贞,姚艳玲,金基强,林容杓,崔水莲.2006.白菜EST-SSR信息分析与标记的建立.园艺学报,33(3):549-554.
赵新,王永,兰青阔,陈锐,李欧静,余景会,朱珠,郭永泽.2013.复合EST-SSR标记在大白菜品种鉴定中的应用.生物技术通报,(1):107-110.
Carlos H G,Marcela G,Lina M R,Matthew W B.2009.CEL I nuclease digestion for SNP discovery and marker development in common bean(Phaseolus vulgarisL.).Crop Sci,49:381-394.
Jung J K,Park S W,Liu W Y,Kang B C.2010.Discovery of single nucleotide polymorphism inCapsicumand SNP markers for cultivar identification.Euphytica,175(1):91-107.
Powell W,Machray G C,Provan J.1996.Polymorphism revealed by simple sequence repeats.Trends Plant Science,1:215-222.
Tommasini L,Batley J,Arnold G M,Cooke R J,Donini P,Lee D,Law J R,Lowe C,Moule C,Trick M,Edwards K J.2002.The development of multiplex simple sequence repeat (SSR) markers to complement distinctness,uniformity and stability testing of rape (Brassica napusL.) varieties.Theor Appl Genet,106:1091-1101.
Wim D,Cristina E,Cristina R,Mireia G T,Iria F S,Daniel G I,José B,Miguel A A,Pere A,Fernando N,Antonio J M,Maria B P,Jordi G M.2009.A set of EST-SNPs for map saturation and cultivar identification in melon.BMC Plant Biology,9:1-9.
