三线性分解算法对液相色谱-质谱联用仪多样本测定数据分辨适用性的比较
2013-07-13张树荣吴海龙翟敏康超尹小丽俞汝勤
张树荣,吴海龙,翟敏,康超,尹小丽,俞汝勤
(化学生物传感与计量学国家重点实验室湖南大学化学化工学院,湖南长沙 410082)
复杂的基于蛋白质的生命现象及其过程已经难以用一两个因素来简单、孤立地描述。传统的荧光探针技术因为受自身光谱分辨能力所限而造成信号重叠,已经无法同时描述多个蛋白质的生命功能。液相色谱-串联质谱联用(LC-MS)技术已经被证明是最有可能承载蛋白质组系统性研究的基础方法之一。目前,基于LC-MS 技术的Shotgun 策略[1-4]已经能够充分利用二级质谱来准确地识别肽段序列,从而识别体系中重要的蛋白质。当蛋白质识别的问题得到很好的解决之后,科研工作者开始前瞻性地考虑蛋白质的定量问题——如何定量地从直接的生物标记蛋白质来描述生命现象及其过程。Shotgun策略虽然擅长于识别蛋白质,自身却非常耗时[5],不能采集到体系中完整的数据[6],也不能同时给出定量性质的结果[1,7-10]。在常规领域,质谱仪器的MRM(multiple reaction monitoring)技术是很好的兼顾鉴定同时保证定量的方案。而肽段本身的二级质谱会出现非常多的碎片离子信号,造成了各个碎片离子未必像常规小分子那样具有一个主导的碎片峰,从而使得选择定性和定量离子的工作非常繁琐,且进一步使电离条件的优化复杂化。同一个肽段质谱会受到多种因素影响,其中包括流动相条件、电离源技术、碎裂策略、环境因素等等,这又使得在一台仪器上获得的结果很难推广到其它基于质谱的定量工作中。MRM 方案具有特殊性,其普适性不明显。而在高丰度肽段共存条件下对低丰度目标肽段的定量分析,MRM 方法依然要面临考验。同时,目前一维色谱(1DLC)的峰容量是不能完全分离蛋白质组学研究对象当中所含有的多肽成分;尽管全二维色谱(2DLC)的峰容量理论上能够达到2500[11,12],而实际上能识别的蛋白质大概在50个,对于数以十万计的多肽数目依然偏少。
然而,质谱的数据特性和一般仪器的信号特性如紫外可见光、荧光等存在一定的差异。某物质的纯质谱信号具有稀疏性,即质荷比信号为正值,且不连续。本文就常规三线性分解算法能否适应这样的数据特性进行探讨,从而提出可行的解决方案。
1 实验数据集
本文利用人血清白蛋白(HSA)水解的动力学体系构建具有代表性的数据集合。将HSA(购自Sigma-Aldrich 公司)用超纯水(UP water)配成25 g/L的工作液备用。配制酸性氯化钙溶液(浓度为0.005 mol/L,用盐酸调节pH 为3),用于配制胰蛋白酶(trypsin)储存液。用酸性氯化钙溶液配制牛胰蛋白酶(购自Aladdin 公司)为2 g/L的储存液。将氯化钙溶于Tris-HCl 缓冲液(pH 8.2)中配成浓度为0.005 mol/L的碱性氯化钙溶液(alkaline CaCl2)。最后实验样品的组成见表1。在线取样用Agilent 1290 Infinity Autosampler 来实现,动态水解样品采取每隔30 min 取样一次。对中等反应速度样本(medium-digest-rate-sample)重复取样30次,对高反应速率样本(high-digest-rate-sample)和低反应速率样本(low-digest-rate-sample)各重复采样15次。
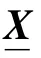
表1 实验样品的配制与组成Table 1 Composition of the samples
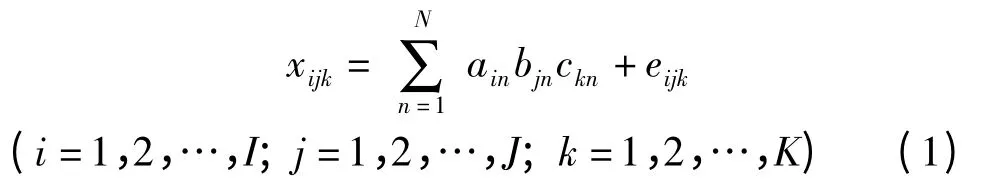
三线性分解算法的目标是将具有这样的三线性结构的数据最终分解为各个含有纯组分信息的矩阵(如图2所示)。根据算法的目标函数不同,有PARAFAC(parallel factor analysis)[24-26]、ATLD(alternating trilinear decomposition)[14]、SWATLD(selfweighted alternating trilinear decomposition)[27]和APTLD(alternating penalty trilinear decomposition)[28]等算法可用于这样的数据数学分离。这些系列算法的目标都是最大限度地拟合三维响应数阵,挖掘其中的定性、定量信息,因而具有很强的普适性,并且不需要人为设置一些条件参数。而对于双线性方法,本研究则选择了相关作者提供的MCR(multivariate curve resolution)工具箱进行计算。
对于MCR 方法,尽管本研究尝试了所有可能的参数设置,依然无法将目标肽段的低丰度信号与背景进行有效的分离。也就是说,对于低丰度肽段的分析,双线性方法几乎不适用。前面提及的多种三线性分解算法的应用结果,其色谱图(B 矩阵)和定
2 计算结果与讨论
在上述动力学采样的数据中,本研究选择了15.80~15.98 min 一段具有代表性的信号作为各个算法的测试基准。该段数据含有一个低丰度肽段(经识别,其序列为IAEVENDEMP),其响应约为实验中主要峰强度的1/100,并且淹没在基线波动当中,是典型的低丰度肽段信号(如图1所示)。该段数据是典型的干扰强、目标分析物响应低的双组分三维数阵。
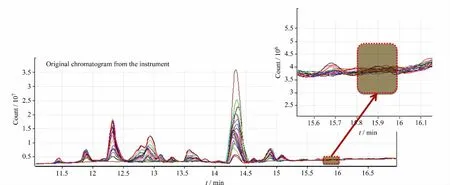
图1 评价算法所选取的数据区间Fig.1 Range of retention time selected to test the algorithms
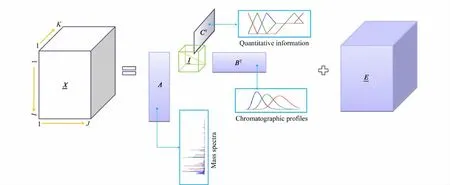
图2 三线性分解的示意图Fig.2 Diagram of trilinear decomposition
三维数阵内含的三线性关系可以用公式(1)表量信息(C 矩阵)的直观结果是可以接受的,将背景干扰和目标肽段的信号分开了。但质谱图的质量却与客观实际不相符合(见图3),在肽段的质谱信号本来为0的地方出现了明显的负值,并且显示出与色谱背景信号负相关的关系。这些经典的算法在寻求对三维数阵最佳拟合的同时,的确没有考虑到质谱数据的稀疏特性。因而这样获得的结果显然有些不符合实际。
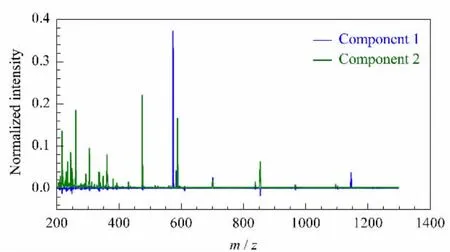
图3 常规三线性方法分解结果中表现出来的对质谱数据特性的不适应性(以ATLD 结果为例)Fig.3 Inadaptability of trilinear decomposition algorithm using the results of ATLD as an example(The resolved components contained obviously negative values)
要提高质谱的三线性分解质量,必须要在原来的三线性分解算法基础上引入一些自动的约束条件,在提高结果质量的同时减少人为操作,以便于高通量的数据处理。而与质谱图的稀疏特性相对应的数学约束为“非负约束”,引入非负约束应该能改善算法的结果质量。对于PARAFAC 算法,在较早前已经有研究者提出了各种非负求解的方案。Bro 等[29]曾提出展开方式求解,但是由于LC-MS 数据是非常庞大的,采用展开成矩阵或者向量求解的策略,即使使用64位的操作系统,依然会耗尽软件的内存地址编码数目。Paatero[30]则提出了通过引入惩罚项的方案来实现非负计算,然而惩罚项部分需要非常复杂的计算,不但衍生了更多的临时变量,还附带了很多对三维阵的计算操作,因而该方案的计算时间消耗非常大。可见,基于PARAFAC 算法的改进会产生对计算资源和时间的严重消耗,这样的算法不适宜推广。根据ATLD算法的计算策略具有计算资源节约和收敛快的特点,本文从ATLD 衍生出了新颖的带有非负约束的算法NNATLD(non-negative alternating trilinear decomposition)。新算法交替优化求解公式(2)中的目标函数,从而最终获得良好的数学分辨结果。新的算法不但使用切片矩阵的方案,还对矩阵进行了有效的压缩,所以非常节约计算资源,同时具有非常快的收敛速度。新算法对于本组测试数据会在10次迭代内收敛到最终结果,比以往的算法都要快。新算法能自动给出组分数估计的建议,用户只要给出比实际组分数多的估计,算法就能自动寻找合理的组分数。
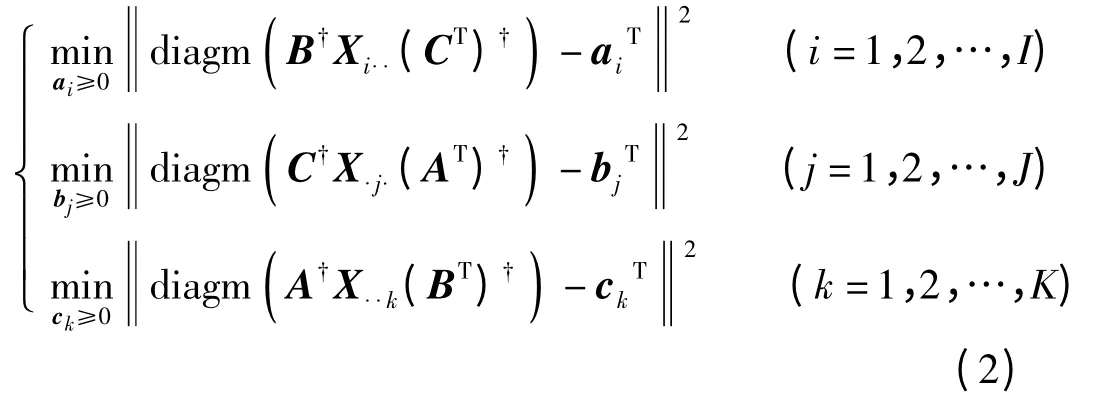
在计算结果方面,新算法能获得非常好的色谱和定量结果,且质谱图的质量能较好地符合定性的客观要求(见图4)。新算法不但满足了质谱图的稀疏性特性,同时也符合肽段质谱图的结果并与理论的肽段质谱图吻合较好。在低丰度肽段的信号保留方面,新算法保留了m/z 1168.5的[M+Na]+准分子离子峰。该峰的强度不到主峰(m/z 573.9)强度的1%。由此可以得出结论,新算法能较好地适应高丰度与低丰度信号强度相差超过1万倍的情况分析。

图4 NNATLD的三线性分解结果Fig.4 Results of NNATLD for the test data
3 结论
三线性分解策略与MRM 方法的不同之处:只需要采集一级质谱谱图并能做到采集全部离子信号,免除了在二级质谱确定定量和定性离子的麻烦,减少了二级质谱对离子信号强度的降低效应;由于自身能够按照物理/化学意义分离信号及其定量信息,所以并不要进行离子选择;并且三线性分解算法要求的只是目标分析物能在质谱仪上有自身的信号,并不要求各个仪器上面的谱图完全一样,因而基于三线性分解算法建立蛋白质定量策略是可以跨离子源/跨仪器使用的,该定量策略具有较好的通用性。
[1]Washburn M P,Wolters D,Yates III J R.Nature Biotechnology,2001,19(3):242
[2]Wolters D A,Washburn M P,Yates III J R.Anal Chem,2001,73(23):5683
[3]Fournier M L,Gilmore J M,Martin-Brown S A,et al.Chem Rev,2007,107(8):3654
[4]Nesvizhskii A I.Meth Mol Biol,2007,367:87
[5]Blonder J,Chan K C,Issaq H J,et al.Nature Protocols,2007,1(6):2784
[6]Liu H,Sadygov R G,Yates III J R.Anal Chem,2004,76(14):4193
[7]Kawamoto S,Matsumoto Y,Mizuno K,et al.Gene,1996,174(1):151
[8]Anderson L,Seilhamer J.Electrophoresis,1997,18(3/4):533
[9]Futcher B,Latter G I,Monardo P,et al.Mol Cell Biol,1999,19(11):7357
[10]Gygi S P,Rochon Y,Franza B R,et al.Mol Cell Biol,1999,19(3):1720
[11]Opiteck G J,Lewis K C,Jorgenson J W,et al.Anal Chem,1997,69(8):1518
[12]Wall D B,Kachman M T,Gong S,et al.Anal Chem,2000,72(6):1099
[13]Wu H L,Nie J F,Yu Y J,et al.Anal Chim Acta,2009,650(1):131
[14]Wu H L,Shibukawa M,Oguma K.J Chemom,1998,12(1):1
[15]Zhang Y,Wu H L,Xia A L,et al.Talanta,2007,72(3):926
[16]Li S F,Wu H L,Yu Y J,et al.Talanta,2010,81(3):805
[17]Yu Y J,Wu H L,Shao S Z,et al.Talanta,2011,85(3):1549
[18]Jaumot J,Gargallo R,de Juan A,et al.Chemom Intell Lab Syst,2005,76(1):101
[19]Jaumot J,Tauler R.Chemom Intell Lab Syst,2010,103(2):96
[20]Olivieri A C,Wu H L,Yu R Q.Chemom Intell Lab Syst,2009,96(2):246
[21]Booksh K S,Kowalski B R.Anal Chem,1994,66(15):782A
[22]Strohalm M,Kavan D,Novak P,et al.Anal Chem,2010,82(11):4648
[23]Strohalm M,Hassman M,Kosata B,et al.Rapid Commun Mass Spectrom,2008,22(6):905
[24]Carroll J,Chang J J.Psychometrika,1970,35(3):283
[25]Harshman R A.UCLA Working Papers in Phonetics,1970,16(1):84
[26]Kiers H,Krijnen W.Psychometrika,1991,56(1):147
[27]Chen Z P,Wu H L,Jiang J H,et al.Chemom Intell Lab Syst,2000,52(1):75
[28]Xia A,Wu H,Fang D,et al.J Chemom,2005,19(2):65
[29]Bro R,De Jong S.J Chemom,1997,11(5):3931
[30]Paatero P.Chemom Intell Lab Syst,1997,38(2):223
