巴西橡胶树HbNTL1基因启动子的克隆与序列分析
2013-07-12康桂娟黎瑜曾日中聂智毅
康桂娟 黎瑜 曾日中 聂智毅
(中国热带农业科学院橡胶研究所 农业部橡胶树生物学与遗传资源利用重点实验室和省部共建国家重点实验室培育基地—海南省热带作物栽培生理学重点实验室,儋州 571737)
植物特有的NAC(NAM,ATAF1/2和CUC2)转录因子组成了植物基因组中最大的转录因子家族之一[1]。膜结合NAC转录因子是植物NAC转录因子家族中一类C端具有跨膜结构域的转录调控因子,该类膜结合NAC蛋白依靠此结构锚着在细胞膜或内质网膜上[3-8]。到目前为止,已经在拟南芥(Arabidopsis thaliana)、大豆(Glycine maxL.)、玉米(Zea mays)、葡萄(Vitis viniferaL.)、水稻(Oryza sative)和毛果杨(Populus trichocarpa)等多种陆生植物中发现具有多个NTLs[1,4,6,8-11]。
NAC家族转录因子广泛参与调控植物体生长发育、生物和非生物胁迫应答以及激素信号转导过程[1,12-13]。越来越多的研究发现膜结合NAC转录因子的表达受非生物胁迫的诱导,在植物的生长发育和逆境胁迫应答中发挥重要作用[1,6,8]。Kim等[5,8]研究发现拟南芥NTLs基因受多种生物和非生物胁迫条件诱导,并且可以调节拟南芥的开花时间。研究结果显示,水稻OsNTL基因与拟南芥相似,都可以被多种非生物胁迫诱导表达[8]。Puranik等[1]研究发现谷子膜结合SiNAC基因可能与胁迫和生长发育调控相关,可以被渗透胁迫、盐胁迫、乙烯和茉莉酸甲酯诱导表达。本实验室已经从橡胶树中克隆了HbNTL1基因的全长cDNA序列,在此基础上,利用基因组步移(Genome Walker)方法克隆了该基因的启动子序列,并通过生物信息学软件分析其核心启动子结构、顺式作用元件及其生物学功能,旨在为进一步研究HbNTL1转录因子的表达调控以及其在橡胶树抵御逆境胁迫中的作用提供信息和依据。
1 材料与方法
1.1 材料
巴西橡胶树(Hevea brasiliensis)无性系热研7-33-97种植在中国热带农业科学院实验农场,幼叶液氮研磨后冻存在-80℃,用于基因组DNA的提取。
1.2 方法
1.2.1 巴西橡胶树基因组DNA的提取 橡胶树基因组DNA的提取采用改进的CTAB法[16]。
1.2.2HbNTL1基因启动子的克隆 启动子克隆采用基因组步移的方法,Genome Walker文库由本研究组夏可灿等按Universal Genome WalkerTMKit(Clontech)使用说明书构建[17]。根据HbNTL1转录因子的cDNA序列,设计一对巢式PCR特异性引物:5'-CTCTGTGGCCCGGTTTGTCTTGGAACCA-3'(NTLGSP1)和5'-GTGAAGCCGCAGAATCACCACGTGCCA-3'(NTLGSP2)。上游引物为Universal Genome WalkerTMKit自带的接头引物AP1和AP2,以PvuII,EcoRV,StuI和DraI酶切连接的产物为模板,反应程序为:94℃ 25 s,72℃ 3 min,7个循环;94℃ 25 s,67℃3 min,32个循环。
1.2.3 生物信息学分析 利用Softberry(http://linu x1.softberry.com/berry.phtml)网站的TSSP(植物启动子预测)数据库分析了所克隆得到的可能为巴西橡胶树HbNTL1转录因子基因启动子的序列;利用Berkeley Drosophila Genome Project(BDGP)的在线软件Neural Network Promoter Prediction Promoter(http://www.fruitfly.org/seq_tools/promoter.html)预测核心启动子区域和转录起始位点;利用在线软件PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和搜索植物顺式作用元件数据库PLACE(http://www.dna.affrc.go.jp/PLACE/)对启动子序列中可能存在的顺式作用元件进行分析[18]。
2 结果
2.1 橡胶树HbNTL1基因启动子的克隆
根据橡胶树HbNTL1基因的cDNA序列设计一对巢式PCR特异性引物NTLGSP1和NTLGSP2,进行PCR扩增,两轮PCR扩增后得到约2.0 kb的条带(图1),将PCR产物连接pMD18-T vector,转化大肠杆菌DH5α,筛选阳性克隆进行测序。将测序结果与已知序列比对分析,去除载体、接头引物和与读码框重叠序列,得到橡胶树HbNTL1基因上游非编码区1 718 bp的序列。
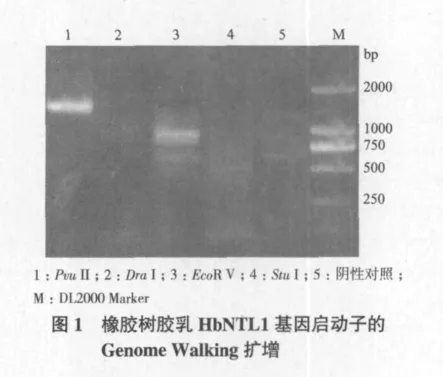
2.2 HbNTL1基因启动子的生物信息学分析
在线软件TSSP预测结果表明,HbNTL1基因5'非编码区序列存在一个可能的启动子区,位置在1 513 bp处,一个对应的TATA框,位置在1 484 bp处,一个增强子CAAT-box,在1 436 bp处(图2)。BDGP在线软件Neural Network Promoter Prediction Promoter预测HbNTL1基因的核心启动子序列和转录起始位点,结果(图2)显示,HbNTL1基因5'非编码区序列中含有真核生物典型的核心启动子区域(1 473-1 522 bp),其中+1位置的碱基A是转录起始位点,位于翻译起始位点(ATG)上游206 bp处。HbNTL1基因的转录起始位点碱基为A,TATA-box在转录起始位点的-30 bp处,符合高等植物启动子的基本结构特征。
利用在线软件PlantCARE和PLACE对HbNTL1基因启动子序列中可能含有的顺式作用元件进行分析,结果发现,除具有12个拷贝的TATA-box和CAAT-box外,还具有9个赤霉素、脱落酸和茉莉酸等激素响应元件(ABRE、GAREAT、 P-box、TATC-box和TGACG-motif)、56个逆境胁迫响应元件(ACGTATERD1、ARE、CCAATBOX1、CURECORECR、DOFCOREZM、HSE、MYBCORE、TC-rich repeats、W-box)、6个光顺式反应元件(GAG-motif、GATA-box和G-box)、28个组织特异性表达元件(CACTFTPPCA1、GTGANTG10、POLLEN1LELAT52、ROOTMOTIFTAPOX1和RY-repeat)以及2个拷贝的昼夜节律控制元件(Circadian)等(表1)。

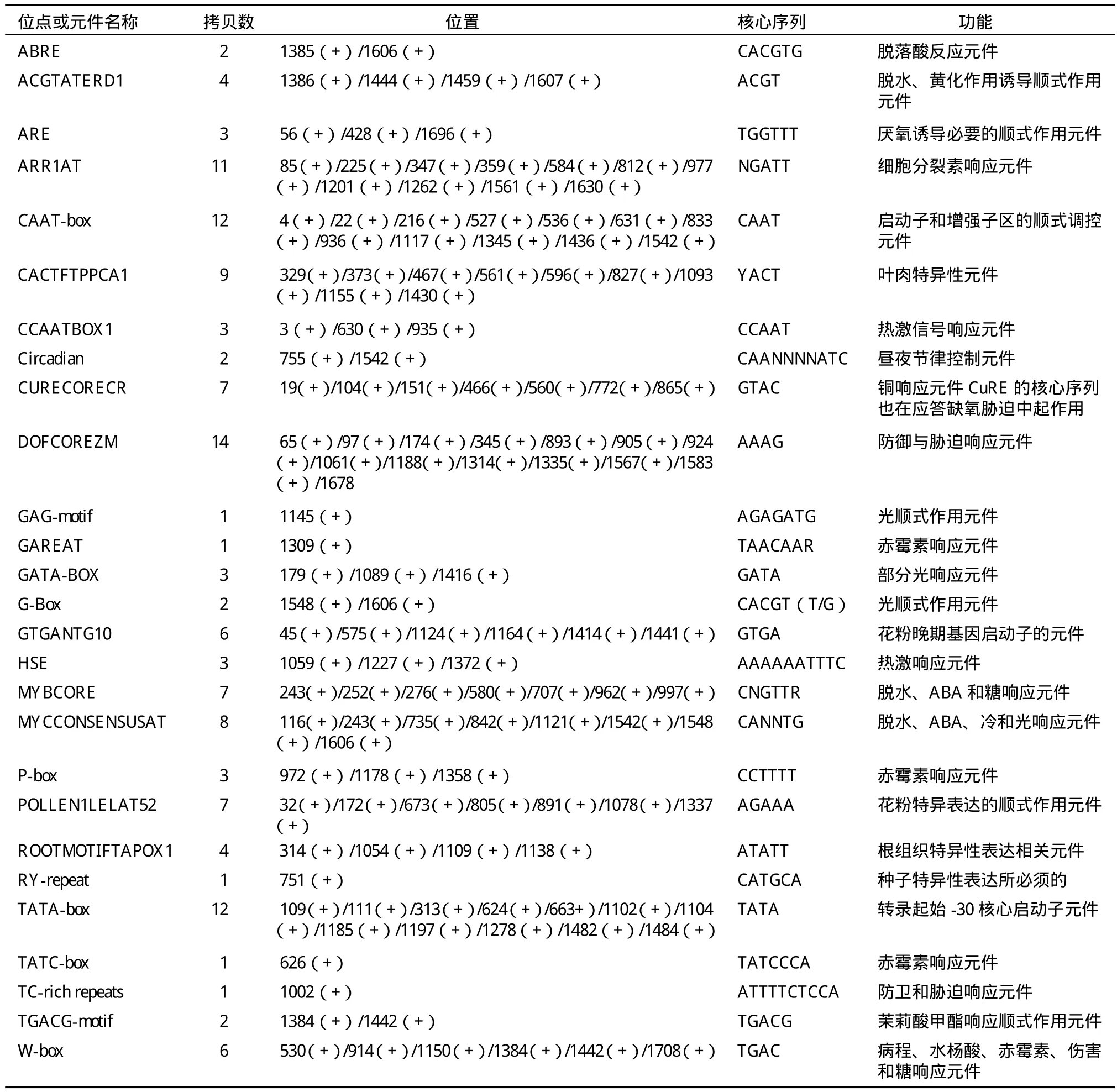
表1 HbNTL1启动子区域主要顺式作用元件分析
3 讨论
NAC转录因子是植物特有的一类转录调控因子,在植物的生长发育、器官建成、激素调节以及生物和非生物胁迫应答等方面具有重要作用[1,2]。但是对于NAC类转录因子的研究还主要集中在基因的克隆与转基因表达分析上,而对他们的上游调控因子和下游靶标基因都不是很清楚。本研究在已知巴西橡胶树膜结合NAC转录因子HbNTL1基因cDNA序列的基础上,利用Genome Walker的方法获得了其5'调控序列1 718 bp,通过在线软件的预测与分析该序列具有真核生物典型的核心启动子区域和大量的顺式作用元件。HbNTL1基因启动子序列中存在多个激素响应元件、光作用顺式元件和组织特异性表达元件,因此,HbNTL1的表达可能受到光信号和激素的调控,并且存在组织特异性。
HbNTL1基因启动子序列中含有大量的逆境胁迫响应元件,占到了总数的50%左右。MYBCORE和MYCCONSENSUSAT元件在HbNTL1基因启动子序列中共发现有15个拷贝,这些元件分别是MYB和MYC转录因子的识别位点,与其相应转录因子在ABA信号通路和非生物胁迫应答中具有重要作用[19,20]。ABRE元件是脱落酸响应元件,在干旱、脱水、盐和低温等非生物胁迫时诱导植物体内积累脱落酸,从而调控相关基因的表达[21]。启动子分析发现HbNTL1基因启动子序列中包含了14个拷贝的DOFCOREZM元件,该元件是Dof 转录因子的识别位点,调控保卫细胞特异基因的表达,在植物防御过程中具有重要作用[22]。在HbNTL1基因启动子中还发现有14个拷贝的W-box元件,该顺式作用元件是WRKY转录因子的识别位点。植物特有的WRKY基因通过与顺式作用元件W-box特异结合调控下游目标基因表达,与植物的防御反应、逆境应答、衰老信号和生长发育等密切相关[23,24]。ABRE、DOFCOREZM、MYBCORE、MYCCONSENSUSATHSE和W-box等反应元件在其他植物胁迫逆境相关NAC转录因子基因的启动子区域都有报导[1,2,10,25-28]。HbNTL1基因启动子序列中这些反应元件的大量存在表明,HbNTL1可能是一个逆境胁迫相关NAC转录因子(stress-responsive NAC),在橡胶树抵抗逆境胁迫的生理过程中具有重要功能。本实验室将利用巴西橡胶树HbNTL1基因的5'调控序列,通过酵母单杂交系统筛选调控HbNTL1转录因子的上游调控因子,从而进一步研究HbNTL1的表达调控及其在橡胶树逆境胁迫中的作用。
4 结论
本研究利用Genome Walker 的方法克隆了巴西橡胶树膜结合NAC转录因子HbNTL15'调控序列1 718 bp,软件预测与分析该序列具有真核生物典型的核心启动子区域和大量的顺式作用元件,其中逆境胁迫相关元件占到50%,HbNTL1可能是一个逆境胁迫相关NAC转录因子,在橡胶树抵抗逆境胁迫的生理过程中具有重要功能。
[1] Puranik S, Sahu PP, Srivastava PS, et al. NAC proteins:regulation and role in stress tolerance[J] . Trends Plant Sci, 2012, 176(6):369-381.
[2] Nakashima K, Takasaki H, Mizoi J, et al. NAC transcription factors in plant abiotic stress responses[J] . BBA-Gene Regul Mech,2012, 1819(2):97-103.
[3] Nuruzzaman M, Manimekalai R, Sharoni AM, et al. Genome-wide analysis of NAC transcription factor family in rice[J] . Gene, 2010,465(1-2):30-44.
[4] Le DT, Nishiyama R, Watanabe Y, et al. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J] .DNA Res, 2011, 18(4):263-276.
[5] Kim SG, Park CM. Membrane-mediated salt stress signaling in flowering time control[J] . Plant Signal Behav, 2007, 2(6):517-518.
[6] Seo PJ, Kim SG, Park CM. Membrane-bound transcription factors in plants[J] . Trends Plant Sci, 2008, 13(10):550-556.
[7] Tran LSP, Nishiyama R, Yamaguchi-Shinozaki K, et al. Potential utilization of NAC transcription factors to enhance abiotic stress tolerance in plants by biotechnological approach[J] . GM Crops,2010, 1(1):32-39.
[8] Kim SG, Lee SM, Seo PJ, et al. Genome-scale screening and molecular characterization of membrane-bound transcription factors inArabidopsisand rice[J] . Genomics, 2010, 95(1):56-65.
[9] Shen H, Yin YB, Chen F, et al. A bioinformatic analysis of NAC genes for plant cell wall development in relation to lignocellulosic bioenergy production[J] . Bioenerg Res, 2009, 2:217-232.
[10] Puranik S, Bahadur RP, Srivastava PS, et al. Molecular cloning and characterization of a membrane associated NAC family gene,SiNAC from foxtail millet[Setaria italica(L.)P. Beauv.] [J] .Mol Biotechnol, 2011, 49:138-150.
[11] Tang YM, Liu MY, Gao SQ, et al. Molecular characterization of novel TaNAC genes in wheat and overexpression of TaNAC2a confers drought tolerance in tobacco[J] . Physiol Plant, 2012,144(3):210-224.
[12] Hao YJ, Wei W, Song QX, et al. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J] .Plant J, 2011, 68(2):302-313.
[13] Distelfeld A, Pearce SP, Avni R, et al. Divergent functions of orthologous NAC transcription factors in wheat and rice[J] . Plant Mol Biol, 2012, 78:515-524.
[14] Yang SD, Seo PJ, Yoon HK, et al. TheArabidopsisNAC transcription factor VNI2 integrates abscisic acid signals into leaf senescence via the COR/RD genes[J] . Plant Cell, 2011, 23(6):2155-2168.
[15] Zhong RQ, Lee CH, Ye ZH. Global analysis of direct targets of secondary wall NAC master switches inArabidopsis[J] . Mol Plant, 2010, 3(6):1087-1103.
[16] An ZW, Wang QT, Hu YS, et al. Co-extraction of high-quality RNA and DNA from rubber tree(Hevea brasiliensis)[J] . Afr J Biotechnol, 2012, 11:9308-9314.
[17] 夏可灿, 康桂娟, 黎瑜, 等.橡胶树胶乳HbPLDα1基因机器启动子的克隆与生物信息学分析[J] .热带作物学报, 2012, 1(33):63-69.
[18] Higo K, Ugawa Y, Iwamoto M, et al. Plant cis-acting regulatory DNA elements(PLACE)database:1999 Nucleic[J] . Acids Res, 1999, 27(1):297-300.
[19] Yamaguchi-Shinozaki K, Shinozaki K. Organization ofcisacting regulatory elements in osmotic-and cold-stress-responsive promoters[J] . Trends Plant Sci, 2005, 10(2):88-94.
[20] Abe H, Yamaguchi-Shinozaki K, Urao T, et al. Role ofArabidopsisMYC and MYB homologs in drought-and abscisic acid-regulated gene expression[J] . The Plant Cell Online, 1997, 9(10):1859-1868.
[21] Narusaka Y, Nakashima K, Shinwari ZK, et al. Interaction between twocis-acting elements, ABRE and DRE, in ABA-dependent expression ofArabidopsisrd29A gene in response to dehydration and high-salinity stresses[J] . Plant J, 2003, 34(2):137-148.
[22] Plesch G, Ehrhardt T, Mueller-Roeber B. Involvement of TAAAG elements suggests a role for Dof transcription factors in guard cellspecific gene expression[J] . Plant J, 2001, 28(4):455-464.
[23] Eulgem T, Rushton PJ, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J] . Trends Plant Sci, 2000, 5(5):199-205.
[24] Rushton PJ, Somssich IE, Ringler P, et al. WRKY transcription factors[J] . Trends Plant Sci, 2010, 15(5):247-258.
[25] Han QQ, Zhang JH, Li HX, et al. Identification and expression pattern of one stress-responsive NAC gene fromSolanum lycopersicum[J].Mol Biol Rep, 2011, 39:1713-1720.
[26] Nakashima K, Tran LSP, Van Nguyen D, et al. Functional analysis of a NAC-type transcription factor OsNAC6 involved in abiotic and biotic stress-responsive gene expression in rice[J] . Plant J, 2007,51(4):617-630.
[27] Fang YJ, You J, Xie KB, et al. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice[J] . Mol Genet Genomics, 2008,280:547-563.
[28] Zheng XN, Chen B, Lu GJ, et al. Overexpression of a NAC transcription factor enhances rice drought and salt tolerance[J] . Biochem Biophys Res Commun, 2009, 379(4):985-989.
