红麻质核互作型雄性不育相关基因atp1的克隆与分析
2013-05-23陈艳翠黄思齐李建军唐慧娟陈安国李德芳
陈艳翠,黄思齐,李建军,唐慧娟,陈安国,李德芳
(中国农业科学院麻类研究所,湖南长沙,410205)
引言
红麻(Hibiscus cannabinus L.)是锦葵科木槿属一年生速生纤维作物,适应性广,除了传统的用途之外,现在主要对其进行多功能用途开发,例如用来制作纸浆、墙布、麻地膜、饲料等,被认为是21世纪最具发展潜力的多用途作物[1]。
利用杂交优势是提高红麻产量的有效方法。自从2003~2004年中国农科院麻类所红麻育种课题在海南三亚南繁基地发现了质核互作型红麻雄性不育株后,有效的避免了在杂交优势利用中化学杀雄以及人工去雄的劣势[2]。随着现代分子生物学技术的发展,了解红麻雄性不育的机理对于利用杂交优势进行红麻杂交制种具有重要意义。
分离、鉴定和克隆与雄性不育相关的基因以及对其某些功能性区域进行分析,是深入研究雄性不育的必然途径[3]。在已经鉴定的各种植物的雄性不育基因中,许多雄性不育基因涉及了ATP合成酶基因[4]。ATP合酶是生物体能量代谢的关键酶,参与氧化磷酸化与光合磷酸化反应,在跨膜质子动力势的推动下催化合成生物体的能量ATP[5]。李友勇的研究指出在BNS温敏性小麦雄性不育转化系中发现,ATP合酶α和β亚基、胞质苹果酸脱氢酶、线粒体醛脱氢酶亚基、Rubisco亚基等呼吸、光合能量代谢蛋白表达丰富,但在不育系中这些蛋白缺失或下调[6]。Mineo Senda等对油菜ATP合酶α亚基基因的基因组结构的分析发现该基因的编码区是一样的,而在起始密码子之前和终止密码子之后存在差异[7]。除此之外,C.D.chase以及Per Bergman等的研究分别在菜豆和烟草中发现在atp1基因编码序列附近有存在一个与atp1基因共转录的开放阅读框,使得基因的表达在不育和可育之间产生差异[8-10]。这些现象都表明ATP合成酶基因可能在植物不育中起到了一定的作用。
目前,红麻的ATP合成酶基因并未克隆,克隆并研究红麻中的ATP合成酶基因,可以在一定程度上揭示红麻雄性不育的机理,同时为红麻杂种优势利用打下基础[11-13]。
1 材料与方法
1.1 材料
红麻不育系品种261N5-19A对应的保持系261N5-19B,由中国农业科学院麻类研究所红麻育种课题选育而成。
RNA提取试剂盒购自Omega公司;DNA marker购自天根公司;rTaq DNA聚合酶、KOD-neoplus购自ToYoBo公司;琼脂糖(Biowest公司);5×TBE缓冲液;10 mg/mL EB;切胶回收试剂盒购自AxyGen公司;5’RACE System for Rapid Amplification of cDNA Ends试剂盒购自Invitrogen公司;SMARTerTMRACE cDNA Amplification Kit试剂盒购自Clontech公司;引物合成及序列测序均由上海生工公司完成。
1.2 红麻幼苗RNA的提取
将红麻保持系种子于28℃、光照0%下培养4~6天待用。
RNA提取所用的枪头、Eppendorf管浸泡过夜,高温高压灭菌,烘干待用;其他玻璃器皿、研钵等按照分子克隆介绍的方法处理。RNA提取具体方法参照Omega公司的RNA提取试剂盒。RNA经分光光度计测定浓度和纯度,并跑电泳检测完整性。
1.3 atp1基因中间片段的扩增
将不同物种中的atp1基因的CDS区序列经DNAssist进行序列比对之后,取比较保守的序列进行引物设计,设计的引物序列如下:上游atp F:5’-GGTCGAGTGGTCTCAGTTGGAG;
下游atp R:5’-TCTAGTGGCATTCGATCACAGAA。以红麻保持系RNA反转录得到的第一链cDNA为模板,采用50μl PCR 反应体系,首先94℃预变性3min,然后以94℃ 30s、51℃ 30s、72℃1min为一个循环重复30次,最后72℃延伸6min。PCR扩增反应同时设置阴性对照,以蒸馏水为模板。
反应完毕后,用1%琼脂糖凝胶电泳检测反应产物,并切胶回收。将回收片段与pGEM-T easy进行连接并转化到感受态大肠杆菌中,摇菌涂平板。检测菌落,得到的阳性克隆送公司测序。
1.4 RACE进行atp1基因5’端和3’端的扩增
根据得到的中间片段序列分别设计5’和3’端的特异引物,分别如下:

5'R638-1:CAAAGACAACAATCCCT 3'363-1:CGAAGTGGCCGCCCTTGCTCAATTTG 5'R638-2:AACGCTATTCCTTTCACACCG 3'363-2:GTGCAAGGCTTACAGAAGTACCGAAAC 5'R638-3:TTCCCCAGCTTGAATCTCGTT 3'363-3:CTGTCAATGGATTCTGTGATCGAATGCC
以红麻保持系RNA为模板,分别按照5’RACE System for Rapid Amplification of cDNA Ends试剂盒以及SMARTerTM RACE cDNA Amplification Kit试剂盒完成atp1基因5’端和3’端序列的扩增。
反应完毕后,用1%琼脂糖凝胶电泳检测反应产物,并分别切胶回收。将回收片段分别与pGEM-T easy进行连接并转化到感受态大肠杆菌中,摇菌涂平板。检测菌落,得到的阳性克隆分别送公司测序。
1.5 atp1基因全长的拼接
将得到的atp1基因的中间片段序列以及5’、3’端序列进行拼接,得到atp1基因的全长。根据拼接的序列预测可能的CDS区。
1.6 atp1基因CDS区全长的扩增
根据预测的CDS区的起始密码子以及终止密码子附近设计引物,引物序列如下:QC638F:GGAACTCTTTATGGAATTCTCTC;QC638R:CTTCATCACATCATAGGTAAGGC。然后以红麻保持系幼苗的RNA反转录得到的第一链cDNA为模板,采用50μl的PCR反应体系,首先94℃预变性3min,然后以94℃ 30s、52℃ 30s、72℃ 75s为一个循环重复30次,最后72℃延伸6min。PCR扩增反应同时设置阴性对照,以蒸馏水为模板。
反应完毕后,用1%琼脂糖凝胶电泳检测反应产物,并切胶回收。将回收片段与pGEM-T easy进行连接并转化到感受态大肠杆菌中,摇菌涂平板。检测菌落,得到的阳性克隆送公司测序。
1.7 生物信息学分析
(1)与atp1基因CDS区同源的序列通过GenBank的BLAST进行;
(2)同源序列之间的比对通过DNAssist软件完成;
(3)进化树的构建:首先通过Clustal X(版本1.8)完成多序列的比对,然后由MEGA4软件构建进化树。
2 结果与分析
2.1 红麻幼苗RNA的提取
提取的RNA首先在分光光度计下检测,D260nm/D280nm值在1.8左右,表明其纯度较高。然后经1%琼脂糖凝胶电泳检测,EB染色,于紫外灯下观察并拍照记录,如图1所示。
图1 红麻幼苗RNAFig.1 RNA of kenaf(Hibiscus cannabinus)seedling
由图1.可以看出,提取的RNA有很明显的18S和28S两条清晰的条带,且两条条带 (28 sRNA:18 sRNA)的亮度比约为2:1,因此可以用来进行后续实验使用。(图中所用marker为200bp ladder)
2.2 中间片段的扩增
通过对不同物种中atp1基因序列的比对分析之后,发现该基因的保守性很高,所以直接根据保守部分的序列设计一对引物(atp F,atp R),扩增得到的产物电泳如图2.所示,该片段经切胶回收、连接、转化、测序以及去载体之后,实际大小为1295bp。
2.3 atp1基因5’端和3’端序列的扩增
由得到的atp1基因中间片段序列设计5’端扩增的R638-1、R638-2和R638-3以及3’端扩增的363-1、363-2和363-3特异引物,PCR扩增产物电泳检测如图3、图4所示,片段经切胶回收、连接、转化、测序以及去载体后,实际大小5’端为374bp,3’端为897bp。
图2 扩增得到的中间片段 图3.扩增得到的5’端序列 图4.扩增得到的3’端序列Fig.2 Intermediate fragments Fig.3 5’end sequences Fig.4 3’end sequences
2.4 atp1基因全长拼接以及CDS区预测
将实验所得的中间片段以及5’3’序列经DNAssist软件比对,通过重叠区域拼接,得到atp1基因的全长,共有2315bp。预测的CDS区全长为1524bp,在5’端216位存在起始密码子ATG;3’端的第1737位存在终止密码子TGA;3’末端存在poly(A)尾巴。
2.5 全长CDS区序列的扩增及测序
根据预测的CDS区设计引物(QC638F,QC638R),扩增红麻保持系中atp1基因的CDS区序列,1%琼脂糖凝胶电泳检测后切胶回收,与载体连接并转化进感受态大肠杆菌中,检测的阳性克隆送公司测序。实际得到的序列与拼接预测的ORF一致,大小为1524bp,编码508个氨基酸残基,蛋白质分子质量约为55.1KD,等电点为6.23。


2.6 基因序列分析及进化树构建
为研究不同物种中atp1基因的同源性以及进化关系,从GenBank下载了胡萝卜等物种中的atp1基因序列进行同源性分析以及构建系统进化树。结果显示,不同物种中的atp1基因的序列同源性为94%~98%。这表明在不同物种中atp1基因的序列比较保守。通过构建系统进化树(图5)发现,与红麻atp1基因进化关系较近的是大豆。
3 讨论
本研究以红麻质核互作型雄性不育保持系为材料,利用同源克隆与RACE结合的方法扩增得到质核互作型红麻保持系中的atp1基因。首次在红麻中克隆得到ATP合酶α亚基的编码基因atp1基因。
将红麻保持系atp1基因的CDS区序列与其他物种中的同源序列进行比对分析,可以看出:红麻atp1基因与其他物种中的同一基因同源性较高,但是在基因的5’和3’端序列差异较明显,这可能与物种之间的特异性有关。得到保持系中的atp1基因序列之后,为下一步克隆红麻雄性不育系
atp1 , 、红麻植物雄性不育的调控机理。
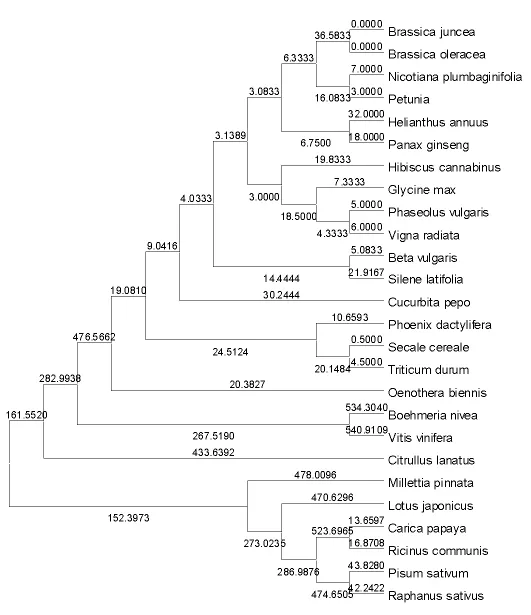
图5 不同物种中atp1基因的系统进化树Fig.5 Phylogenetic tree of gene atp1of 26 species
[1]李德芳,陈安国,唐慧娟.红麻质核互作型雄性不育系的发现及初步创制[J].中国麻业科学,2007,29(2):78.
[2]李辉,李德芳,陈安国,等.红麻雄性不育系的选育和不育基因的ISSR分子标记[J].农业生物技术科学,2008,24(8):80-83.
[3]陈烨丽,薄天岳,陈学好,等.球茎甘蓝细胞质不育相关基因orf138的克隆[J].园艺学报,2009,36(增刊):1966.
[4]石晓冰,沈允钢.ATP合酶:自然界最小的旋转发动机[J].生命的化学,1999,19(5):225-229.
[5]王军卫,王小利,张改生,等.粘类小麦细胞质雄性不育基因的初步研究[J].西北植物学报,2009,29(5):0900-0904.
[6]李友勇,茹振刚,苏晴,等.小麦BNS雄性不育系及其转换系花药差异蛋白鉴定与分析[J].作物学报,2011,37(9):1540-1550.
[7]Mineo Senda,Tetsuo Mikami,Toshiro Kinoshita.The sugar beet mitochondrial gene for the ATPase alpha-subunit:sequence,transcription and rearrangements in cytoplasmic male-sterile plants[J].Current Genetics,1993,24:164-170.
[8]C.D.Chase and V.M.Ortega.Organization of ATPA coding and 3'-flanking sequence associated with cytoplasmic male sterility in Phaseolus vulgaris L[J].Current Genetics,1992,22:147-153.
[9]Rainer Hans Köhler,Renate Horn,Andreas Lössl,et al.Cytoplasmic male sterility in sunflower is corelated with the co-transcription of a new open reading frame with the atpA gene[J].Molecular and General Genetics,1991,227:369-376.
[10]Per Bergman,Johan Edqvist,Isabelle Farbos,et al.Male-sterile tobacco display abnormal mitochondrial atp1 transcript accumulation and reduced floral ATP/ADP ratio[J].Plant Molecular Biology,2000,42:531-544.
[11]陈鹏,周瑞阳.红麻线粒体中细胞质雄性不育候选基因cox3的克隆与分析[J].生物技术通讯,2011,22(1):37-40.
[12]赵扬.红麻细胞质雄性不育相关基因nad的克隆与再生体系初探[D].广西大学,2011.
[13]陈鹏,周瑞阳,赵扬,等.红麻不育系和保持系中cob基因的克隆与分析[J].生物技术通报,2011,2:93-97.
