精神分裂症相关基因的预测——基于大规模数据的整合分析
2013-04-08史艺宾田卫东
梁 燕,史艺宾,田卫东
(复旦大学 生命科学学院 生物统计研究所,上海 200433)
精神分裂症是最严重的精神疾病之一,以思维过程障碍以及情绪反应迟钝为特征.患者的精神活动与环境不协调,最常见的表现为幻听、妄想、胡言乱语等,从而造成个性、思维、行为以及情感等方面的分裂[1].精神分裂症是第七大治疗成本最高的疾病,影响着全世界0.5%~1%的人口[2].精神分裂症的年万人发病率为2~4人,终身发病率高达1%[1],先天遗传率高达80%~85%[3].由于精神分裂症疾病的复杂性,对它的研究一直是生物学和医学的热点,寻找精神分裂症相关的基因,对开发潜在的药物靶标及治疗该疾病具有重大意义.在基于遗传家系的连锁分析方法(Linkage Analysis)、基于高通量基因芯片的病例对照全基因组关联分析(GWAS)甚至全基因组测序等新方法的广泛应用下,像精神分裂症这类复杂疾病致病基因的鉴定已取得了很大进展,但其表型往往多样而且鲜有明显遗传家系,限制了连锁分析的应用,通过GWAS获得的疾病易感基因也不能解释其全部的表型[4],因此致病基因鉴定以及分子机制研究仍然还是遗传学领域的难题.
由于传统方法的限制,利用生物信息学模型预测候选基因成为寻找精神分裂症相关基因的另一思路.近几年来,针对精神分裂症的候选基因预测和筛选的工作正逐渐进入视野,Sun等使用综合优势比的方法[5]对SZGene网站中已报道的和精神分裂有潜在相关性的1008个候选基因进行了排序;随着基因组数据库的完善及精神分裂症基因组关联研究的增多,2009年Sun等通过整合4个数据库调整排序比重筛选出同精神分裂症最为相关的优先基因[6].这些预测复杂疾病相关基因的生物信息模型除在精神分裂症得到运用以外,也被用于其他复杂疾病上,如自闭症(Autism)[7]和抑郁症(Depression)[8]等精神类复杂疾病;2003年Frances S Turner等人也运用基因功能注释关联的生物信息学算法[9],对29种人类孟德尔遗传数据库(OMIM,Online Mendalian Inheritance in Man)收录的疾病进行了疾病基因预测.这些预测得到的疾病基因于未来研究该疾病的致病机理、生化路径和药物靶点等有很大的指导作用.也为后来进行复杂疾病基因预测提供了生物信息学模型的借鉴.
已知同一疾病表型的致病基因往往在功能水平上具有较紧密的联系,如都参与同一条生物通路、之间存在蛋白相互作用或遗传相互作用等.如果能在基因组中寻找到与已知致病基因有功能关联的基因,那就可缩小需要验证的候选相关基因的数目.高通量技术手段的快速发展已产生了包括基因组、表观遗传组、转录组及蛋白质组等各个水平上的大规模组学数据,这使得利用生物信息学方法对这些数据进行整合、分析和建模后,从基因组层次上研究基因功能和基因相互作用关系成为了可能.在之前的研究工作中,我们已开发了一套整合组学数据预测基因功能的生物信息学方法,并成功应用于酵母[10]和小鼠[11]基因的功能预测.在该模型中随机森林模型具有最优秀的预测表现,在此基础上,我们可通过整合最新的组学数据来预测与已知精神分裂症相关基因有功能关联的候选基因(图1).
近几年的全基因组关联研究发现,精神分裂症不仅同常见等位基因的SNP位点[12]及罕见等位基因变异相关[13],迄今为止发表的4项拷贝数全基因组关联研究发现精神分裂症同一些基因拷贝数异常(CNV,Copy Number Variation)也有很大关系[12,14-17].虽然已有多项精神分裂症GWAS研究,但找到的显著性SNP位点和基因却不多.截至目前只找到两个基因间区位点以及一个未知功能的基因ZNF804A(22q11.2)拷贝数缺失[15,18].令人感到沮丧的是,也有一些研究宣告和反驳曾经被深入研究的精神分裂症同基因组相关区域的关联[19,20];同时,对于已被学界关注的精神分裂症易感基因或易感基因组区域,它们的基因组结构、功能调控关联以及进化模式都十分复杂,缺乏进一步探究的规律性.这些都是大规模的、且成本高昂的全基因组关联研究在疾病基因预测方面所遇到的障碍和难题.
基于上述功能关联基因预测以及全基因组关联研究的优缺点,本文试图利用目前不断公开的大规模数据,架设连接功能预测和全基因组关联研究的桥梁,优选出精神分裂症相关基因,为精神分裂症基因探究的领域提供新思路.
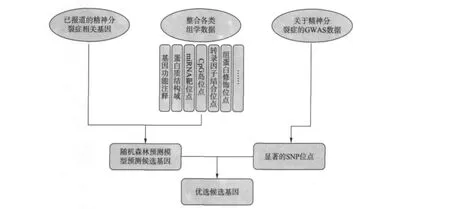
图1 利用大规模数据预测精神分裂症相关基因的流程图Fig.1 Framework for schizophrenia related gene prediction
1 方 法
1.1 精神分裂症相关基因的搜集
对于复杂多基因疾病的相关基因,目前没有如OMIM[21]的直接数据来源,但文献搜索可以较为全面的搜集到复杂疾病的相关基因,且这一方法已被学界认可并得到广泛应用[22].我们使用Genecards[23]、SZgene[24]、SZGR[25]这3个数据平台结合文献搜索搜集精神分裂症相关基因作为训练集.首先在SZgene数据库中搜索到在文献中提及的同精神分裂症相关的基因1008个,通过genecards数据库从中筛选出334个基因在疾病注释上与精神分裂症相关,然后结合SZGR数据库的基因信息,最终得到205个“同精神分裂症相关”为表型的基因(见第268页附表1).
附表1 精神分裂症相关基因列表SupplementTab.1 Schizophrenia related gene list
1.2 整合大规模组学数据
本文生物信息学预测模型中运用的大规模组学数据详情如表1.主要包括了基因功能注释、同源蛋白数据、蛋白质结构域、miRNA、药物靶点、重复序列以及表观遗传学上的CpG岛、转录因子、组蛋白甲基化和乙酰化修饰、转录因子、组蛋白H2AZ修饰、DNA聚合酶I敏感位点等方面的数据类型.对于所有下载的数据,所有基因以对应到Ensembl数据库中的基因编号为准,所有属性依次编号,最终整合成一个基因对应一个属性的二元数据格式,共21147个基因及75752个属性,应用于随机森林模型预测候选基因.
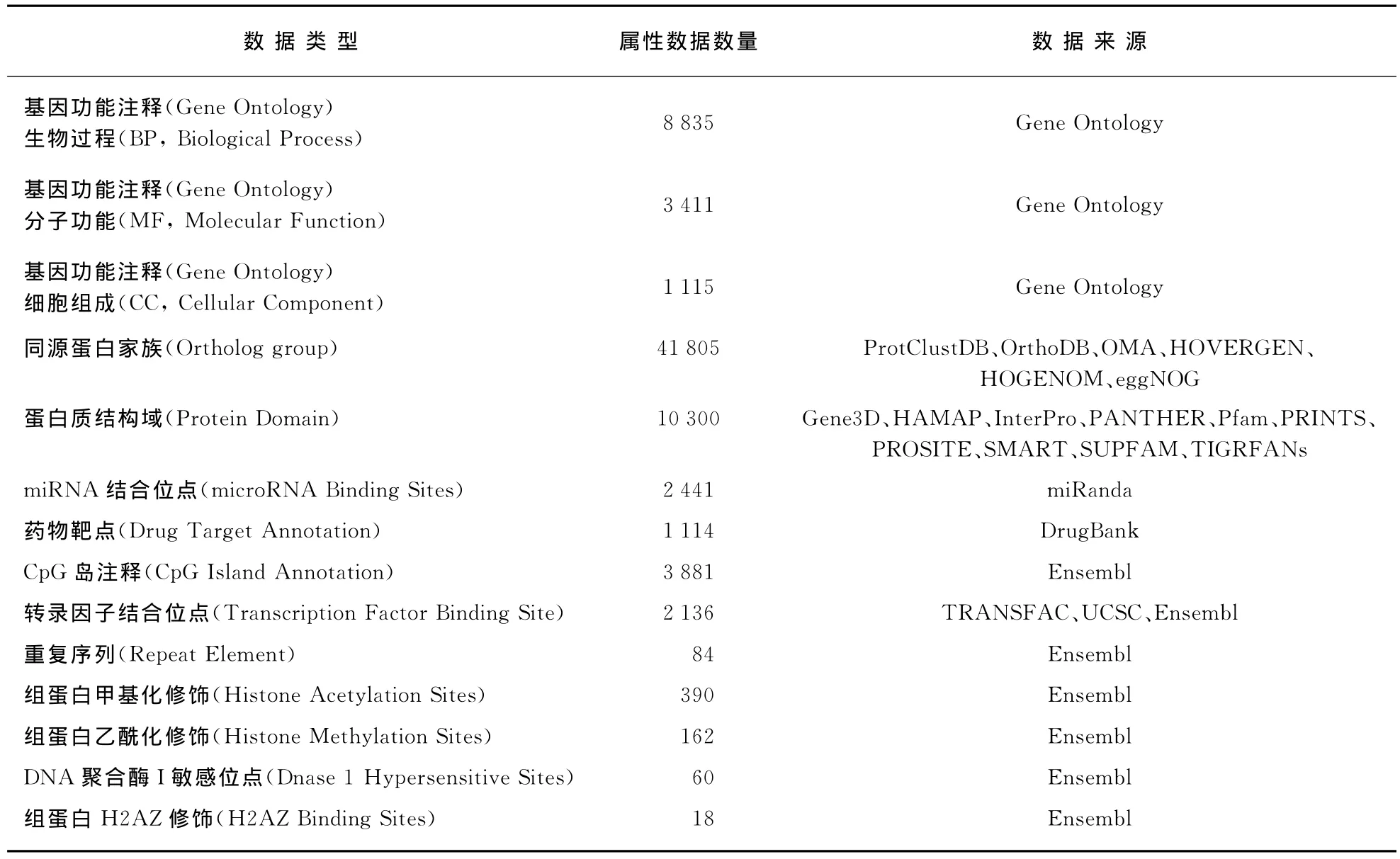
表1 随机森林预测模型中所用的数据类型Tab.1 Multiple datasets utilized in the random forests model to predict schizophrenia related genes
1.3 生物信息学预测模型
本预测模型是以随机森林(Random Forests)为机器学习算法进行精神分裂症基因的预测[26],数据是如上所述的基于每个基因在不同数据库中被标注的功能特征或者基因组特征.我们将“同精神分裂症相关”作为一种基因功能属性来做预测,因而共得75753个属性.随机森林分类器是一个包含多个决策树(Decision Tree)的分类器,其树根节点(Root Node)是通过Bootstrap取样(非放回取样法,Sampling Without Replacement)方法得到的训练基因数据(Training Gene Data),在每一节点,一部分对象特征被用于分离对象数据,分割的原理是通过某一个特征的预测变量递归方式(Recursively Splitting)分离对象,这个用于分离对象的变量依照基尼不纯度测定(Gini Impurity Measure)而确定,该方法被用于遵循给定变量来测试对象的一致性程度[27].对于给出最大Gini Impurity减少量的预测变量值,我们按照以下方法计算相对应其的超几何分布概率(Hypergeometric Distribution Probability):
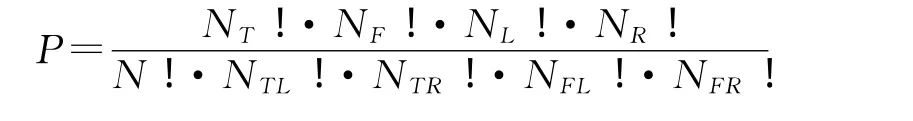
其中N表示母节点(Parent Node)的基因个数,NR和NF分别表示被标注和没有被标注有某一个基因功能(如某一个GO或自定义的“同精神分裂症相关”功能)的基因个数,NTL,NTR,NFL和NFR是相对应的分离后左右子节点(Daughter Node)的基因数.如果P>α(除特殊说明外,默认情况下取α=0.01),则该分离被弃去,并将这个节点作为叶节点(Leaf Node).最后某一个给定基因关于一个给定基因注释(GO)的分数既为将这个基因归类为阳性(Positive)的决策树的比例.关于随机森林算法的详细内容可参考文献[26].
由于用于预测变量的大规模数据(75753个特征数据)会影响到随机森林方法的效率,我们用“特征筛选”(Feature Selection)的方法移除不提供信息的变量(Marginally Uninformative Variables).最终得到的某一个给定基因关于一个给定基因注释(GO)的RF值为:在所有GO注释的决策树中,该基因位于叶节点的平均概率.对于随机森林分类器这一模型来说,有3个参数,它们分别是:每个分离点的随机变量值(the Number of Random Variables at Each Split),特征选择的临界值(the Threshold for Feature Selection)和提前终止的临界值(the Threshold for Early Stopping).为了优化这些参数,我们也采用了交叉验证(Cross Validation)的方法.在本次预测精神分裂症相关基因的模型中,我们选择了200棵树,α值默认为0.01.关于该模型的具体介绍请参见文献[10]中的Random Forest部分.预测结果的查准率(Precision)和召回率(Recall,又称查全率)定义为:
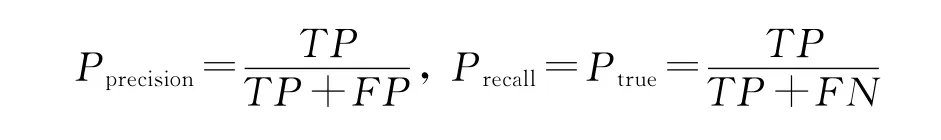
在这一模型中,TP(FP)定义为目前(没有)被注释为与精神分裂相关的、预测值大于临界值的基因数量;FN(TN)定义为目前(没有)被注释为与精神分裂症相关的、预测值小于临界值的基因数量.依据查准率和召回率得到查准率-召回率曲线(Precision Recall Curve)和AUC(Area Under Curve)分值来评估该模型的预测表现.同时根据基因特征和功能数据的预测,得到每个基因的预测表现数值,作为相关性高低的评估.
1.4 精神分裂症相关的GWAS数据
为了验证随机森林预测模型得到的候选基因是否确实在功能上同精神分裂症相关,GWAS中的显著性SNP位点能够作为有力的证据,我们结合GWAS数据做进一步的数据挖掘来验证预测得到的候选基因的SNP显著性.截至目前,已有8个大规模的关于精神分裂症的全基因组关联研究(见第269页附表2),但迄今为止公开的精神分裂症GWAS数据库只有两个,CAITE数据库(Clinical Antipsychotic Trials of Intervention Effectiveness)和GAIN数据库(Genetic Association Information Network[28]).本文采用GAIN数据库中的两个数据集,数据库具体信息详见ftp:∥ftp.ncbi.nlm.nih.gov/dbgap/GAIN/Schizophreni a/phs000021.v3.p2/manifest/.总共4591个样本,包括1217个白种病人样本(European Ancestry Cases)和1442个白种人对照样本(European Ancestry Controls),953个印第安血统病人(American Ancestry Cases)和979个印第安血统对照样本(American Ancestry Controls)用于普通研究数据,另有475个混合样本(187European Ancestry Cases,288American Ancestry Cases)数据用于精神分裂症和相关疾病研究数据.这两个GWAS数据都来自于AFFYMETRIX AFFY_6.0平台对934940个SNP位点进行筛查.我们试图通过这两个样本的GWAS数据来对候选基因的SNP显著性做验证并得到优选的精神分裂症相关基因.
2 结果与分析
2.1 精神分裂症相关基因的功能相似性
通过方法1.1我们找到了205个同精神分裂症相关的基因(见第268页附表1).我们用Fisher精确检验(Fisher's Exact Test)对这些基因的GO做了富集分析,根据P值的显著性(P<0.01)由小到大排序,我们得到了205个已知精神分裂症相关基因显著富集的功能注释,GO的生物通路(BP,Biological Process)、分子功能(MF,Molecular Function)、细胞成分(CC,Cellular Component)分析结果分别如表2、表3、表4(见第259~261页)(只列出前30个富集的GO).结果表明,205个基因有200个都已有功能注释,且富集的GO数目非常多(P<0.01,共有474个富集的基因功能:362个BP、61个MF、51个CC).结果显示,同神经系统密切相关的生物功能在这些基因中有非常明显的富集.以BP为例(表2),富集的功能主要集中在神经元的发生、发育及发育调控,神经冲动、神经递质、突触等传递与调控,也涉及核苷酸、环化酶、裂解酶等物质的生物合成、代谢与调控,在行为上也涉及行为、学习、认识等功能注释.由此可见搜集到的精神分裂症相关基因在功能上具有很大的相似性,彼此之间的生理功能紧密相连,且同神经系统密切相关.这一结果表明通过寻找到与已知致病基因有功能关联的基因来预测精神分裂症候选基因这一思路是可行的.
?
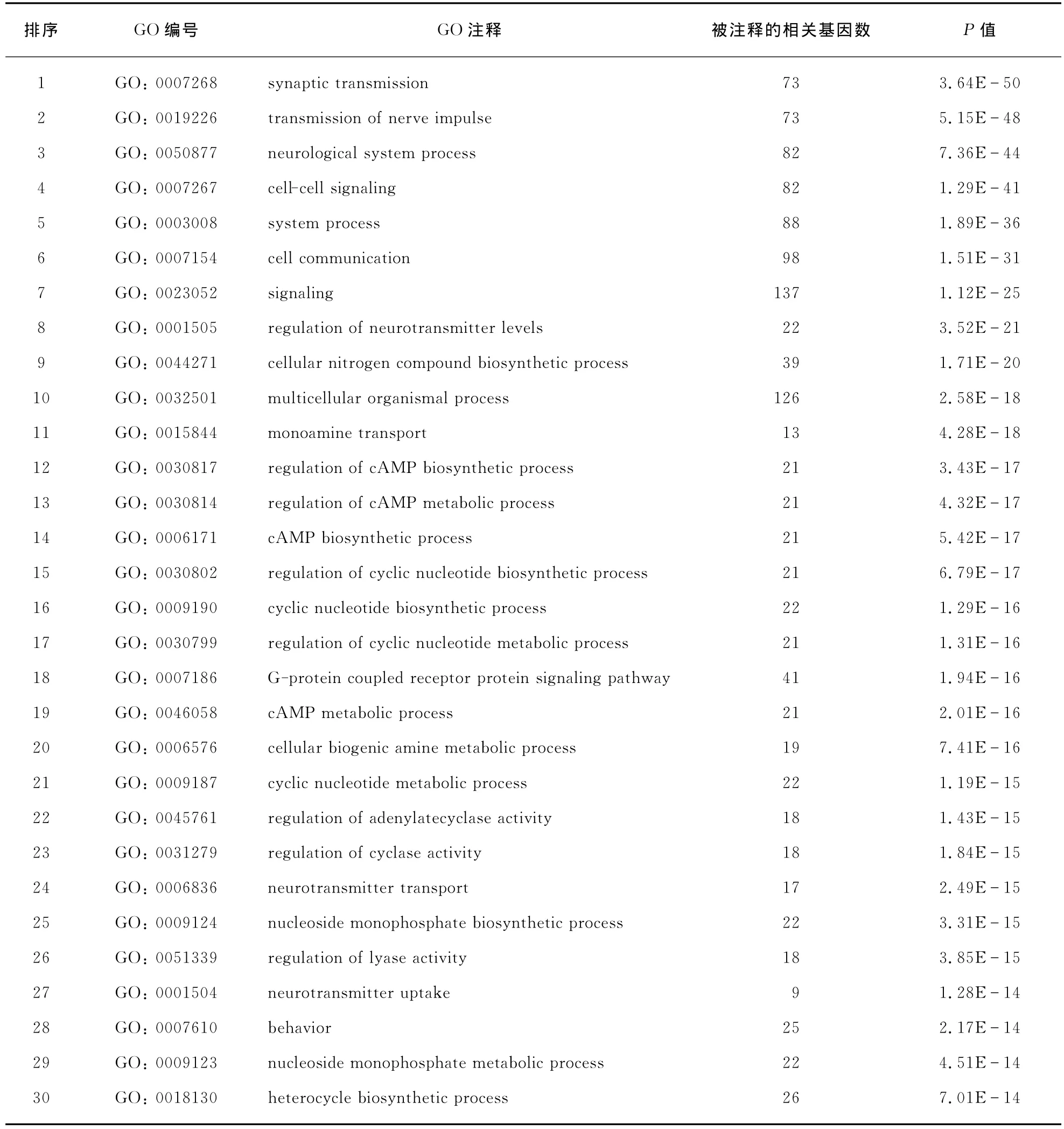
表2 已知的精神分裂症相关基因富集的GO(前30个生物通路注释)Tab.2 Enriched GO terms for known schizophrenia related genes(top 30BP)
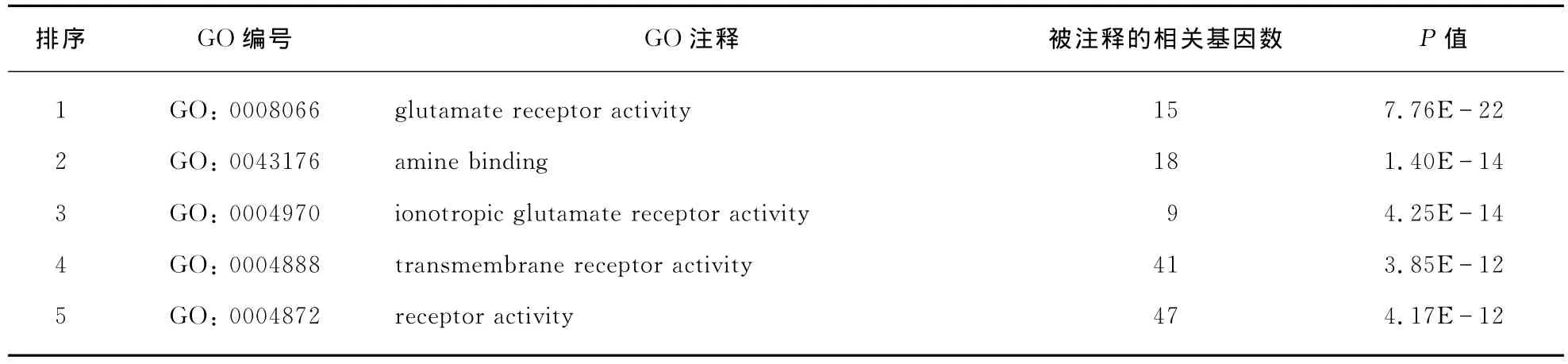
表3 已知的精神分裂症相关基因富集的GO(前30个分子功能注释)Tab.3 Enriched GO terms for known schizophrenia related genes(top 30MF)
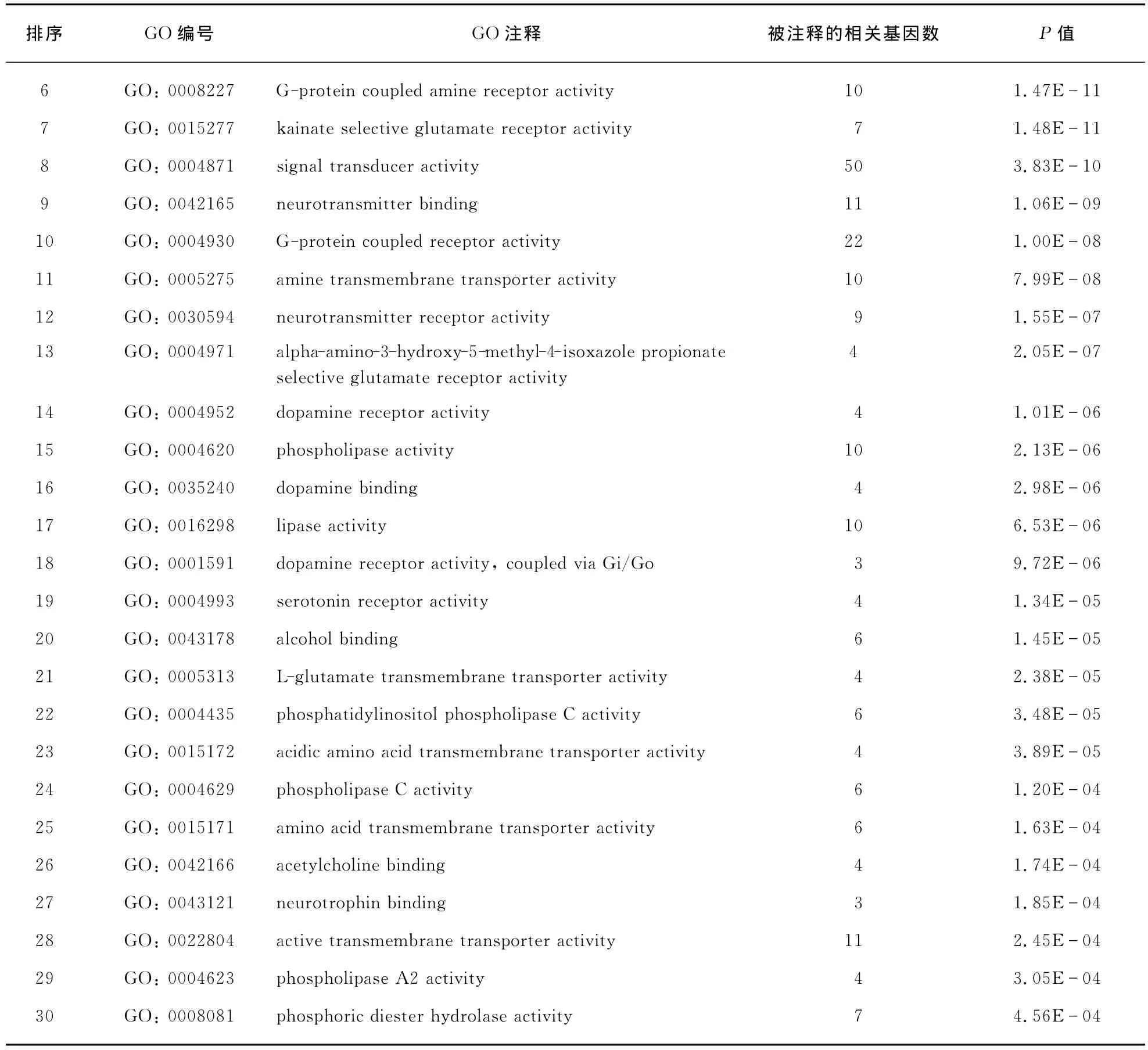
(续表)

表4 已知的精神分裂症相关基因富集的GO(前30个细胞成分注释)Tab.4 Enriched GO terms for known schizophrenia related genes(top 30CC)
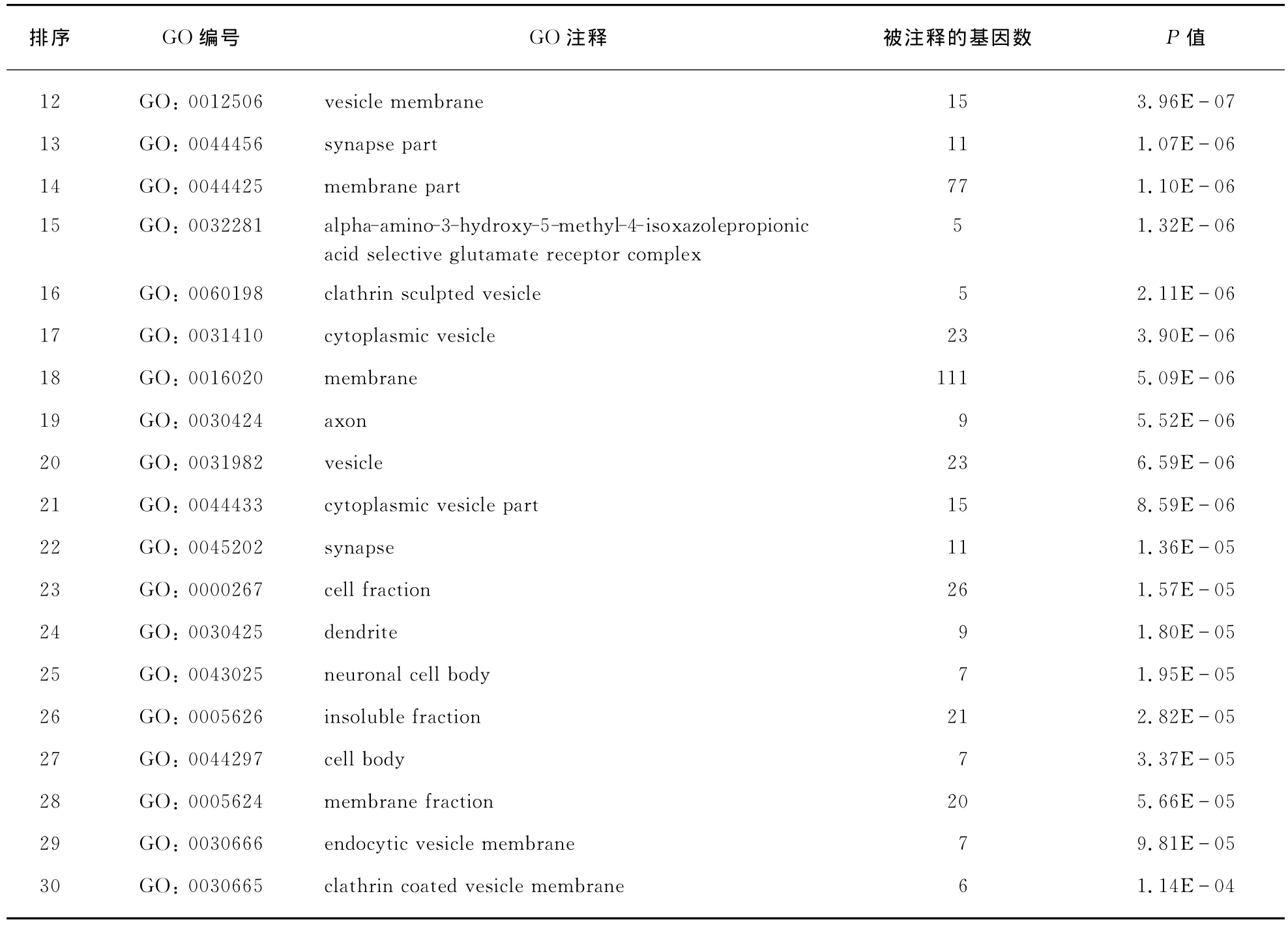
(续表)
2.2 随机森林预测模型的表现评估及获得的候选基因
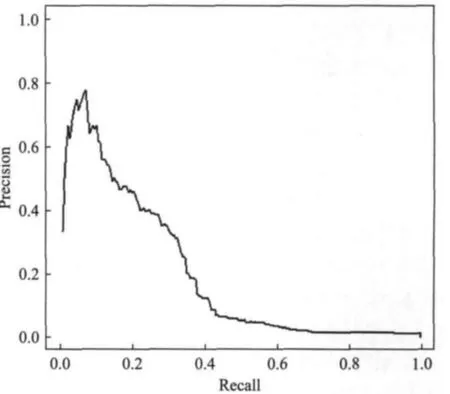
图2 随机森林模型预测精神分裂症相关基因的ROC曲线(AUC=0.1948)Fig.2 ROC curve of schizophrenia related gene prediction(AUC=0.1948)
将205个精神分裂症相关基因作为“同精神分裂症相关”这一表型输入到随机森林预测模型,通过预测分数筛选得到一组精神分裂症候选相关基因(Candidate Genes).预测模型的Precision Recall Curve如图2,AUC值为0.1948,说明通过该模型预测得到的精神分裂症相关基因相比随机基因的集中度达到19.48%,预测效果较好.
在本模型中共包含21147个基因,按照预测分数,要从这个基因库中选取同精神分裂症相关度较高的候选基因进行重点分析.在这21147个基因中,预测分数最大为0.6377,最小为0.0052,为了获取最可靠的候选基因,我们倾向于选择较高的预测分数为阈值.结合查准率 召回率曲线(图2),预测分数为0.20时对应的查准率为0.48,召回率为0.16,因此选择预测分数0.20为筛选候选基因的阈值,既保证了非常高的预测准确率,又能保证良好的召回率.筛选所有预测分数大于0.20的基因,共得到精神分裂症候选相关基因33个.若需要更多的精神分裂症候选基因进行进一步验证,即降低查准率,提高召回率,亦可自主选择更低的阈值以满足需求.按预测值从高到低排列,这33个候选基因的列表见表5.
为了验证这些基因同精神分裂症的相关性,我们在Pubmed门户网站中对其进行文献检索,发现其中20个基因在其他研究中被发现是精神分裂症的疑似相关或者易感基因[29-48],其中4个基因被发现同其他神经系统疾病如躁动症(Bipolar Disease)等相关[33,49-51].研究这33个基因的功能注释,发现这些候选基因基本上都同神经系统及其相关功能有较密切的联系.综上所述,我们初步证明了随机森林模型预测得到的候选基因的可靠性.
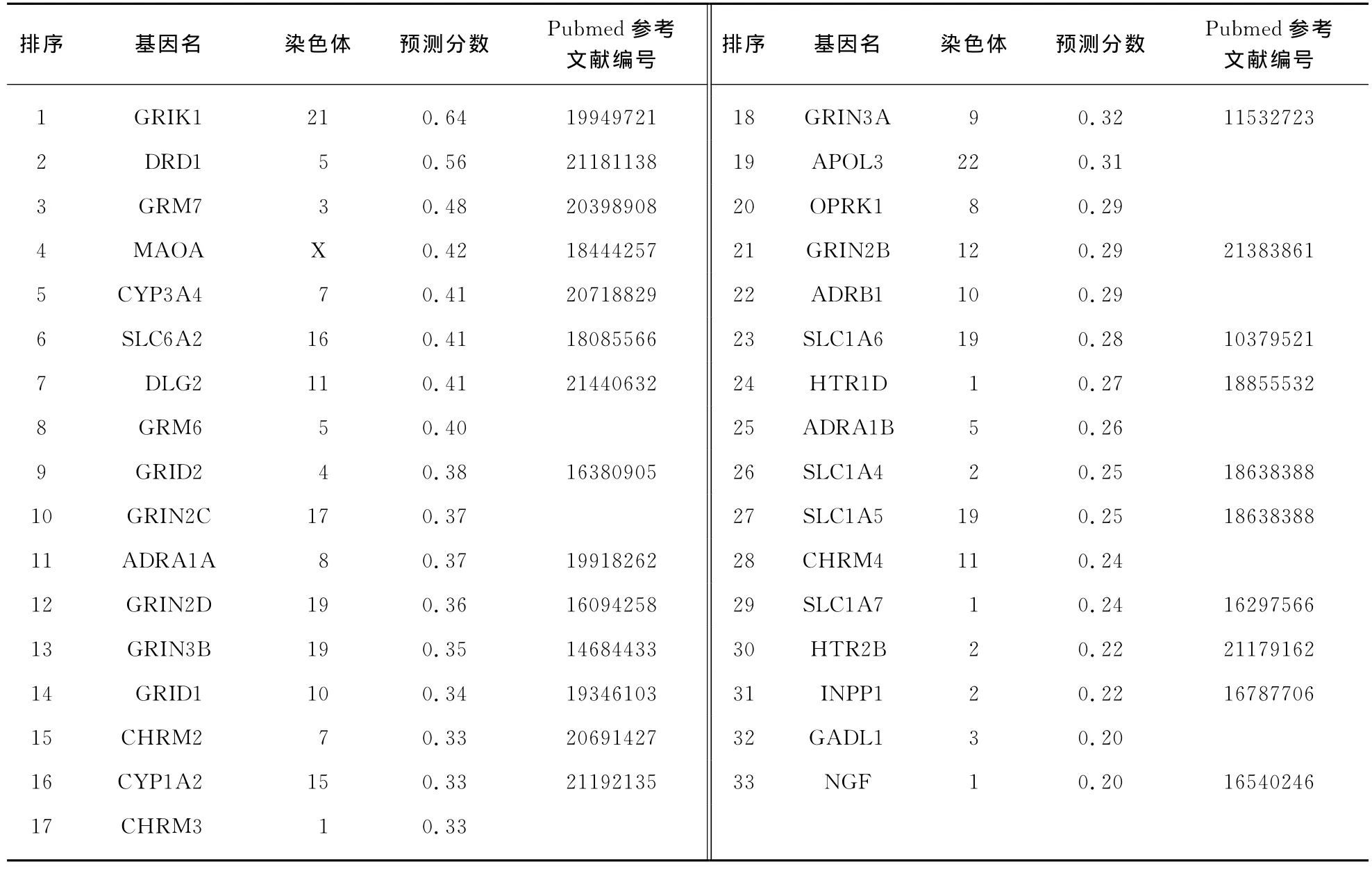
表5 随机森林模型预测的候选基因列表Tab.5 Candidate genes predicted by random forests model
2.3 GWAS数据的验证结果及优选的精神分裂症候选基因
使用白种人血统(EA,European Ancestry)和印第安人血统(AA,African Ancestry)的两组数据,筛选出位于以上33个候选基因上下游各100bp区域的SNP显著性位点(P<0.01).在这33个基因中,总共筛选到位于其中10个基因上的58个显著SNP位点(EE:28个,AE:30个),具体位点和基因列表如表6.33个基因中有10个被GWAS数据验证,验证率为30.3%.这10个具有显著SNP位点的基因被定义为最终优选的精神分裂症候选相关基因.
观察这58个SNP位点,发现在其他文献或研究中提及,与精神分裂症这一疾病或相近的疾病(如躁郁症Bipolar Schizoaffective Disorder)有关联的SNP有28个[32,52-56],占预测得到的显著SNP位点个数的48.2%.得到验证的SNP位点主要集中在GADL1,GRIN2B,GRM7,SLC6A2这几个基因中.结合预测数值的结果,我们发现这四个基因的预测分数分别为0.2003(GADL1),0.2914(GRIN2B),0.4797(GRM7),0.4093(SLC6A2),按照预测数值从高到低排序分别排在第3位(GRM7),第6位(SLC6A2),第21位(GRIN2B),第32位(GADL1).这一结果表明,通过模型预测得到的精神分裂症相关度和通过GWAS数据得到的显著性SNP位点和精神分裂症的相关性存在一致性.借助两种结果共同分析和验证,我们着重挑选GRM7和SLC6A2这两个基因,既在基因功能上存在同精神分裂症的密切关系,在大规模全基因关联研究中也能证明其SNP位点的显著性,值得进一步做相关性的研究.
结合GWAS研究中显著的SNP位点,我们在之前通过预测模型富集到的33个核心基因中进一步得到有SNP显著性位点的10个基因,其中9个有同精神分裂症相关的基因功能注释.这些分析说明该复杂疾病相关基因的预测模型能够优选到相关度较高的疾病基因,可以作为筛选基因的手段,同时为进一步研究精神分裂症的相关基因提供了范围和基础.

表6 具有显著SNP位点的候选基因列表Tab.6 Candidate genes with significant SNP site according to GWAS data
3 讨 论
借助数据库和文献搜索,我们得到了205个同精神分裂症相关的基因,通过随机森林的生物信息预测模型,取预测临界值0.20初筛得到33个候选基因.通过两个GWAS数据集验证得到其中10个基因存在58个SNP显著性位点.其中发现SNP显著性位点集中在GRM7,SLC6A2,GRIN2B和GADL1这四个基因区域,结合已有的研究和文献,发现GRM7基因有较为可靠的证据证明可能同精神分裂症相关,2010年O'Connor RM等提到GRM7基因是相关神经认知和情感的基因[57],另外除GAIN的两个数据集外还有其他全基因组关联研究证明GRM7同精神分裂症的相关性[58-59].对于SCL6A2基因,目前已进行的关联研究并没有发现该基因同精神分裂症的直接关系,但它同情绪紊乱的发病机制可能相关,并被认为是惊恐障碍的相关基因[60].探究GRIN2B同精神分裂症关联的研究已经比较深入,都认为可以将其作为精神分裂症的易感基因[61-64].同时还有一些研究可以证明GRIN2B还与其他一些复杂的神经类疾病相关,如帕金森综合征(Parkinson Disease)[65]、躁动症(Bipolar Disease)[62]、老年痴呆症(Alzheimer's Disease)[66].GADL1作为一个功能还没有被研究透彻的基因,只有为数不多的研究检测到其精神分裂症和躁动症的显著性位点.这些文献结果表明本文结合随机森林模型预测以及GWAS验证分析得到的优选基因同精神分裂症这一疾病的相关度是很高的.
除了关注以上文献中有提及的优选候选基因以外,我们也应该关注一些新被筛选出的基因(Novelty Disease Genes),这些基因位于预测分数的前列,同时也存在显著性的SNP位点,比如GRIN3A和DLG2,经过更多的研究和验证,他们可能会成为新的疾病靶位基因.
值得一提的是,虽然本疾病基因预测模型建立在功能相关和基因组关联SNP显著位点这两个特性的结合,但仍然存在一些问题值得探讨.一方面,在GAIN GWAS的数据库选择上,由于目前开放的GWAS数据库没有完全整合,数据较为松散和不规整,本研究中只选取了两个数据集(EA和AA)作为验证候选基因的数据库,并得到了10个优选基因.我们认为,随着GWAS数据平台建立的成熟和GWAS数据公开性的扩展,能够得到更多有价值的显著性SNP位点,帮助进一步优选模型中预测的候选基因.另一方面,除了通过SNP位点进行分析验证以外,CNV也是一个验证方向.GWAS数据也包括CNV数据,有不少研究表明精神分裂症同基因拷贝数的异常相关(附录表2中已列出),如有研究发现1q21.1,15q11.2和15q13.3三个位点同精神分裂症相关[12].因此,从基因拷贝数异常的显著性来优选精神分裂症相关基因这一方向也可以进一步做探究.
从随机森林模型的预测表现来看,AUC值为0.1948,意味着相比随机选取,基因优选度达到19.48%.相比其他优选复杂疾病基因的生物信息学方法[6],本模型使用的数据库类型更为多样,依托机器学习的计算生物学方法,更有助于从功能注释与关联以及全基因组关联研究两个层面预测复杂疾病的相关基因.
[1]Mueser K T,McGurk S R.Schizophrenia[J].Lancet,2004,363(9426):2063-2072.
[2]Christopher A,Ross R L M,Sarah A J.Neurobiology of Schizophrenia[J].Neuron,2006,52:14.
[3]Shi J,Levinson D F,Duan J,et al.Common variants on chromosome 6p22.1are associated with schizophrenia[J].Nature,2009,460(7256):753-757.
[4]Schork N J,Murray S S,Frazer K A,et al.Common vs.rare allele hypotheses for complex diseases[J].Current Opinion in Genetics &Development,2009,19(3):212-219.
[5]Sun J,Kuo P H,Riley B P,et al.Candidate genes for schizophrenia:A survey of association studies and gene ranking[J].American Journal of Medical Genetics Part B:Neuropsychiatric Genetics,2008,147B(7):1173-1181.
[6]Sun J,Jia P,Fanous A H,et al.A multi-dimensional evidence-based candidate gene prioritization approach for complex diseases-schizophrenia as a case[J].Bioinformatics,2009,25(19):2595-6602.
[7]Yonan A L,Palmer A A,Smith K C,et al.Bioinformatic analysis of autism positional candidate genes using biological databases and computational gene network prediction[J].Genes Brain Behav,2003,2(5):303-320.
[8]Reif A,Kao C F,Fang Y S,et al.Prioritization and evaluation of depression candidate genes by combining multidimensional data resources[J].PLoS ONE,2011,6(4):e18696.
[9]Turner F S,Clutterbuck D R,Semple CAM.POCUS:mining genomic sequence annotation to predict disease genes[J].Genome Biology,2003,4(11):R75.
[10]Tian W D,Zhang L V,Murat-Taşan M,et al.Combining guilt-by-association and guilt-by-profiling to predict Saccharomyces cerevisiae gene function[J].Genome Biology,2008,9:S7.
[11]Taşan M,Tian W D,Hill D P,et al.An en masse phenotype and function prediction system for Mus musculus[J].Genome Biology,2008,9:S8.
[12]Stefansson H,Rujescu D,Cichon S,et al.Large recurrent microdeletions associated with schizophrenia[J].Nature,2008,455(7210):232-261.
[13]McClellan J M,Susser E,King M C.Schizophrenia:a common disease caused by multiple rare alleles[J].British Journal of Psychiatry,2007,190:194-199.
[14]Xu B,Roos J L,Levy S,et al.Strong association of de novo copy number mutations with sporadic schizophrenia[J].Nature Genetics,2008,40(7):880-885.
[15]McCarthy M I,Need A C,Ge D,et al.A genome-wide investigation of SNPs and CNVs in schizophrenia[J].PLoS Genetics,2009,5(2):e1000373.
[16]Stone J L,O'Donovan M C,Gurling H,et al.Rare chromosomal deletions and duplications increase risk of schizophrenia[J].Nature,2008,455(7210):237-241.
[17]Walsh T,McClellan J M,McCarthy S E,et al.Rare structural variants disrupt multiple genes in neurodevelopmental pathways in schizophrenia[J].Science,2008,320(5875):539-543.
[18]Stefansson H,Ophoff R A,Steinberg S,et al.Common variants conferring risk of schizophrenia[J].Nature,2009,460(7256):744-799.
[19]Sanders A R,Duan J,Levinson D F,et al.No significant association of 14candidate genes with schizophrenia in a large European ancestry sample:implications for psychiatric genetics[J].American Journal of Psychiatry,2008,165(4):497-506.
[20]Munafo M R,Bowes L,Clark T G,et al.Lack of association of the COMT(Val158/108Met)gene and schizophrenia:a meta-analysis of case-control studies[J].Mol Psychiatry,2005,10(8):765-770.
[21]McKusick V A.Mendelian inheritance in man:a catalog of human genes and genetic disorders[M].12th edition.Baltimore:Johns Hopkins University Press,1998.
[22]Jensen L J,Saric J,Bork P.Literature mining for the biologist:from information retrieval to biological discovery[J].Nature Reviews Genetics,2006,7(2):119-129.
[23]Safran M,Dalah I,Alexander J,et al.GeneCards Version 3:the human gene integrator[DB/OL].2010-12-01.http:∥www.genecards.org/.
[24]Allen N C,Bagade S,McQueen M B,et al.Systematic meta-analyses and field synopsis of genetic association studies in schizophrenia:the SzGene database[J].Nature Genetics,2008,40(7):827-834.
[25]Jia P,Sun J,Guo A Y,et al.SZGR:a comprehensive schizophrenia gene resource[J].Molecular Psychiatry,2010,15(5):453-462.
[26]Breiman L.Random forest[J].Machine Learn,2001(45):5-32.
[27]Breiman L.Classification and regression trees.The Wadsworth statistics/probibility series[M].Belmont,Calif.:Wadsworth International Group,1984:358.
[28]Manolio T A,Rodriguez L L,Brooks L,et al.New models of collaboration in genome-wide association studies:the genetic association information network[J].Nature Genetics,2007,39(9):1045-1051.
[29]Jockers-Scherubl M C,Rentzsch J,Danker-Hopfe H,et al.Adequate antipsychotic treatment normalizes serum nerve growth factor concentrations in schizophrenia with and without cannabis or additional substance abuse[J].Neurosci Lett,2006,400(3):262-266.
[30]Liu Y R,Loh E W,Lan TH,et al.ADRA1Agene is associated with BMI in chronic schizophrenia patients exposed to antipsychotics[J].Pharmacogenomics J,2010,10(1):30-39.
[31]Lai I C,Mo G H,Chen M L,et al.Analysis of genetic variations in the dopamine D1receptor(DRD1)gene and antipsychotics-induced tardive dyskinesia in schizophrenia[J].Eur J Clin Pharmacol,2011,67(4):383-388.
[32]Deng X,Sagata N,Takeuchi N,et al.Association study of polymorphisms in the neutral amino acid transporter genes SLC1A4,SLC1A5and the glycine transporter genes SLC6A5,SLC6A9with schizophrenia[J].BMC Psychiatry,2008,8:58.
[33]Michelon L,Meira-Lima I,Cordeiro Q,et al.Association study of the INPP1,5HTT,BDNF,AP-2beta and GSK-3beta GENE variants and restrospectively scored response to lithium prophylaxis in bipolar disorder[J].Neurosci Lett,2006,403(3):288-293.
[34]Fallin M D,Lasseter V K,Avramopoulos D,et al.Bipolar I disorder and schizophrenia:a 440-singlenucleotide polymorphism screen of 64candidate genes among Ashkenazi Jewish case-parent trios[J].Am J Hum Genet,2005,77(6):918-936.
[35]Klimek V,Rajkowska G,Luker S N,et al.Brain noradrenergic receptors in major depression and schizophrenia[J].Neuropsychopharmacology,1999,21(1):69-81.
[36]Saus E,Brunet A,Armengol L,et al.Comprehensive copy number variant(CNV)analysis of neuronal pathways genes in psychiatric disorders identifies rare variants within patients[J].J Psychiatr Res,2010,44(14):971-978.
[37]Treutlein J,Muhleisen TW,Frank J,et al.Dissection of phenotype reveals possible association between schizophrenia and Glutamate Receptor Delta 1(GRID1)gene promoter[J].Schizophr Res,2009,111(1-3):123-130.
[38]Smith R E,Haroutunian V,Davis K L,et al.Expression of excitatory amino acid transporter transcripts in the thalamus of subjects with schizophrenia[J].Am J Psychiatry,2001,158(9):1393-1399.
[39]Choi K H,Zepp M E,Higgs B W,et al.Expression profiles of schizophrenia susceptibility genes during human prefrontal cortical development[J].J Psychiatry Neurosci,2009,34(6):450-458.
[40]Jagannathan K,Calhoun V D,Gelernter J,et al.Genetic associations of brain structural networks in schizophrenia:apreliminary study[J].Biol Psychiatry,2010,68(7):657-666.
[41]Cacabelos R,Martinez-Bouza R.Genomics and pharmacogenomics of schizophrenia[J].CNS Neurosci Ther,2010,17(5):541-565.
[42]Makino C,Shibata H,Ninomiya H,et al.Identification of single-nucleotide polymorphisms in the human N-methyl-D-aspartate receptor subunit NR2Dgene,GRIN2D,and association study with schizophrenia[J].Psychiatr Genet,2005,15(3):215-221.
[43]Maclaren E J,Charlesworth P,Coba M P,et al.Knockdown of mental disorder susceptibility genes disrupts neuronal network physiology in vitro[J].Mol Cell Neurosci,2011,47(2):93-99.
[44]Haavik J,Blau N,Thony B.Mutations in human monoamine-related neurotransmitter pathway genes[J].Hum Mutat,2008,29(7):891-902.
[45]Meary A,Brousse G,Jamain S,et al.Pharmacogenetic study of a typical antipsychotic drug response:involvement of the norepinephrine transporter gene[J].Am J Med Genet B Neuropsychiatr Genet,2008,147B(4):491-494.
[46]Myers R A,Casals F,Gauthier J,et al.A population genetic approach to mapping neurological disorder genes using deep resequencing[J].PLoS Genet,2011,7(2):e1001318.
[47]Lauriat T L,Dracheva S,Chin B,et al.Quantitative analysis of glutamate transporter mRNA expression in prefrontal and primary visual cortex in normal and schizophrenic brain[J].Neuroscience,2006,137(3):843-851.
[48]Ikeda M,Yamanouchi Y,Kinoshita Y,et al.Variants of dopamine and serotonin candidate genes as predictors of response to risperidone treatment in first-episode schizophrenia[J].Pharmacogenomics,2008,9(10):1437-1443.
[49]Lipsky R H,Goldman D.Genomics and variation of ionotropic glutamate receptors[J].Ann N Y Acad Sci,2003,1003:22-35.
[50]Skogh E,Sjodin I,Josefsson M,et al.High correlation between serum and cerebrospinal fluid olanzapine concentrations in patients with schizophrenia or schizoaffective disorder medicating with oral olanzapine as the only antipsychotic drug[J].J Clin Psychopharmacol,2011,31(1):4-9.
[51]Bevilacqua L,Doly S,Kaprio J,et al.A population-specific HTR2Bstop codon predisposes to severe impulsivity[J].Nature,2010,468(7327):1061-1066.
[52]Holmans P A,Riley B,Pulver A E,et al.Genomewide linkage scan of schizophrenia in a large multicenter pedigree sample using single nucleotide polymorphisms[J].Molecular Psychiatry,2009,14(8):786-795.
[53]Hamshere M L,Green E K,Jones I R,et al.Genetic utility of broadly defined bipolar schizoaffective disorder as a diagnostic concept[J].British Journal of Psychiatry,2009,195(1):23-29.
[54]Yeh Y W,Lu R B,Tao P L,et al.A possible association of the norepinephrine transporter gene in the development of heroin dependence in Han Chinese[J].Pharmacogenetics and Genomics,2011,21(4):197-205.
[55]Shibata H,Tani A,Chikuhara T,et al.Association study of polymorphisms in the group III metabotropic glutamate receptor genes,GRM4and GRM7,with schizophrenia[J].Psychiat Res,2009,167(1-2):88-96.
[56]Neale B M,Faraone S V.Perspective on the genetics of attention deficit/hyperactivity disorder[J].American Journal of Medical Genetics Part B-Neuropsychiatric Genetics,2008,147B(8):1334-1336.
[57]O'Connor R M,Finger B C,Flor P J,et al.Metabotropic glutamate receptor 7:At the interface of cognition and emotion[J].European Journal of Pharmacology,2010,639(1-3):123-131.
[58]Ganda C,Schwab S G,Amir N,et al.A family-based association study of DNA sequence variants in GRM7with schizophrenia in an Indonesian population[J].Int J Neuropsychoph,2009,12(9):1283-1289.
[59]Ohtsuki T,Koga M,Ishiguro H,et al.A polymorphism of the metabotropic glutamate receptor mGluR7(GRM7)gene is associated with schizophrenia[J].Schizophrenia Research,2008,101(1-3):9-16.
[60]Sand P G,Mori T,Godau C,et al.Norepinephrine transporter gene(NET)variants in patients with panic disorder[J].Neuroscience Letters,2002,333(1):41-44.
[61]Di Maria E,Gulli R,Begni S,et al.Variations in the NMDA receptor subunit 2Bgene(GRIN2B)and schizophrenia:A case-control study[J].American Journal of Medical Genetics Part B-Neuropsychiatric Genetics,2004,128B(1):27-29.
[62]Martucci L,Wong A H C,De Luca V,et al.N-methyl-D-aspartate receptor NR2Bsubunit gene GRIN2B in schizophrenia and bipolar disorder:polymorphisms and mRNA levels[J].Schizophrenia Research,2006,84(2-3):214-221.
[63]Li D W,He L.Association study between the NMDA receptor 2Bsubunit gene(GRIN2B)and schizophrenia:A HuGE review and meta-analysis[J].Genet Med,2007,9(1):4-8.
[64]Ohtsuki T,Sakurai K,Dou H,et al.Mutation analysis of the NMDAR2B(GRIN2B)gene in schizophrenia[J].Molecular Psychiatry,2001,6(2):211-216.
[65]Tsai S J,Liu H C,Liu T Y,et al.Association analysis for genetic variants of the NMDA receptor 2b subunit(GRIN2B)and Parkinson's disease[J].J Neural Transm,2002,109(4):483-488.
[66]Tsai S J,Liu H C,Liu T Y,et al.Association analysis for the genetic variants of the NMDA receptor subunit 2band Alzheimer's disease[J].Dement Geriatr Cogn,2002,13(2):91-94.
[67]Lencz T,Morgan T V,Athanasiou M,et al.Converging evidence for a pseudoautosomal cytokine receptor gene locus in schizophrenia[J].Molecular Psychiatry,2007,12(6):572-580.
[68]Sullivan P F,Lin D,Tzeng J Y,et al.Genomewide association for schizophrenia in the CATIE study:results of stage 1[J].Molecular Psychiatry,2008,13(6):570-584.
[69]O'Donovan M C,Craddock N,Norton N,et al.Identification of loci associated with schizophrenia by genome-wide association and follow-up[J].Nature Genetics,2008,40(9):1053-1055.
[70]Shifman S,Johannesson M,Bronstein M,et al.Genome-wide association identifies a common variant in the reelin gene that increases the risk of schizophrenia only in women[J].PLoS Genetics,2008,4(2):e28.
[71]Kirov G,Zaharieva I,Georgieva L,et al.A genome-wide association study in 574schizophrenia trios using DNA pooling[J].Molecular Psychiatry,2009,14(8):796-803.
[72]Stefansson H,Ophoff R A,Steinberg S,et al.Common variants conferring risk of schizophrenia[J].Nature,2009,460(7256):744-747.
[73]Purcell S M,Wray N R,Stone J L,et al.Common polygenic variation contributes to risk of schizophrenia and bipolar disorder[J].Nature,2009,460(7256):748-752.
