胰腺癌细胞株PANC1异常甲基化miRNA的分析
2012-11-06彭泉张立洁蔡辉华高文涛赵成功钱祝银苗毅
彭泉 张立洁 蔡辉华 高文涛 赵成功 钱祝银 苗毅
·论著·
胰腺癌细胞株PANC1异常甲基化miRNA的分析
彭泉 张立洁 蔡辉华 高文涛 赵成功 钱祝银 苗毅
目的分析胰腺癌细胞株PANC1与正常胰腺组织表达有差异的启动子区甲基化miRNA,寻找与胰腺癌相关的高甲基化miRNA。方法抽提PANC1与正常胰腺组织基因组DNA,超声断裂。应用抗5-甲基化嘧啶核苷抗体和免疫磁珠法获取甲基化DNA片段。通过DNA甲基化芯片筛选出PANC1与正常胰腺组织表达差异的高甲基化miRNA,采用重亚硫酸盐修饰的PCR(BSP)和TA克隆测序的方法进行验证。提取胰腺癌细胞株BxPC3、CFPAC1、PANC1、SW1990基因组DNA,采用结合重亚硫酸盐的限制性内切酶法(combined bisulfite restriction analysis,COBRA)验证芯片筛选出的差异表达的甲基化miRNA。结果PANC1细胞与正常胰腺组织存在8个差异表达的甲基化miRNA,从中挑选出5个进行BSP+TA克隆测序验证,其中miR-615、miR-663、miR-663b在PANC1细胞的甲基化率明显高于正常组织(60.6%比7.6%,88.8%比22.2%,94.4%比13.0%);miR-675的甲基化率与正常胰腺组织无明显差异(76.0%比100%);miR1826因测序结果误差较大而舍去。经COBRA验证,PANC1的上述4种miRNA均高甲基化;BxPC除miR-675外,其他3种均高甲基化;CFPAC1的miR-663、miR-663b高甲基化;SW1990的miR-615、miR-663高甲基化。结论胰腺癌细胞株与正常胰腺组织间存在差异表达的高甲基化miRNA,其中miR-663高甲基化与胰腺癌可能相关。
胰腺肿瘤; DNA甲基化; 微RNAs; TA克隆
DNA甲基化(DNA methylation)是最早被发现的表观遗传学改变途径之一。位于启动子区的CpG岛甲基化通常会导致基因表达沉默,是许多肿瘤发生中一个频发的早期事件。微小RNA(miRNAs)是重要的转录后调节机制[1],在肿瘤中表达异常,并且调节信号转导、细胞增殖、凋亡等重要生物学过程。自Saito等[2]在2006年首次报道miRNA-127受到DNA甲基化调控起抑癌基因作用以来,miRNA的启动子高甲基化导致其表达下调的报道增多[3-7]。本研究旨在寻找胰腺癌相关的异常甲基化的miRNA,以期对胰腺癌的诊断和治疗发挥推动作用。
材料与方法
一、材料
胰腺癌细胞株PANC1、BxPC3、CFPAC1、SW1990均购自上海细胞库,常规培养、传代,取对数生长期细胞。3例正常胰腺组织取自意外死亡者,均获得家属同意,并签署知情书。
二、DNA甲基化芯片分析
应用DNA提取试剂盒(Omega公司)抽提胰腺癌细胞株PANC1与正常胰腺组织基因组DNA,超声打断成400~500 bp的DNA片段,加热变性为单链DNA。部分单链DNA样品中加入抗5-甲基化胞嘧啶核苷抗体,使用免疫磁珠法分离甲基化的DNA片段。将甲基化DNA样本及未处理的DNA片段样本分别进行扩增,荧光标记,标记后将两样本混合、变性、与上海康成生物公司提供的甲基化DNA微阵列芯片杂交,用高解析度芯片扫描仪检测杂交信号,对结果进行数据分析。
三、重亚硫酸盐修饰的PCR(BSP)和TA克隆测序
对PANC1和正常胰腺组织的基因组DNA进行重亚硫酸盐处理,使未甲基化的胞嘧啶转化成胸腺嘧啶,而甲基化的胞嘧啶不变。亚硫酸氢盐修饰过程见文献[8]。以重亚硫酸盐处理后的DNA作为模板,采用Touchdown-PCR方法扩增。引物由Invitrogen公司设计并合成。引物尽量设计位于miRNA发夹序列的上游500 bp之内[9],miRNA引物序列及与相邻CpG位置、扩增范围见表1和图1。扩增产物经电泳分离,回收、纯化。纯化的扩增产物行TA克隆。挑选6个阳性单克隆菌落的菌液行PCR,并送Invitrogen公司测序。

表1 miRNA引物序列及扩增位置
注:+:表示在发夹序列上游,-:表示在下游

图1UCSC数据库上miRNA与相邻CpG岛位置及BSP扩增范围
四、结合重亚硫酸盐的限制性内切酶法(combined bisulfite restriction analysis,COBRA)
提取胰腺癌细胞株BXPC3、CFPAC1、PANC1、SW1990基因组DNA,行BSP,应用内切酶对PCR产物进行酶切。酶切8 h后以2%琼脂糖凝胶电泳分离PCR产物,伊文思兰(EB)染色,成像。
结 果
一、DNA甲基化芯片分析结果
芯片分析公司提供序列上游2000 bp范围内覆盖10个以上探针的miRNA共52个,其中PANC1细胞株与正常胰腺组织甲基化程度差异明显的miRNA共8个,分别为miR-483、miR-574、miR-615、miR-663、miR-663b、miR-675、miR-1247、miR-1258、miR-1826(图2)。

图2在PANC1和正常胰腺组织差异表达的甲基化miRNA(绿色部分)
二、BSP+TA克隆测序结果
随机挑选5个miRNA(miR-615、miR-663、miR-663b、miR-675、miR-1826)进行BSP。PCR反应:95℃ 5 min,94℃ 30 s、68℃ 30 s、72℃ 30 s,2个循环;95℃ 5 min,94℃ 30 s、66℃ 30 s、72℃ 30 s,3个循环;95℃ 5 min,94℃ 30 s、64℃ 30 s、72℃ 30 s,4个循环;95℃ 5 min,94℃ 30 s、62℃ 30 s、72℃ 30 s,35个循环,均获得目的条带(图3)。TA克隆阳性菌液测序结果见图4。miR-615、miR-663、miR-663b在PANC1中的甲基化率明显高于正常组织(60.6%比7.6%,88.8%比22.2%,94.4%比13.0%);miR-675在PANC1和正常胰腺组织中的甲基化率无明显差异(76.0%比100%);miR-1826因测序结果误差大而舍去。

图3 5个miRNA的 BSP产物电泳图

图4miR-615、miR-663、miR-663b、miR-675经TA克隆后测序结果 实心圆:甲基化的CpG岛;空心圆:未发生甲基化的CpG岛
三、COBRA验证结果
PANC1的上述4种miRNA均高甲基化;BxPC3除miR-675外,其他3种均高甲基化;CFPAC1的miR-665、miR-663b高甲基化;SW1990的miR-615、miR-663高甲基化(图5)。
讨 论
miRNA是21~25 nt长的单链小分子RNA,广泛存在于真核生物中。miRNA参与生命过程中一系列的重要进程,包括早期胚胎发育、细胞分化、增殖和凋亡等,其中的一些miRNA可以调控正常细胞周期和终末分化,如发生突变可导致细胞增殖和凋亡异常。近年来专家们对与肿瘤相关的miRNA的研究取得不少新进展。Chen等[10]发现,miR-17-192与miR-155在鼻咽癌中表达上调;miR-34、miR-143和miR-145的表达则显著下调。这些表达下调的miRNA与多种生物学途径相关,如TGF-Wnt途径、VEGF信号转导通路和IP3信号转导通路等。此外,在包括胰腺癌在内的多种肿瘤组织中亦发现多种miRNA的表达和功能异常[11-14]。Lu等[15]还证明miRNA的表达特征能用于划分人类癌症以及区别正常细胞和癌细胞,表明miRNA的活动模式还能够被用于诊断癌症。2009年Rabinowits等[16]的研究已指出,miRNA可以用来对肺癌进行筛选。
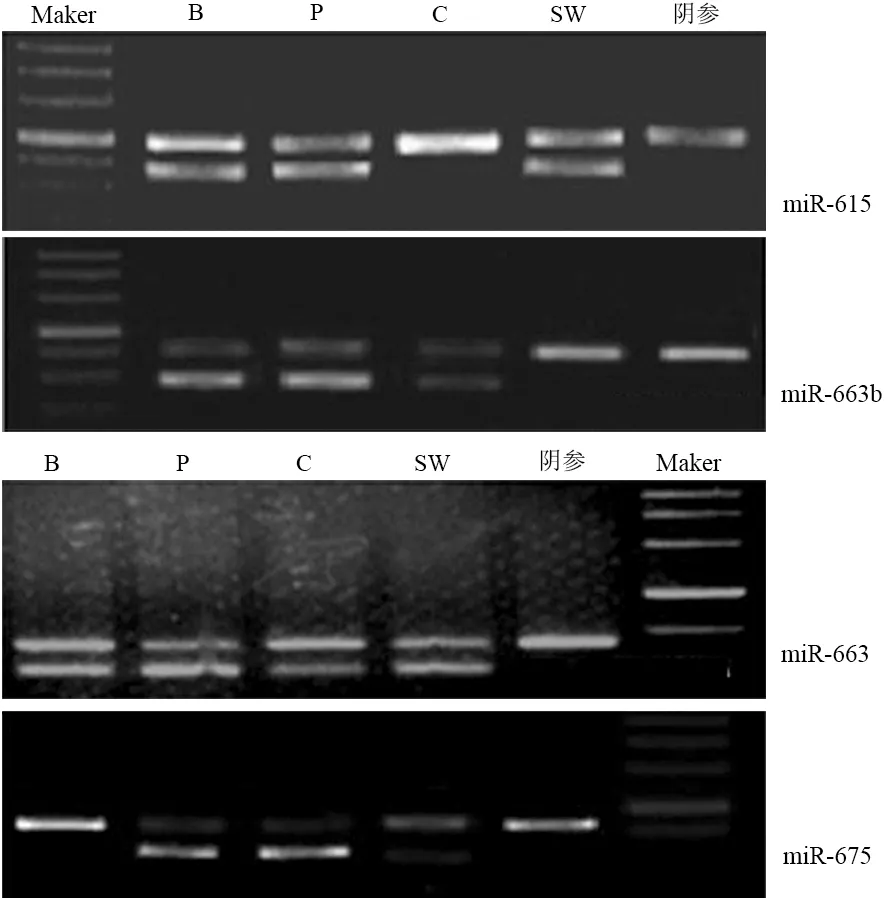
B:BxPC1;P:PANC1;C:CFPAC1;SW:SW1990
图54个miRNA在胰腺癌细胞株中的甲基化状况
目前,对miRNA发生异常表达的调控机制研究尚处于初级阶段,但已有研究证实miRNA受多种因素调控,例如染色质结构异常、miRNA加工受损等。近几年有多篇报道指出,启动子区的甲基化程度会影响miRNA的表达水平,表明DNA甲基化异常也是miRNA表达异常的重要机制之一[3-7]。
本实验通过甲基化芯片筛选出52个miRNA,选取其中5个进行TA 克隆验证,发现miR-615、miR-663、miR-663b在胰腺癌细胞株PANC1中存在高甲基化,并通过BxPC3、CFPAC1、SW1990胰腺癌细胞验证,只有miR-663在4株胰腺癌细胞中均为高甲基化,它可能是胰腺癌相关的特异性的高甲基化miRNA。当然,这还有待于在大样本的胰腺癌组织标本中进行验证。
[1] Croce CM, Calin GA. miRNAs, cancer, and stem cell division. Cell,2005,122:6-7.
[2] Saito Y, Liang G, Egger G, et al. Specific activation of microRNA-127 with downregulation of the proto-oncogene BCL6 by chromatin-modifying drugs in human cancer cells. Cancer Cell, 2006,9:435-443.
[3] Datta J, Kutay H, Nasser MW, et al.Methylation mediated silencing of MicroRNA-1 gene and its role in hepatocellular carcinogenesis. Cancer Res,2008,68:5049-5058.
[4] Toyota M, Suzuki H, Sasaki Y, et al.Epigenetic silencing of microRNA-34b/c and B-cell translocation gene 4 is associated with CpG island methylation in colorectal cancer. Cancer Res,2008,68:4123-4132.
[5] Grady WM, Parkin RK, Mitchell PS, et al.Epigenetic silencing of the intronic microRNA hsa-miR-342 and its host gene EVL in colorectal cancer. Oncogene, 2008,27:3880-3888.
[6] Lehmann U, Hasemeier B, Christgen M, et al.Epigenetic inactivation of microRNA gene hsa-mir-9-1 in human breast cancer. J Pathol,2008,214:17-24.
[7] Meng F, Wehbe-Janek H, Henson R, et al.Epigenetic regulation of microRNA-370 by interleukin-6 in malignant human cholangiocytes. Oncogene,2008,27:378-386.
[8] Herman JG, Graff JR, Myöhänen S, et al. Methylation specific PCR: A novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci USA,1996,93:9821-9826.
[9] Tost J, Gut IG. DNA methylation analysis by pyrosequencing.Nat Protoc, 2007,2:2265-2275.
[10] Chen HC, Chen GH, Chen YH, et al. MicroRNA deregulation and pathway alterations in nasopharyngeal carcinoma. Br J Cancer,2009,100:1002-1011.
[11] Tam W, Dzhlberg JE.miR-155/BIC as an oncogenic microRNA.Genes Chromosomes Cancer,2006,45:211-212.
[12] Hiyoshi Y, Kamohara H, Karashima R, et al. MicroRNA-21 regulates the proliferation and invasion in esophageal squamous cell carcinoma.Clin Cancer Res,2009,15:1915-1922.
[13] Liu X, Sempere LF, Galimberti F, et al. Uncovering growth-suppressive MicroRNAs in lung cancer. Clin Cancer Res,2009,15:1177-1183.
[14] Yamakuchi M, Lowenstein CJ. MiR-34, SIRT1 and p53: the feedback loop. Cell Cycle,2009,8:712-715.
[15] Lu J, Getz G, Miska EA, et al. MicroRNA expression profiles classify human cancers. Nature,2005,435:834-838.
[16] Rabinowits G, Gerçel-Taylor C, Day JM, et al. Exosomal microRNA: a diagnostic marker for lung cancer.Clin Lung Cancer,2009,10:42-46.
AbnormalmethylationofmiRNAinpancreaticcancercelllinePANC1
PENGQuan,ZHANGLi-jie,CAIHui-hua,GAOWen-tao,ZHAOCheng-gong,QIANZhu-yin,MIAOYi.
DepartmentofGeneralSurgery,The105thCentralHospitalofPeople′sLiberationArmy,Hefei230032,China
QIANZhu-yin,Email:qianzhusilver@163.com
ObjectiveTo investigate the methylation of the promoter region in miRNA in pancreatic cancer cell line PANC1 and normal pancreatic tissue, to discover the miRNA with hypermethylation associated with pancreatic cancer.MethodsThe genomic DNA of PANC1 and normal pancreatic tissue was extracted, and fractured by ultrasound. Methylation DNA fragments were obtained by 5-methyl of pyrimidine nucleoside antibodies and immunomagnetic beads. The hypermethylation miRNA differentially expressed between PANC1 and normal pancreatic tissue was selected by using methylation DNA chip. BSP (bisulfite genomic sequencing PCR) and TA clone sequencing was performed for further validation. The genomic DNA of pancreatic cancer cell lines BXPC3, CFPAC1, PANC1 and SW1990 was extracted. The COBRA (combined bisulfite restriction analysis) was used to validate differentially expressed hypermethylation miRNA.ResultsEight differentially expressed hypermethylation miRNAs were screened from the DNA methylation chips, then five of them were selected for sequencing. The methylation status of miRNA-615,-663,-663b was significantly higher in the PANC1 than in normal tissues (60.6%vs7.6%, 88.8%vs22.2%, 94.4%vs13.0%); the methylation status of miRNA-675 was not significantly different between PANC1 and normal pancreatic tissue (76.0%vs100%). Due to large error in sequencing, miRNA1826 was excluded. The results of COBRA confirmed all the 4 miRNAs were highly methylated in PANC1; except for miRNA-675, other 3 miRNAs were highly methylated in BxPC,miRNA-663, miRNA-663b were highly methylated in CFPAC1, while miRNA-615, miRNA-663 were highly methylated in SW1990.ConclusionsHypermethylation miRNAs were differentially expressed between pancreatic cancer cell lines and normal pancreatic tissue, among them, highly methylated miRNA-663 was possibly associated with pancreatic cancer.
Pancreatic neoplasms; DNA methylation; miRNA; TA cloning
10.3760/cma.j.issn.1674-1935.2012.01.004
江苏省自然科学基金(BK2006241);人事部留学回国人员基因(303070460IA10)
230032 安徽合肥,解放军第105医院普外科(彭泉、赵成功),肿瘤科(张立洁);南京医科大学第一附属医院普外科(蔡辉华、高文涛、钱祝银、苗毅)
钱祝银,Email:qianzhusilver@163.com
2011-04-06)
(本文编辑:屠振兴)
