牛筋草ISSR-PCR反应体系的建立与优化
2012-09-11张朝贤王金信魏守辉黄红娟
张 杨, 张朝贤, 王金信*, 魏守辉, 黄红娟*
(1.山东农业大学植物保护学院,泰安 271018;2.中国农业科学院植物保护研究所,中国农业科学院杂草鼠害生物学与治理重点开放实验室,北京 100193)
牛筋草[Eleusine indica(Linn.)Gaertn.]俗称油葫芦草、蟋蟀草、千人踏等,属禾本科属一年生草本植物,生长于较湿润的农田或路旁,广布全国各地。对棉花、玉米、大豆、花生等作物危害较重,为近年来较难防除的杂草之一,灭生性除草剂草甘膦防除效果不理想。目前,有关牛筋草的研究主要集中在其对除草剂产生抗性方面[1-2],针对其遗传多样性的研究未见报道。
简单重复序列区间扩增(inter-simple sequence repeat,ISSR)是 Zietkiewicz等[3]于1994年在微卫星技术基础上发展起来的分子标记技术。ISSR技术是一种显性标记技术,结合了SSR和RAPD的优点[3-4],克 服 了 RFLP 和 RAPD 的 限 制[4-5],具 有 引物序列较长、退火温度较高、重复性较好、稳定性高、多态性高[6]、成本低、操作简单、所需DNA模板量少等特点,因而备受青睐。但ISSR是基于PCR的一种分子标记技术,受多种因素的影响,而且反应因子所需条件不合适会导致扩增产物消失,图谱弥散等,因此在进行ISSR分子标记时应首先对其反应条件进行优化[7-8]。
目前,许多领域都已利用ISSR技术进行科学研究,例如种质资源鉴定、遗传图谱构建、植物进化和遗传多样性分析等领域。国内将ISSR技术用于杂草相关基因标记的研究报道较少[9-12],也未见应用于牛筋草遗传多样性研究的报道。本文通过研究影响牛筋草ISSR反应体系的相关因素、建立与优化牛筋草ISSR-PCR反应体系,为ISSR标记在杂草遗传多样性的研究方面提供技术依据。
1 材料与方法
1.1 材料
供试材料:牛筋草,其种子于2009年采自安徽阜阳路旁。将饱满的牛筋草种子播在高9.2cm,直径10.5cm的花盆中。培养土壤为黏土,pH 6.8,有机质含量在1.9%左右,土壤与草木炭按体积3∶1混合使用。室内白天温度为(28±5)℃,晚上温度为(23±5)℃,每天光照11h,相对湿度为(75±5)%,正常水肥管理。取3~4叶期幼苗供试。
供试引物、试剂及仪器设备:用于ISSR-PCR反应的试剂,dNTP、Taq DNA 聚合酶、Mg2+、10×PCR Buffer,均购自宝生物(TaKaRa)工程公司。所用引物参照哥伦比亚大学(University of Columbia)公布的ISSR引物序列,由上海生物工程技术有限公司合成,试验所用仪器设备均由中国农业科学院杂草鼠害生物学与治理重点开放实验室提供。
1.2 方法
1.2.1 基因组DNA提取
采用新型快速植物基因组DNA提取试剂盒(北京百泰克生物技术有限公司,离心柱型)提取新鲜牛筋草地上组织DNA,方法按照说明书进行。经l%琼脂糖凝胶电泳分析,核酸蛋白快速检测仪(Eppendorf Bio Photometer)测定其在260nm和280nm处的吸光值,计算DNA纯度,稀释至30ng/μL并保存在-20℃冰箱中备用。
1.2.2 PCR正交试验设计与单因素处理
采用L16(45)正交试验设计[13],对ISSR-PCR的主要影响因素Taq DNA 聚合酶、dNTP、引物、Mg2+浓度及模板DNA浓度进行5因素4水平筛选分析,共16个处理,每处理3次重复,ISSR-PCR反应各因素水平及正交试验见表1。反应体系总体积为25μL,包括10×PCR Buffer 2.5μL,其他各成分按照表1加样,不足体积用ddH2O补足。

表1 PCR反应因素水平L16(45)正交试验设计表
单因素试验在正交试验最优体系的基础上,将单个因子按照一定梯度变化,其他因子保持不变,逐个筛选出各因子的最优扩增条件,组成适合于牛筋草的ISSR-PCR最佳反应体系。各因子浓度梯度处理见表2。
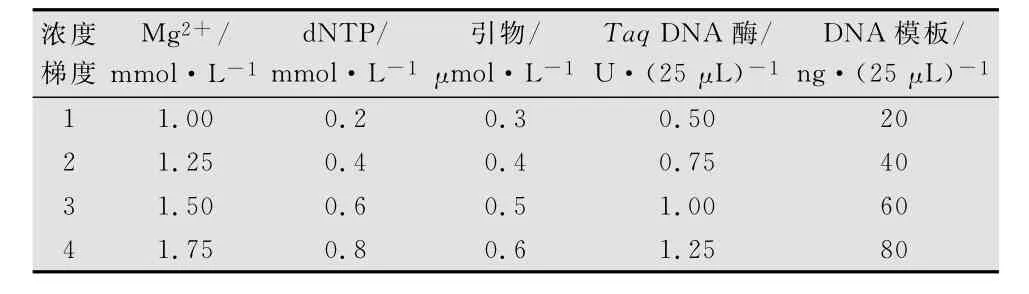
表2 各因子浓度梯度
1.2.3 PCR扩增及电泳检测
PCR扩增程序为94℃预变性5min;94℃变性30s,52 ℃退火30s,72 ℃延伸1min,35个循环;72℃延伸10min;4℃保存备用。扩增产物采用1.8%琼脂糖凝胶电泳,l×TAE缓冲液,电压120V,电泳1.5h,于UVP凝胶成像系统上拍照保存,并对结果进行直观分析,确定牛筋草ISSR-PCR反应各影响因素的最佳水平。
2 结果与分析
2.1 正交试验结果
引物UBC835对安徽阜阳牛筋草扩增试验结果见图1。谱带较为丰富清晰的为组合13、14、15和16,组合5、12条带模糊,组合2、3、8、9和11谱带较弱主带不明显且出现杂带,组合1、4、7和10多态性条带少,而组合6无条带。比较分析发现,组合14谱带清晰,且多态性较高,确定为最优组合,即5μL的PCR反应体系中含:0.4mmol/L dNTP、0.3μmol/L ISSR引物、1.75mmol/L Mg2+、1UTaq DNA 聚合酶、2.5μL 10×PCR Buffer和80ng DNA模板。
为获得理想的ISSR-PCR体系,在组合14的基础上,其他因素保持不变的情况下进行第2轮的单因素优化筛选试验。
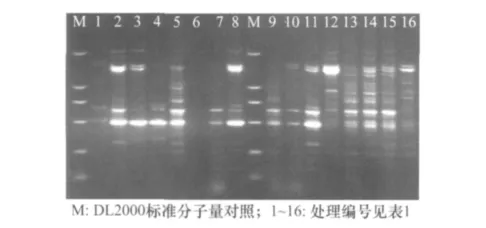
图1 ISSR-PCR正交设计扩增结果
2.2 退火温度对ISSR-PCR扩增效果的影响
在正交试验最佳反应体系基础上,对退火温度进行梯度试验以确定引物合适的退火温度。根据所选引物序列计算理论退火温度Tm,Tm=4(G+C)+2(A+T)[14],在 梯 度 PCR 仪 上 设 定 退 火 温 度(Tm±5)℃,仪器自动形成12个温度,在其中的10个温度上进行扩增,其他反应程序与PCR正交试验相同。扩增结果显示(图2),退火温度过低情况下,扩增特异性差,而退火温度过高又会导致引物与模板结合差,出现电泳条带减弱的现象。为提高反应的特异性,扩增结果相近时,优先选择相对较高的退火温度,图2中泳道7、8条带清晰明亮,因此确定引物UBC835的退火温度为52℃。
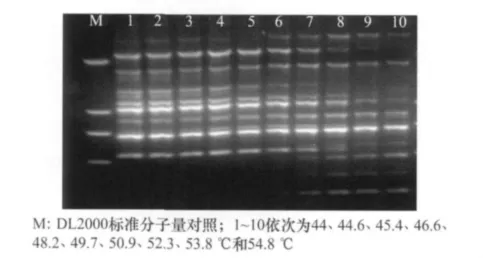
图2 退火温度对ISSR-PCR扩增效果的影响(引物为UBC835)
2.3 Taq DNA聚合酶用量对ISSR-PCR扩增效果的影响
Taq DNA聚合酶的用量是影响PCR的一个重要因素,TaqDNA聚合酶浓度过低导致PCR扩增产物的合成效率降低。Taq DNA 聚合酶为0.5、0.75、1.00U和1.25U4个用量下(图3),扩增结果不一致。当Taq酶用量0.5U和1.25U时合成效率低,条带少且不清晰;用量为0.75U和1.00U时,所得扩增条带清晰,结合正交试验结果确定Taq酶用量1.00U。
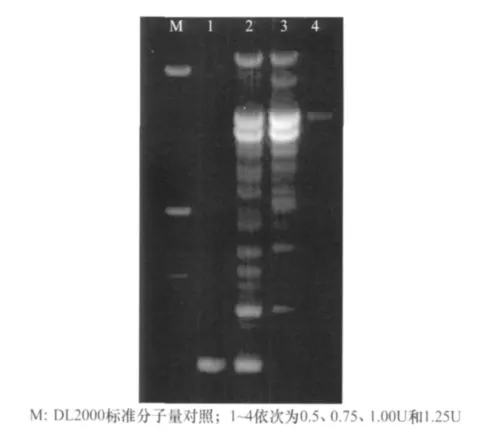
图3 TaqDNA聚合酶浓度对ISSR-PCR扩增效果的影响(引物为UBC835)
2.4 Mg2+浓度对ISSR-PCR扩增效果的影响
Mg2+浓度也是影响PCR结果的重要因素之一,它是TaqDNA聚合酶的激活剂,是Taq DNA聚合酶活性重要的影响因素之一。Mg2+与反应液中的引物、dNTP及模板相结合,影响扩增产物的特异性、引物与模板的结合效率以及引物二聚体的形成[15]。从扩增结果(图4)中可以看出,Mg2+浓度在1.50mmol/L和1.75mmol/L时,所得扩增结果无显著差异,而在正交试验结果中1.75mmol/L Mg2+浓度扩增结果优于1.50mmol/L,因此确定Mg2+最佳浓度为1.75mmol/L。
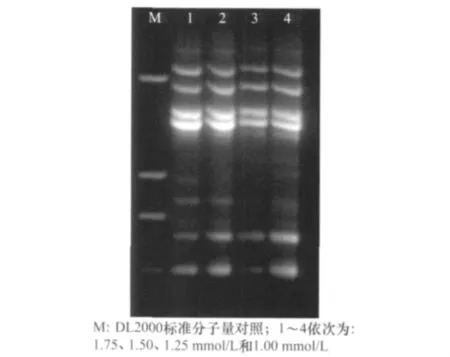
图4 Mg2+浓度对ISSR-PCR扩增效果的影响(引物为UBC835)
2.5 dNTP浓度对ISSR-PCR扩增效果的影响
dNTP是PCR的原料,过高浓度的dNTP会造成错误掺入,而浓度太低又会导致扩增产率下降。从图5中可以看出,dNTP浓度为0.8mmol/L时扩增条带弱,多态性差;浓度为0.2mmol/L和0.4mmol/L时扩增所得电泳条带清晰,多态性相对较好。结合正交试验结果,选择0.4mmol/L作为ISSR-PCR反应体系中dNTP的最佳浓度。

图5 dNTP浓度对ISSR-PCR扩增效果的影响(引物为UBC835)
2.6 引物浓度对ISSR-PCR的影响
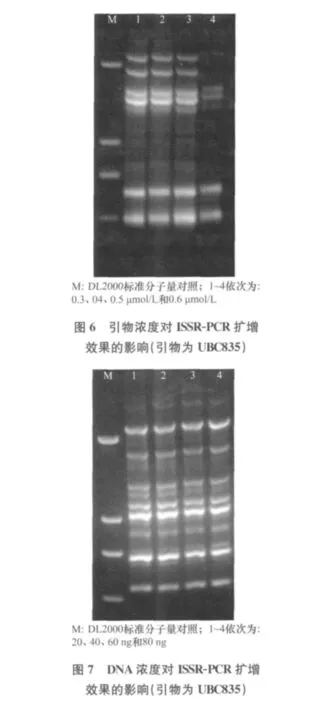
引物浓度与扩增产物质量相关,浓度偏高产生错配和非特异性扩增产物几率高,引物二聚体的形成几率大;浓度太低导致有效扩增条带减少。图6中各引物浓度扩增产生的电泳条带一致,当浓度为0.6μmol/L时条带亮度降低,浓度为0.3、0.4、0.5μmol/L时,反应稳定,条带清晰,基本一致,扩增效果无显著差别,综合考虑正交试验结果,选择0.3μmol/L作为ISSR-PCR反应体系中引物的最佳浓度。
2.7 模板DNA浓度对ISSR-PCR扩增效果的影响
DNA模板浓度范围由所研究的物种、类群以及DNA模板纯度所决定。浓度过低致使扩增产物不稳定,过高则可能出现非特异性扩增[16],但在一定浓度范围内ISSR反应对模板含量不甚敏感。大量的研究表明ISSR-PCR对DNA模板的要求并不严格,浓度对PCR反应体系的影响,不同的试材获得的数据不尽相同[17-21]。本研究结果表明,DNA浓度在20~80ng范围内对扩增结果的一致性无明显影响(图7),因此选定L16(45)设计中优势组合14组中DNA浓度80ng。
3 讨论
在PCR反应体系中,Taq酶、Mg2+、dNTP和引物是4个主要的影响因子。物种不同,试验条件不同,各因子之间相互影响产生的扩增结果也不同。在本研究中不同处理间存在差异,因此需要对这些因子的浓度进行优化,选取最佳组合。Taq酶用量是影响试验结果的重要因素之一,Taq酶浓度高不仅容易产生非特异性扩增产物,而且试验成本高;Taq酶浓度过低则容易造成新链的合成效率下降。TaqDNA聚合酶还是Mg2+的依赖性酶,对Mg2+浓度非常敏感。Mg2+浓度也是影响PCR结果的重要变量之一,Mg2+浓度能影响反应的特异性和扩增片段的产率。本试验在Mg2+浓度范围内低浓度下的扩增效果差于高浓度。底物dNTP浓度过高,造成延伸时核苷酸错误掺入,引起错配。浓度过低,不仅会影响扩增效率,而且会因dNTP过早消耗而使产物单链化,降低了PCR产物的产量。引物浓度影响扩增产物的质量,浓度偏高会引起错配和非特异性扩增,浓度太低则无法进行有效扩增。
另外,循环次数、退火温度以及延伸时间的确定也是反应体系建立与优化的重要内容。但在本试验中,上述3种因子对扩增结果的影响不明显,可能与引物的特异性较强以及本试验建立的牛筋草ISSRPCR扩增体系比较稳定有关,导致反应体系对反应条件的变化具有较强的承受能力。延伸时间的长短和扩增片段的长度有关,两者呈正相关。对于低浓度模板的扩增,则需要稍延长延伸时间,但延伸时间过长会导致非特异性扩增带的出现。本试验延伸时间为1min时,产物稳定且带型丰富适合牛筋草ISSR-PCR体系。
目前主要有单因素水平优化试验和正交优化设计试验两种方法应用于ISSR-PCR体系的建立和优化。单因子试验主要针对每个影响因子的不同水平进行分别优化,多被应用于PCR反应体系中各因子情况都有一定了解的体系;正交优化设计试验主要针对各因子影响程度未知的情况,利用正交设计,综合考查各种因素及其交互作用,得出体系反应中各影响因子的最佳组合。本研究同时采用以上两种方法,综合优化分析,增加了试验结果的可靠性,为后续牛筋草的遗传多样性分析奠定了技术和理论基础。
正交设计采用直观分析,有的研究还采用方差分析法对试验结果进行分析[22],但这仍不可避免由主观打分造成试验误差。且PCR受制约因素比较多,加之ISSR大部分情况下是显性标记,不能区分纯合体与杂合体[23],因此尽量在相同的条件下进行扩增反应是使试验成功的决定因素。建立客观的ISSR-PCR扩增结果评价标准有助于该方法更好地应用。
[1] Powles S B,Preston C.Evolved glyphosate resistance in plants:biochemical and genetic basis of resistance[J].Weed Technology,2006,20:282-289.
[2] Baerson M S,Brinker R,Casagrande L,et al.Characterization of glyphosate resistant eleusine indica biotypes from Malaysia[C]∥Proceedings(B)17th Asian-pacific Weed Science Society Conference,Thailand:Bangkok,1999:527-536.
[3] 周延清.DNA分子标记技术在植物研究中的应用[M].北京:化学工业出版社,2005:146-161.
[4] Bornet B,Branchard M.Nonanchored inter simple sequence repeat(ISSR)markers:reproducible and specific tools for genome fingerprinting[J].Plant Molecular Biology Reporter,2001,19:209-210.
[5] Zietkiewicz E,Rafalski A,Labuda D.Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20:176-183.
[6] Nagaoka T,Ogihara Y.Applicability of inter-simple sequence repeat polymorphisms in wheat for use as DNA markers in comparison to RFLP and RAPD markers[J].Theoretical and Applied Genetics,1997,94:597-602.
[7] 刘海河,侯喜林,张彦萍.西瓜ISSR-PCR体系的正交优化研究[J].果树学报,2004,21(6):615-617.
[8] 桂腾琴,乔爱民,孙敏,等.果梅ISSR-PCR反应体系的建立和优化[J].西南大学学报(自然科学版),2007,29(10):124-125.
[9] 马森.加拿大一枝黄花的入侵生物学研究[D].上海:复旦大学,2003.
[10]张玉琴.外来入侵植物银胶菊遗传多样性的ISSR分析[D].南宁:广西师范大学,2007.
[11]桂富荣.紫茎泽兰的遗传多样性及其种群结构分析[D].北京:中国农业科学院,2006.
[12]刘美.ISSR标记的早熟禾遗传多样性分析[D].兰州:甘肃农业大学,2009.
[13]盖钧镒.试验统计方法[M].北京:中国农业出版社,2000:286-287.
[14]卢圣栋.现代分子生物学实验技术[M].第2版.北京:中国协和医科大学出版社,1999:57-58.
[15]席嘉宾,郑玉忠,杨中艺.地毯草ISSR反应体系的建立与优化[J].中山大学学报:自然科学版,2004,43(3):80-84.
[16]欧文军,李开绵,王文泉,等.小桐子基因组DNA的提取及ISSR-PCR反应体系的优化[J].中国农学通报,2008,24(5):409-413.
[17]王桂娟,陈茂盛,向振勇.小桐子ISSR-PCR体系的优化[J].生物技术通报,2008(3):162-165.
[18]李海生.ISSR分子标记技术及其在植物遗传多样性分析中的应用[J].生物学通报,2004,39(2):19-20.
[19]潘坤,王文泉,吴翼,等.椰子ISSR体系优化[J].中国农学通报,2009,25(4):24-29.
[20]姜静,杨传平,刘桂丰,等.桦树ISSR-PCR反应体系的优化[J].生态学杂志,2003,22(3):91-93.
[21]曾黎辉,洪自同,许家辉,等.龙眼ISSR反应体系的建立和优化[J].中国农学通报,2007,23(9):111-113.
[22]谢运海,夏德安,姜静,等.利用正交设计优化水曲柳ISSRPCR反应体系[J].分子植物育种,2005,3(3):445-450.
[23]Sánchez de la Hoz M P,Dávila J A,Loarce Y,et al.Simple sequence repeat primers used in polymerase chain reaction amplifications to study genetic diversity in barley[J].Genome,1996,39:112-117.
