正交设计优化山野豌豆SRAP-PCR反应体系与引物筛选
2012-08-20刘颖王显国张巨明刘芳
刘颖,王显国,张巨明*,刘芳
(1.华南农业大学农学院,广东 广州510642;2.中国农业大学动物科技学院,北京100193;3.全国畜牧总站,北京100193)
山野豌豆(Vicia amoena)又叫落豆秧、高蔓草藤、宿根苕子,为豆科巢菜属多年生草本植物[1]。它表现出耐寒、抗旱、叶量大、粗蛋白及动物必需氨基酸含量高、适应性广、再生力强、容易繁殖、产量高、品质好等优点[2-5]。另外,山野豌豆不仅是一种优良牧草,而且还是很好的防风固沙水保植物[6]、改土肥田绿肥作物[7]和草坪及药用植物[8],具有推广和应用价值。目前SRAP(sequence related amplified polymorphism)标记已在农作物和园艺作物上有了较多成功的应用,但SRAP在豆科牧草种质资源上的应用还非常少,仅在苜蓿(Medicago)[9-15]、大豆(Glycine)[16-18]、三叶草(Trifolium)[19,20]、山羊豆(Galege)[21]、小扁豆(Lens)[22,23]等属有相关报道。本研究主要采用正交设计法对SRAP-PCR反应条件中主要因子:Mg2+、dNTP、引物、Taq酶进行优化分析并确定了模板DNA浓度,旨在筛选出一个适用于野豌豆属的稳定性及重复性好,多态性高的最佳反应条件。此外,本研究还对部分SRAP引物组合进行了多态性筛选,希望获得一些多态性丰富的SRAP引物组合,为开展SRAP标记技术在山野豌豆种质鉴定、遗传多样性等方面的分子生物学研究提供有利依据。
1 材料与方法
1.1 供试材料
试验所用5份山野豌豆材料采自全国3个不同的地域,优化SRAP-PCR体系的DNA模板来自内蒙古呼伦贝尔的9号、山西五台山的6号及北京百花山的14号DNA;筛选引物所用DNA模板来自山西沁源的11号、北京灵山的5号及内蒙古呼伦贝尔的9号DNA。
1.2 试验设计与方法
1.2.1 PCR反应体系设计与引物组合 采用L9(34)正交试验设计,对 Mg2+、dNTP、引物、Taq DNA 聚合酶进行4因素3水平筛选(表1)。体系优化分别采用3份不同DNA进行,每份DNA做2次重复,取其平均值作为统计数据。体系总体积为25μL,除表中变化的因素外,每管中还加入2μL 10×PCR buffer和50ng的模板DNA,所用引物组合为Me2+Em4。
1.2.2 SRAP-PCR扩增程序及扩增产物的检测SRAP-PCR扩增程序为:94℃预变性4min;94℃变性1min,37℃退火45s,72℃延伸1min,5个循环;94℃变性1min,50℃退火45s,72℃延伸1min,35个循环;循环结束后,72℃延伸7min,4℃保存。扩增结束后,在扩增产物中加入5μL 6×loading Buffer缓冲液混匀,取8μL上样于2%的琼脂糖凝胶中进行分离。
1.2.3 模板DNA浓度优化 应用上述体系最佳组合,引物组合为 Me2+Em4,DNA模板为5号DNA。将25μL扩增体系中模板DNA用量分别设置6个梯度处理,即10,20,30,40,50和60ng,对反应体系中的模板DNA浓度进行优化。
1.2.4 多态性SRAP引物组合的筛选 根据上述试验结果确定的SRAP-PCR最佳反应体系,选取Li和Quiros[24]及Riaz等[25]发表的引物序列,包括7条正向引物和14条反向引物两两组合成98个SRAP引物组合(表2),对3份山野豌豆种源材料进行多态性SRAP引物组合的筛选。
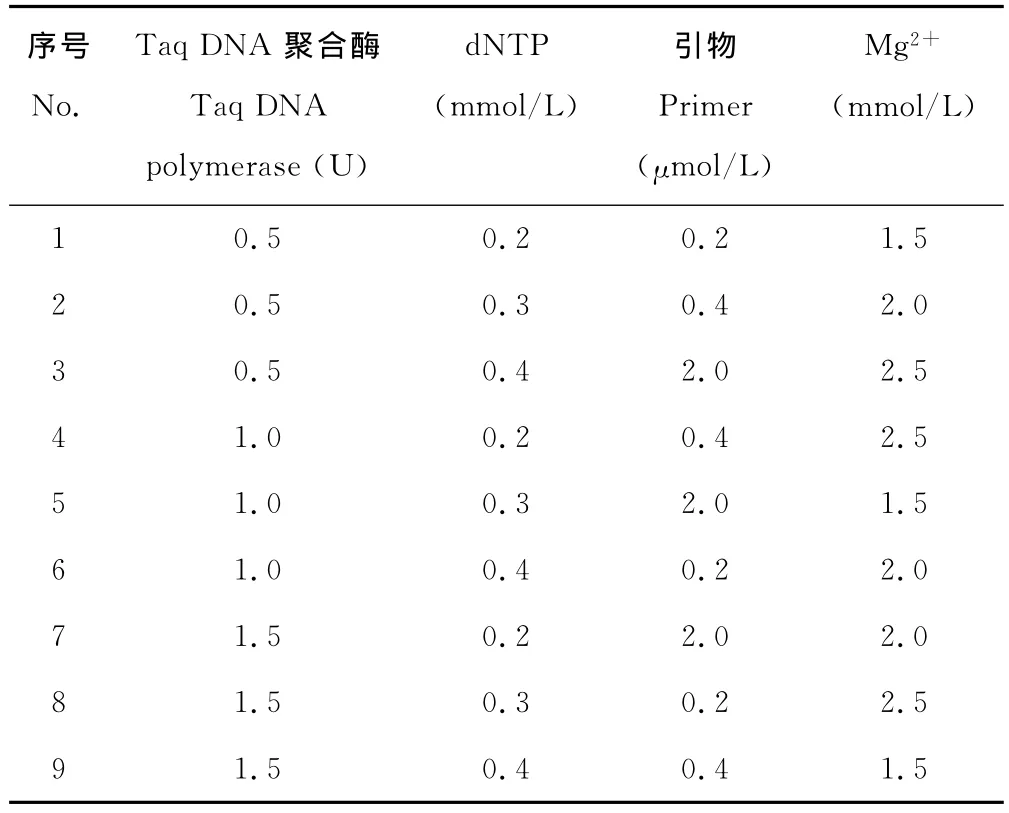
表1 SRAP-PCR[L9(34)]正交试验设计Table 1 Orthogonal design for SRAP-PCR [L9(34)]
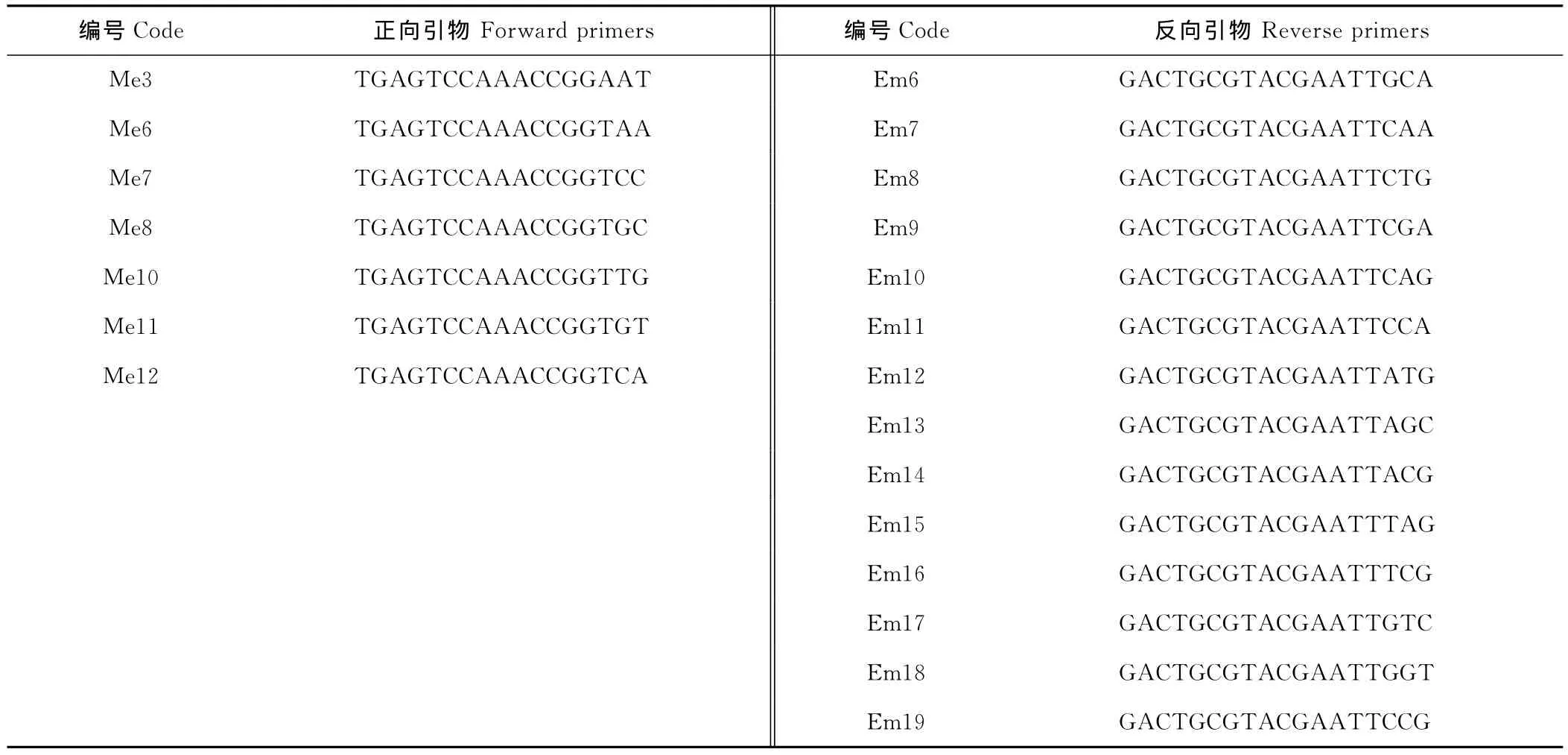
表2 SRAP引物序列Table 2 Primer sequences used for SRAP analysis
2 结果与分析
2.1 SRAP-PCR正交试验设计的扩增结果分析
山野豌豆SRAP-PCR体系优化的正交试验结果如图1所示。参照何正文等[26]的方法,对扩增结果进行直观分析,依据谱带的亮度、清晰度以及条带数,将9个处理结果划分为9个评分等级以便统计分析,内蒙古呼伦贝尔的DNA扩增结果得分为1,3,2,5,6,7,9,4和8。山西五台山与北京百花山的DNA扩增结果得分分别为1,6,7,8,5,2,9,3和4及6,7,4,3,5,1,9,2和8。3份不同种源的9个处理结果的总平均得分为2.67,5.33,4.33,5.33,5.33,3.33,9.00,2.67,6.67。得分最高为组合7带型清晰,亮度高,且杂带少,扩增效果最好,选为山野豌豆基因组SRAP-PCR的最佳扩增反应体系。
参照张丽等[27]的方法,对各组分浓度组合的正交试验结果进行了统计分析(表3),其中T值代表某水平下某因子参与反应所产生的扩增条带的总和;K代表某因子在某水平参与反应所产生的扩增条带的平均值;R为某因子的极差,即某因子在不同水平下最大平均值与最小平均值之差。R值的大小反映了该因子对试验结果影响的大小,R值越大,影响越显著。从R值可看出,在选定的3个水平范围内,4个因素对结果的影响由大到小依次为:引物> Mg2+>Taq DNA聚合酶>dNTP。K值反映了影响因素各水平对反应体系的影响情况,K值越大,反应水平越好。从K值结果来看,引物以水平3最好,Mg2+以水平2最好,Taq DNA聚合酶水平3最好,dNTP以水平1最好。综上,各因素最佳的水平具体为引物2.0μmol/L、Mg2+2.0mmol/L、Taq DNA 聚合酶1.5U、dNTP 0.2mmol/L,该组合与直观分析得出的最佳组合7一致,进一步确定组合7为最优组合。
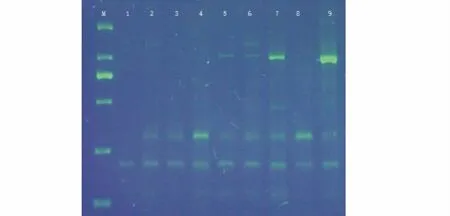
图1 SRAP-PCR正交试验设计结果Fig.1 Electrophoresis of SRAP-PCR orthogonal design
2.2 模板DNA浓度的确定
应用上述最佳反应体系,对模板DNA设置了10,20,30,40,50和60ng 6个浓度梯度进行优化。扩增结果表明,在25μL反应体系中6种模板DNA用量均能扩增出谱带,且带型一致。但模板DNA浓度不同,谱带的清晰度存在一定差异。模板DNA用量为10和20ng时条带清晰度不佳,30~60ng条带清晰度稍强且表现一致。考虑到节省模板用量,最终选定在25μL反应体系中加30ng模板DNA为宜。
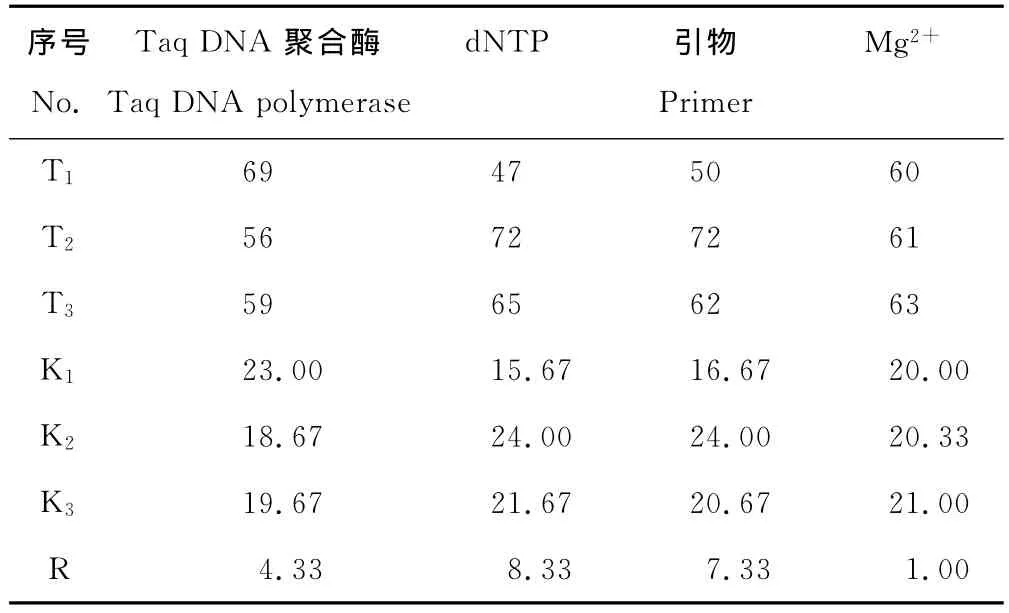
表3 正交试验结果的统计分析Table 3 Statistic result of the orthogonal design
2.3 反应体系的验证及多态性引物组合的筛选
应用上述最佳反应体系,随机组合了98对SRAP引物组合对3份山野豌豆种源进行了SRAP扩增(图2)。结果表明,1)在筛选的98对引物组合中,94对引物组合扩增得到清晰稳定的谱带,占总数的95.9%,表明该反应体系具有较好的稳定性;在有扩增产物的94对引物组合中有20对引物组合扩增出多态性条带,占总数21.3%。2)98对引物共扩增出983条带,其中20对具有多态性的引物组合扩增出的条带总数为164条,占总条带数的16.7%,具有多态性的条带有30条,平均每对引物组合扩增出1.5条。3)不同引物组合检测到的多态性位点数目存在差异,变异幅度为1~7。
3 讨论与结论
3.1 正交试验设计优化SRAP-PCR反应体系
PCR扩增体系主要影响因素包括 Mg2+、dNTP、引物、Taq DNA聚合酶以及模板DNA。本研究优化出的体系表明引物浓度对扩增结果影响最大,这与黄春琼等[28]、周良彬等[29]的结果不一致,这主要是因为本研究在引物梯度设计时加入了以前文献中不常见到的高浓度——2.0μmol/L,仅在昝逢刚等[30]对荔枝(Litchichinensis)和许永华等[31]对人参(Panaxginseng)的研究中分别出现过5 mmol/L和2.0μmol/L的高引物浓度。虽说文献中常见引物浓度要以最低引物量产生所需要的结果为好,引物浓度偏高会引起错配和非特异性扩增,但本研究结果表明高浓度的引物浓度不但没有引起错配反而得到更清晰的条带,从图1可看到7道与9道均得到1kb处的条带,而9道的引物浓度仅为0.2μmol/L,这说明此带并非非特异性条带。此后以6号与14号为DNA模板进行的重复试验,也证实了2.0μmol/L的引物浓度并无非特异性扩增条带,反而条带清晰这一论断。其次,影响较大的是Mg2+浓度。这与王芳[18]、王俊娥[21]、艾鹏飞等[32]、仲丛来等[33]在大豆、山羊豆、小麦(Triticumaestivum)和麻疯树(Jatrophacurcas)上的试验结果一致,这说明在SRAP-PCR反应体系中Mg2+浓度的影响是很大的,在以后的优化试验中要重点关注。再次,影响因子较小的是Taq DNA聚合酶用量和dNTP浓度,其中不同dNTP浓度的扩增效果差异很小。在正交试验之后,本研究又对DNA模板用量进行了单因素优化。虽然DNA模板用量对扩增的影响不大[34,35],但本研究证实用量过少会明显影响条带的数量与清晰度,DNA模板用量为10~20ng明显没有30~60ng的条带清晰度高,所以本实验选择30ng的DNA模板用量,此结果与孙佩光等[36]、陈庆榆等[37]的研究结果一致。

图2 优化的SRAP-PCR反应体系在部分引物组合中的扩增结果Fig.2 Results amplified by optimized system using some primer combinations
3.2 SRAP-PCR反应优化体系的验证
本研究不仅通过98对引物对优化出的SRAP-PCR反应体系进行了验证,而且在优化过程中选用了3种来自北京、山西、内蒙古不同地域的模板DNA取得了二次验证,以此保证了优化体系的稳定性与良好的适用性。与此同时,本研究亦筛选出20对多态性良好的引物,对以后山野豌豆的分子生物学研究打下了良好的基础。
3.3 结论
本研究利用正交试验设计对山野豌豆SRAP-PCR反应体系进行优化,得到的最佳反应体系为:2μL 10×PCR buffer(不含 Mg2+)、30ng的模板 DNA、引物2.0μmol/L、Mg2+2.0mmol/L、Taq DNA 聚合酶1.5U、dNTP 0.2mmol/L,总体积为25μL。
[1] 陈默君,贾慎修.中国饲用植物[M].北京:中国农业出版社,2002.
[2] 陈默君.五种野豌豆饲用价值的初步探讨[J].中国草地学报,1986,3:43-46.
[3] 马章全,冯忠义.三个适合农牧区种植的优质牧草品种[J].农村百事通,2008,(19):40-41.
[4] 郜玉田,张雪艳.有发展前途的新草种——山野豌豆[J].吉林畜牧兽医,1991,(2):39-40.
[5] 洪锐民,肖文一.黑龙江省野豌豆属野生牧草的特征、习性及其利用[J].黑龙江畜牧兽医,1992,(7):14-15.
[6] 吴学敏.一种优良野生牧草——山野豌豆[J].山西水土保持科技,1983,(3):93.
[7] 王鹤桥.野生牧草绿肥植物——山野豌豆[J].中国草原与牧草,1986,3(6):49.
[8] 杨巧荷,罗素琴,刘乐乐,等.蒙药材山野豌豆中的化学成分定性分析[J].内蒙古医学院学报,2010,32(5):475-477.
[9] 周良彬,卢欣石,王铁梅,等.杂花苜蓿种质SRAP标记遗传多样性研究[J].草地学报,2010,18(4):544-549.
[10] 何庆元,吴萍,张晓红,等.不同秋眠性苜蓿SRAP体系优化及遗传多样性分析[J].草业学报,2011,20(2):201-209.
[11] 张宇.利用SRAP分子标记研究苜蓿杂种优势[D].呼和浩特:内蒙古农业大学,2010.
[12] Ariss J J,Vandemark G J.Assessment of genetic diversity among nondormant and semidormant alfalfa populations using sequence-related amplified polymorphisms[J].Crop Science,2007,47(6):2274-2284.
[13] Vandemark G J,Ariss J J,Bauchan G A,et al.Estimating genetic relationships among historical sources of alfalfa germplasm and selected cultivars with sequence related amplified polymorphisms[J].Euphytica,2006,152(1):9-16.
[14] Castonguay Y,Cloutier J,Bertrand A,et al.SRAP polymorphisms associated with superior freezing tolerance in alfalfa(Medicago sativa spp.sativa)[J].Theoretical and Applied Genetics,2010,120(8):1611-1619.
[15] 关潇.野生紫花苜蓿种质资源遗传多样性研究[D].北京:北京林业大学,2009.
[16] Baloch F S,Kurt C,Arioglu H.Assaying of diversity among soybean(Glycin max (L.)Merr.)and peanut(Arachis hypogaea L.)genotypes at DNA level[J].Turkish Journal of Agriculture and Forestry,2010,34(4):285-301.
[17] 王贤智.大豆产量相关性状的遗传与稳定性分析及QTL定位研究[D].武汉:中国农业科学院,2008.
[18] 王芳.大豆的SRAP遗传多样性及蛋白含量多样性研究[D].新乡:河南师范大学,2009.
[19] 张婧源,彭燕,罗燕,等.不同产地白三叶种质遗传多样性的SRAP分析[J].草业学报,2010,19(5):130-138.
[20] 李润芳.三叶草的细胞、分子标记及多倍体诱导研究[D].武汉:华中农业大学,2007.
[21] 王俊娥.山羊豆属植物遗传多样性研究[D].北京:中国农业科学院北京畜牧兽医研究所,2008.
[22] Saha G C,Sarker A,Chen W D,et al.Identification of markers associated with genes for rust resistance in Lens culinaris Medik[J].Euphytica,2010,175(2):261-265.
[23] Saha G C,Sarker A,Chen W D.Inheritance and linkage map positions of genes conferring resistance to stemphylium blight in lentil[J].Crop Science,2010,50(5):1831-1839.
[24] Li G,Quiros C F.Sequence related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J].Theoretical and Applied Genetics,2001,103(2-3):455-461.
[25] Riaz A,Li G,Quresh Z,et al.Genetic diversity of oil seed Brassica napus inbred lines based on sequence-related amplified polymorphism and its relation to hybrid performance[J].Plant Breeding,2001,120(5):411-415.
[26] 何正文,刘运生,陈立华,等.正交设计直观分析法优化PCR条件[J].湖南医科大学学报,1998,23(4):403-404.
[27] 张丽,周兰英,肖千文,等.正交试验设计在建立杜鹃花RAPD-PCR反应体系中的应用[J].北方园艺,2007,(5):124-126.
[28] 黄春琼,周少云,刘国道,等.狗牙根SRAP-PCR反应体系的优化[J].基因组学与应用生物学,2009,28(5):981-989.
[29] 周良彬,卢欣石,赖黎丽.杂花苜蓿SRAP-PCR反应体系的正交优化[J].草地学报,2010,18(3):345-351.
[30] 昝逢刚,吴转娣,曾淇,等.荔枝SRAP-PCR反应体系的优化[J].基因组学与应用生物学,2009,28(1):132-136.
[31] 许永华,王士杰,陈晓林,等.人参SRAP-PCR体系优化条件的建立(英文)[J].农业科学与技术,2010,11(4):56-58.
[32] 艾鹏飞,武玉芬,魏景芳.小麦SRAP-PCR体系的正交设计优化(英文)[J].农业科学与技术,2011,12(3):366-368,374.
[33] 仲丛来,丁贵杰,沈凌麻.麻疯树SRAP-PCR反应体系的优化[J].贵州农业科学,2010,38(4):19-22.
[34] Guo D L,Luo Z R.Genetic relationships of some PCNA persimmons(Diospyros kaki Thunb.)from China and Japan revealed by SRAP analysis[J].Genetic Resources and Crop Evolution,2006,53(8):1597-1603.
[35] 郭大龙,罗正荣.部分柿属植物SRAP-PCR反应体系的优化[J].果树学报,2006,23(1):138-141.
[36] 孙佩光,奚如春,钮世辉,等.油茶SRAP-PCR反应体系的建立和优化[J].基因组学与应用生物学,2010,29(6):1192-1199.
[37] 陈庆榆,缪成贵,刘晓锋.棉花高质量DNA的提取及SRAP反应体系的优化[J].生物学杂志,2010,27(5):31-34.
