铅锌矿区土壤细菌群落多样性分析1)
2012-06-13任广明曲娟娟闫立龙孙兴滨
任广明 张 琦 曲娟娟 闫立龙 孙兴滨
(东北农业大学,哈尔滨,150030) (哈尔滨工程大学) (东北农业大学) (东北林业大学)
铅锌通常是矿区土壤中含量较高的有毒重金属,它们在土壤中总是不断地发生时空迁移和价态、形态转化[1]。这不仅影响了土壤微生物的种类和数量、土壤养分的转化等生化转化过程,同时影响着植物的生长质量、产量等[2]。铅锌等大多数重金属具有可迁移性差,不能降解等特点,会在生态系统中不断积累,毒性不断增强,从而导致生态系统的退化[3]。土壤微生物的群落结构是表征土壤生态系统群落结构和稳定性的重要参数,它们不仅调节着土壤动植物残体和土壤有机物质及其它有害化合物的分解、生物化学循环和土壤结构的形成等过程,且对外界干扰比较灵敏,能够较早地预测土壤环境质量的变化过程,被认为是最有潜力的敏感性生物指标之一[4-5]。土壤中铅锌等重金属的存在无疑会对土壤微生物的群落构成产生一定的影响,甚至会引起优势种群的改变,同样会引起微生物多样性的降低。因此,土壤重金属污染与土壤微生物群落之间的关系是国内外环境科学领域的一个研究热点[6]。本试验对黑龙江省某铅锌矿区土壤细菌群落结构多样性及土壤酶活性进行了研究,旨在探讨该矿区铅锌污染程度与土壤酶活性、土壤细菌群落结构多样性之间的相互关系,为同类污染矿区土壤环境质量评价提供科学依据。
1 材料与方法
样品采集:以铅锌矿区采矿口为基点,在距离矿口 100、200、400、600、800、1 000 m 处按对角线取样法多点混合方式取样,取样深度20 cm。非矿区土壤样品采自东北农业大学园艺学院试验站。将采取土样装入无菌封口塑料袋内,带回实验室。一部分新鲜土样置于冰箱内保存用于土壤细菌总DNA的提取;另一部分土样置于室内自然风干用于土壤理化性质及土壤酶活性测定。
土壤重金属含量、理化性质及酶活性测定:土壤铅锌全量采用王水—高氯酸强酸消解法消化[7],原子吸收分光光度法测定;土壤理化性质按常规方法测定[8];过氧化氢酶、脲酶、磷酸酶酶活性按关松荫方法测定[9]。
土壤细菌群落结构多样性分析:土壤总DNA的提取采用改良的Zhou等提出的方法[10]。纯化采用天根通用型DNA纯化回收试剂盒。应用细菌16S rDNA V3区域通用引物对提取的矿区土壤总DNA样品进行扩增[10]。PCR反应条件为:94℃预变性5 min,94℃变性1 min,65 ℃退火45 s,72 ℃延伸1 min,30 个循环,72℃最终延伸10 min。扩增产物1.0%琼脂糖凝胶电泳检测。采用Bio-Rad公司DcodeTM的基因突变检测系统对PCR反应产物进行分离。变性梯度为30% ~60%,使用的电泳缓冲液为1×TAE,上样量为20 μL DNA+10 μL 6×溴酚蓝二甲苯氰溶液,电泳电压150 V,60℃,300~350 min。凝胶染色采用银染法。
数据统计分析:数据采用SPSS 13.0、NTSYS等统计软件分析。将凝胶成像上出现的DNA片段计为1,不出现的计为0,统计后输入电脑,用NTSYS聚类分析软件进行UPGMA法聚类分析,同时运用软件构建7个土壤样品细菌群落结构相似性树状图。DGGE条带的数目可代表微生物群落的丰富度(S)。基于DGGE条带的迁移率和条带的强度分析细菌群落结构多样性,细菌群落结构多样性指数用Shannon-Weaver指数H来表示。Shannon-Weaver指数:H=-∑(ni/N)log(ni/N)。式中:ni为峰面积,N为所有峰总面积。
2 结果与分析
2.1 土壤重金属铅锌全量及酶活
2.1.1 土壤重金属铅锌全量
供试土壤重金属铅锌全量分析结果如表1所示。从矿口到距离矿口1 000 m处矿区土壤的重金属铅锌全量逐渐降低,重金属铅全量分别是非矿区土壤(7 号)的 32.98、18.93、15.49、9.48、9.01、6.07倍;重金属锌全量分别高出非矿区土壤10.21、10.09、9.63、8.48、7.47、5.82 倍,表明矿区土壤均受到不同程度的重金属复合污染。数据经LSR测验表明,从矿口到外围各土壤中铅锌全量与非矿区土壤中铅锌全量均存在显著性差异(P<0.05)。土壤C/N比的变化与所在环境的水热条件和成土作用过程密切相关[11]。从表1可以看出,供试土壤C/N比较低,这可能是由于矿区特定的地理环境所致。pH及阳离子交换量一定程度上反应土壤胶体结构、缓冲能力、土壤肥力等,可见供试的矿区土壤缓冲能力、肥力较低,其pH和阳离子交换量与非矿区土壤均存在显著性差异(P<0.05)。

表1 供试土壤的理化性质
2.1.2 土壤酶活性测定
由表2可见,从矿口到外围土壤过氧化氢酶、磷酸酶、脲酶活性与非矿区土壤酶活性均存在极显著性差异(P<0.01)。3种土壤酶活性的变异系数分别为 37.60%、26.75%、47.22%,其中脲酶活性变异系数最大,说明矿区土壤脲酶活性的变化程度最大。从表3可知,土壤过氧化氢酶活性与全铅、全锌质量分数呈显著负相关(P<0.05);土壤磷酸酶活性与全铅质量分数呈显著负相关(P<0.05),与全锌质量分数呈极显著负相关(P<0.01);土壤中的脲酶活性与全铅质量分数呈显著负相关(P<0.05),与全锌质量分数呈极显著负相关(P<0.01)。土壤中的酶对土壤重金属污染均较为敏感,矿区土壤中的重金属污染均抑制了过氧化氢酶、磷酸酶、脲酶活性。
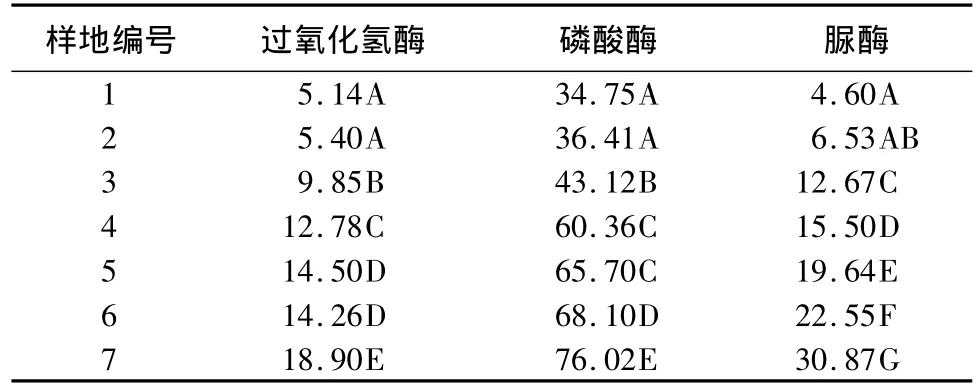
表2 重金属污染下土壤酶活性变化 μg·g-1
2.2 土壤细菌群落结构多样性
2.2.1 PCR-DGGE 指纹图谱
用改良的化学裂解法提取土壤总DNA,DNA片段大小为23.1 kb。用天根土壤DNA纯化回收试剂盒纯化土壤总DNA,纯化后进行土壤总DNA的PCR扩增,得到大小在230 bp左右的扩增产物,如图1。

图1 土壤细菌16S rDNA基因V3区PCR扩增结果

表3 土壤酶活性与铅锌全量之间的相关系数
DGGE指纹图谱中条带越丰富表明土壤微生物群落种类相对越丰富,条带颜色越深在一定程度上可以反映该种微生物所占比例越大,数量越多[12]。图2中,1号、2号、3号图谱条带相对较少,说明重金属污染严重的土壤中铅锌对细菌产生了明显的毒害作用,使得少部分细菌不能进行正常生长,致使细菌种群数量减少甚至衰亡,发生菌群的缺失,直接导致细菌的群落结构发生改变;从图2正三角标注的条带可知,重金属污染土壤中存在具有较强耐受重金属能力的共有细菌,并作为优势细菌稳定存在。图谱中条带的明暗变化表明这些细菌虽然稳定存在,但仍受到重金属铅、锌不同程度上的影响。4号、5号、6号图谱细菌种群及数量有明显增加(倒三角标注),这是由于土壤中的细菌长期在重金属的选择作用下,不断增强自己的耐性、抗性,具有了高度的选择性,通过各种氧化还原、酶化等代谢活动来适应重金属污染的环境;7号图谱表现出较丰富的细菌群落结构多样性。

图2 土壤细菌16S rDNA的PCR-DGGE指纹图谱
细菌群落相似性聚类分析主要根据DGGE图谱中每个样品中不同条带的强度和迁移率对每个样品的条带图谱进行细菌群落相似性聚类分析。通过NTSYS聚类分析软件将供试的7个土壤样品的指纹图谱进行UPGMA法聚类分析。从图3可以看出:供试的7个土壤样品在相似距离为10时分为4簇,第1簇是4号、5号、6号;2号、3号归为一簇;1号,7号各成一簇。

图3 DGGE聚类分析图谱
2.2.2 土壤细菌群落丰度和多样性指数
DGGE条带的数目可代表微生物群落的丰富度(S)。从表4可知,不同土壤细菌群落的丰富度存在差异,其中非矿区土壤(7号)图谱中条带数最多(19条),其细菌群落丰富度最大,矿区1号土壤的条带数最少(7条),其微生物群落丰富度最小。与非矿区土壤相比矿区土壤的微生物群落丰富度分别为非矿区土壤的 26.92%、30.77%、34.62%、38.46%、50%、69.23%,土壤细菌群落的丰富度简明地表达了土壤细菌群落多样性的一个方面,但它未能反映群落功能多样性相对多度的信息[13]。

表4 供试土壤微生物群落丰富度和多样性指数
Shannon-Weaver(H)多样性指数是表征群落中物种数目多少、衡量群落结构和重要性的基础。物种多样性指数越大说明各个种个体数量分布越均匀、种类越多[14]。从表4可知,土壤(1~6号)的细菌群落多样性Shannon-Weaver指数明显低于非矿区土壤(7号),最大为1.15,最小为0.84。统计分析显示,除1号和2号土壤细菌群落 Shannon-Weaver指数不存在差异性外,其它供试土壤细菌群落Shannon-Weaver指数的差异达极显著水平(P<0.01)。可见,土壤中细菌的群落结构受到了重金属不同程度上的影响,导致细菌的群落结构及数量出现了相应的降低。
3 结论与讨论
PCR-DGGE是1993年由Myuzer首先应用到微生物分子生态学的一门分子生物技术[15-16],是研究微生物群落多样性、分析环境中微生物群落动态变化情况、了解生境对微生物群落多样性及菌群活性影响的一种分子技术[5,17]。运用DGGE方法可以揭示环境污染条件下微生物群落结构多样性[18]。kozdrój等[19]、赵祥伟等[20]、张洪勋等[21]已成功将PCR-DGGE方法应用于微生物群落结构多样性分析。本试验通过测定土壤理化性质及酶活发现,矿区土壤重金属铅锌污染严重,铅锌质量分数与土壤酶活均成显著负相关,这说明土壤中的重金属抑制了细菌的正常生长,土壤细菌群落结构发生改变,进一步影响土壤酶活性变化,土壤酶活性较非矿区土壤偏低;试验建立了适用于PCR-DGGE技术的重金属污染土壤基因组DNA的快速提取方法,获得了纯度较高的DNA模板,保证了PCR扩增反应的稳定性和重现性,进而使得细菌指纹图谱及聚类分析真实有效。DGGE聚类分析结果显示,距离矿口100、200、400 m土壤样品中细菌群落结构相似性较强,相似距离相近,距离矿口600、800、1 000 m归为一簇表观上条带有所增多,这是由于较低质量分数铅、锌对矿区土壤细菌影响较小,重金属污染矿区土壤中重金属铅、锌质量分数越高土壤中细菌群落及数量越少,这与 Hiroki[22]、滕应等[11]、Bisessar等[23]的研究结果相一致。重金属污染土壤真菌、放线菌试验正在进行中。从上述研究结果可以看出,土壤细菌群落结构多样性是指示矿区土壤环境质量变化灵敏、有效的生物学指标之一,并为进一步研究重金属污染条件下土壤的质量与功能提供了基础。
[1]Aksoy A,Hale W H,Dixon J M.Capsella bursa-pastoris(L.)Medic.as a bio-monitor of heavy metals[J].The Science of the Total Environment,1999,226(2/3):177-186.
[2]Ustyak S,Petrikova V.Heavy metal pollution of soils and crops in Northern Bohemia[J].Applied Geochemistry,1996,11(1/2):77-80.
[3]黄铭洪.环境污染与生态恢复[M].北京:科学出版社,2003.
[4]陈怀满.土壤—植物系统中的重金属污染[M].北京:科学出版社,1996.
[5]滕应,黄昌勇.重金属污染土壤的微生物生态效应及修复研究进展[J].土壤与环境,2002,11(1):85-89.
[6]王秀丽,徐建民,姚槐应,等.重金属铜、锌、镉、铅复合污染对土壤环境微生物群落的影响[J].环境科学学报,2003,23(1):22-27.
[7]国家环境保护局.GB 15618—1995 土壤环境质量标准[S].北京:中国环境科学出版社,1996:11-12.
[8]鲁如坤.土壤农业化学分析法[M].北京:中国农业科学技术出版社,1999:107-240.
[9]关松荫.土壤酶及其研究法[M].北京:农业出版社,1986:274-320.
[10]Zhou Jizhong,Bruns M A,Tiedje J M.DNA recovery from soils of diverse composition[J].Applied and Environmental Microbiology,1996,62(2):316-322.
[11]滕应,黄昌勇,龙健,等.铅锌银尾矿污染区土壤微生物区系及主要生理类群研究[J].农业环境科学学报,2003,22(4):408-411.
[12]肖勇,杨朝晖,曾光明,等.PCR-DGGE研究处理垃圾渗滤液序批式生物膜反应器(SBBR)中的细菌多样性[J].环境科学,2007,28(5):1095-1101.
[13]Bronwyn D H,Raymond L C,Wendy M,et al.Using the gini coefficient with BIOLOG substrate utilization data to provide an alternative quantitative measure for comparing bacterial soil communities[J].Journal of Microbiological Methods,1997,30(1):91-101.
[14]Magurran A E.Ecological diversity and its measurement[M].Princeton:Princeton University Press,1988:34-59.
[15]Muyzer G,de Waal E C,Uitterlinden A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied and Environmental Microbiology,1993,59(5):695-700.
[16]Watanabe M E.Phytoremediation on the brink of commercialization[J].Environ Sci Technol,1997,31(4):182-186.
[17]刘惠军,杨凤林,张捍民,等.炭膜曝气生物膜反应器硝化作用及其微生物群落结构分析[J].环境科学,2007,28(9):2123-2128.
[18]Kozdrój J,Van Elsas J D.Structural diversity of microbial communities in arable soils of a heavily industrialised area determined by PCR-DGGE fingerprinting and FAME profiling[J].Appl Soil Ecol,2001,17(1):31-42.
[19]Kozadrój J,Van Elsas J D.Application of polymerase chain reaction-denaturing gradient gel electrophoresis for comparison of direct and indirect extraction methods of soil DNA used for microbial community fingerprinting[J].Biol Fertil Soils,2000,31(5):372-378.
[20]赵祥伟,骆永明,腾应,等.重金属复合污染农田土壤的微生物群落遗传多样性研究[J].环境科学学报,2005,25(2):186-191.
[21]张洪勋,王晓谊,齐鸿雁.微生物生态学研究方法进展[J].生态学报,2003,23(5):988-995.
[22]Hiroki M.Eeffets of Heavy metal contamination on soil microbial population[J].Soil science and plant nutrition,1992,38(1):141-147.
[23]Bisessar S.Eeffet of heavy metal on microorganisms in soil near a secondary lead smelter[J].Water,Air,and Soil Pollution,1982,17:305-308.
