赤点石斑鱼群体遗传结构的微卫星分析*
2012-05-09陈省平丁少雄陈嘉慧
陈省平,丁少雄,陈嘉慧
(1.中山大学海洋功能分子广东高校重点实验室,广东 广州510080;2.中山大学中山医学院,广东 广州510080;3.厦门大学海洋科学与环境学院,福建 厦门361005)
赤点石斑鱼Epinephelusakaara,主要分布在日本南部,朝鲜半岛,以及中国南海和东海[1],经济价值高,是在水产养殖中很有经济价值的品种之一。然而,大规模水产养殖的赤点石斑鱼的种苗仍然依赖于天然的种质资源。幼体成活率低和人工养殖条件不稳定,使建立人工育种生产技术受到阻碍[2-3]。再加上近年来,中国的赤点石斑鱼因被过度捕捞,迅速减少。因此,必须采取有效的措施,监测、保护和利用这一物种与其遗传资源。
通过遗传信息和地理数据的分析,评估在中国的赤点石斑鱼的遗传多样性,也是保护种质资源的一个重要措施。目前常用的各种遗传标记中,简单重复序列标记(SSR)已被用在评估在植物的遗传多样性,以及动物遗传信息的高度多态性、基因组多样性、共显性与适应性等方面[4-5]。SSR是一类均匀地分布在真核生物基因组上的串联重复序列(一般为2~6 bp)[5]。已经从夏威夷石斑鱼中已分离得到的9个SSR位点,为其群体遗传结构和基因流的研究提供了有力的证据[6]。Nugroho等[7]研究了6种石斑鱼的SSR变化规律。细鳞鼻鲈和黑石斑鱼、黑鼻鲈的SSR,已被用于红石斑鱼E.morio的种群遗传分析[8]。因此,SSR作为石斑鱼及其相近的类群之间遗传学分析的分子标记是合理的。然而,因为SSR的开发成本高和工作量大,在赤点石斑鱼的研究中并没有SSR的报道。
本文参考已发表的赤点石斑鱼近缘种的微卫星序列,设计了赤点石斑鱼的微卫星引物,开展了赤点石斑鱼群体的微卫星分析,对赤点石斑鱼沿海7个群体的遗传变异进行了研究,以其为赤点石斑鱼的种质资源研究和遗传育种提供遗传学基础,奠定进一步开展遗传育种的标记基础。
1 材料和方法
1.1 样品收集和地理分布
2004-2005年期间,在中国南海和东海的7种不同的典型的渔场(图1,表1)用分层抽样的方法随机收集赤点石斑鱼样本,共采集87个样品。按照有关生物样品募集协议和中国保护动物福利和的法律要求,获得审查机构委员会批准。
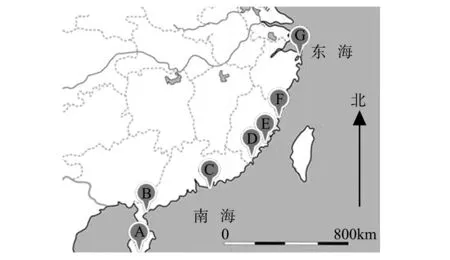
图1 赤点石斑鱼7个群体的地理分布
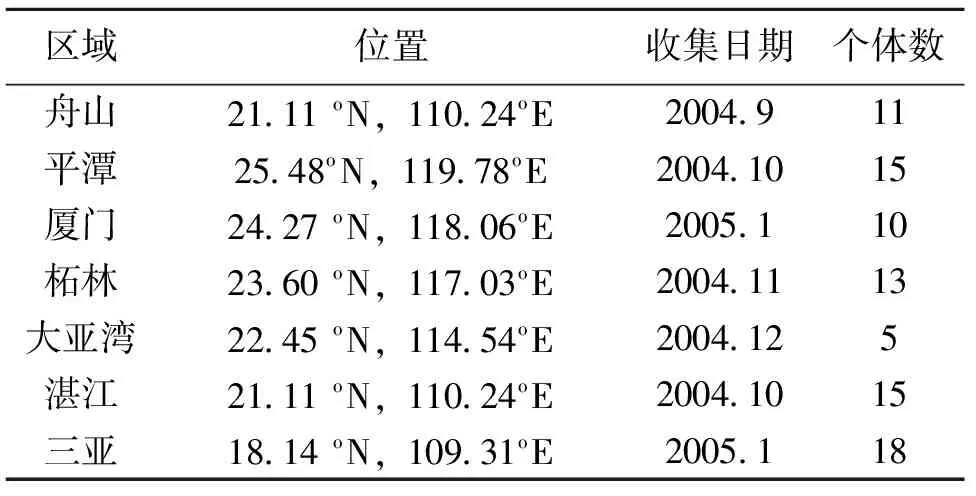
表1 实验用采集的赤点石斑鱼详细信息
1.2 基因分型
从石斑鱼开发的25个SSR引物用来测试赤点石斑鱼(表2)。5个引物可用于跨物种的转移性测试(表3, 4)。PCR反应在GeneAmp PCR System 9700 PCR仪上进行,PCR反应混合物中约20 ng的模板DNA,每个引物各0.2 M,各种dNTPs 200 M,2.0 mN MgCl2, ExTaq聚合酶1U(TAKARA),最后加入1 × PCR缓冲液使终体积为25 L。PCR反应程序为:95 ℃预变性5 min,然后进行35个循环,每个循环包括95 ℃ 30 s,退火30 s(表4),72 ℃ 30 s,最后72 ℃延伸5 min。采用w=6%的变性聚丙烯酰胺凝胶分离PCR反应产物,50 w恒压50 ℃恒温电泳。参照Bassam[9]将凝胶银染染色。在版本4.4(BIO-RAD)的软件中采用20 bp Marker(Sigma)来估算条带大小。对87个个体进行了微卫星位点分析。所有样品扩增和电泳使用的方法如上所述。

表2 实验用微卫星标记
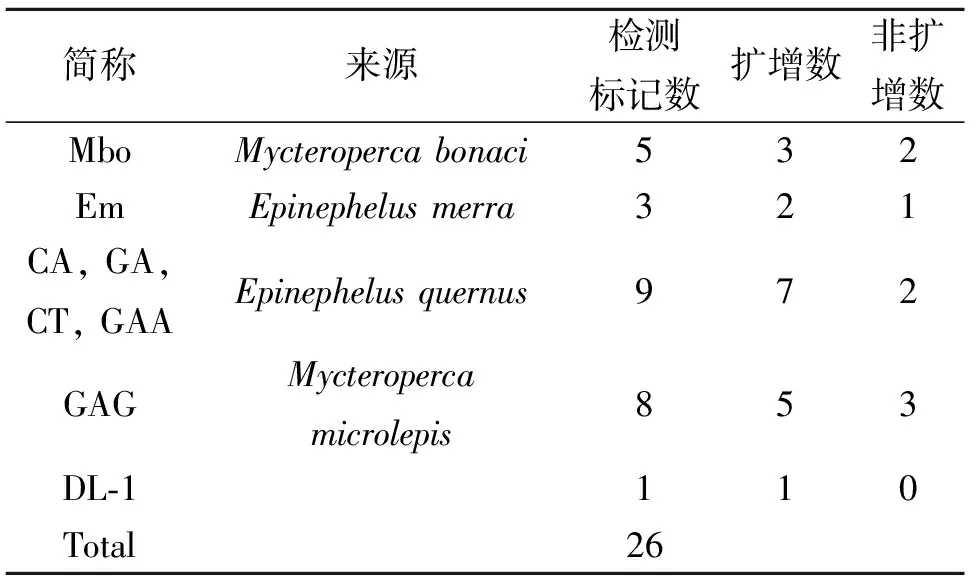
表3 赤点石斑鱼微卫星标记跨物种扩增

表4 遗传分析用7个微卫星标记
1.3 数据分析
为了分析不同种群的的微卫星位点的多样性,在每个位点都测定了等位基因数(A)、等位基因大小范围(S)、大小(Sc)和最常见的等位基因频率(Fc)。通过POPGENE软件(http://wbiomed.curtin.edu.au/genepop/)计算等位基因频率。如果一个等位基因的频率小于0.05,则是低频率基因。等位基因丰富度指数(Ar)[10],是通过软件Fstat计算获得的[11],可以在不考虑群体样品量的情况下,直接比较所获得的等位基因平均数。观测杂合度(Ho)是作为一个比值显示杂合子基因型在每个位点的得分。He值是由公式He=1-∑p2得到,其中pi是某个位点i个等位基因出现的频率[12]。有效等位基因数(ne)根据公式ne=1/∑x2计算得到,其中xi为每个微卫星位点的某个位点i个等位基因[13]。观察基因型构象的Hardy-Weinberg平衡(HWE),通过软件GENEPOP标准的假设检验进行测试。Bonferroni校正法进行多重比对序列[14]。通过2.9.3版本FSTSAT软件计算种群之间固定指数(FST)[11,15]。根据群体之间Nei’s标准遗传距离DA[12],分析7组之间的亲缘关系,并用MEGA软件(3.1版)绘制UPGMA聚类树。
2 结 果
2.1 赤点石斑鱼基因组中SSR位点扩增
从其他石斑鱼开发的25个SSR标记,其中15个(60%)在所有被测样品中扩增(表1)。这些选择的标记(表2中下划线表示)呈现多态性,并用作进一步的遗传分析。从石斑鱼中获得的SSR-PCR产物大小范围相似,表明15个SSR标记能有效地应用到赤点石斑。另一方面,剩下的10个SSR标记(40%)没有得到扩增。
交叉扩增法可以节约样本和反应次数,并且已有研究证明对于SSRs分析也是有效的。有报道称,35.7%的银鲫的微卫星扩增和多态性特征与全局性的PCR产物结果一致[16]。之前在其他物种有报道称,SSR可能在相近物种间高频率的迁移[17-18]。可转移性表明,引物结合位点基因组DNA可能在长期的进化中保守。成功地利用依据斑节对虾Penaeusmonodon设计的通用引物对其相近种群,包括中国明对虾Fenneropenaeuschinensis,凡纳滨对虾Litopenaeusvannamei和日本囊对虾Marsupenaeusjaponicus进行微卫星分析,就是一个很好的例子[19]。张等[20]提出,微卫星标记达到10%(3 / 30)则可认为是其它三个相关的虾种中的常见标记。微型卫星的转移率的高低可从物种间的遗传关系确定[21]。由此推测,研究其他石斑鱼的SSR标记有利于对研究赤点石斑鱼遗传的研究。
2.2 微卫星标记多样性分析
利用7对微卫星标记对7个群体的87个体的赤点石斑鱼基因多样性进行分析。选择这7个SSR标记,是因为它们的多态性程度各异。包括鲈鱼Mycteroperca的3个SSR标记(Mbo061,Mbo066和GAG007),石斑鱼Epinephelus的3个SSR标记(EM-1,CA-2和CA-6),以及由我们的实验室根据石斑鱼公共数据库设计的DL - 1。结果显示只有一对引物(CA-2)在大亚湾和湛江两群体扩增结果为单态,其余都为多态。对87个个体进行研究,在基因微位点上共检测到45个等位基因,平均每个引物获得6.4个等位基因,其中,引物Mbo066获得11个等位基因,等位基因数最多;Em-1获得4个等位基因,等位基因数最少。等位基因丰富度指数(Ay)在2.4~3.7之间。三亚种群的Mbo066位点等位基因丰富度指数最大(Ar= 6.3)。7个基因微点上的等位基因片段大小在102~781 bp之间。在所有种群中EM-1位点显示等位基因(B)基因频繁最高的,在其他6个位点的检测到的等位基因数量不一:, CA-2 三个,CA- 6二个,DL- 1四个, GAG007三个,Mbo061 三个,Mbo066五个。等位基因的大小,最常见的等位基因的大小,每个位点常见的等位基因频率如表5所示。
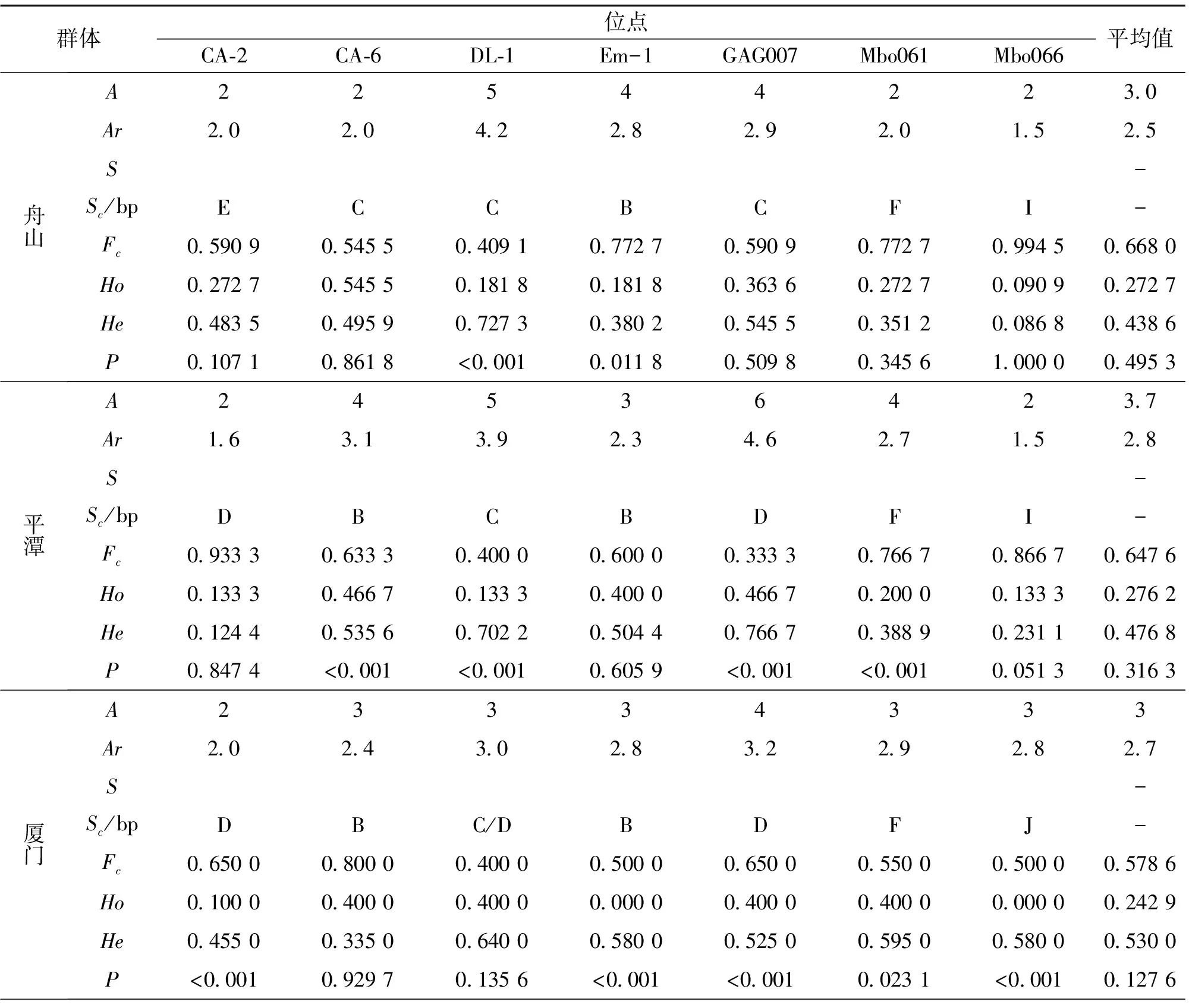
表5 种群名称、等位基因数、主要等位基因、预期杂合度、观测杂合度、基因丰富度指数、有效等位基因数和Hardy-Weinberg检验P值
(续上表)
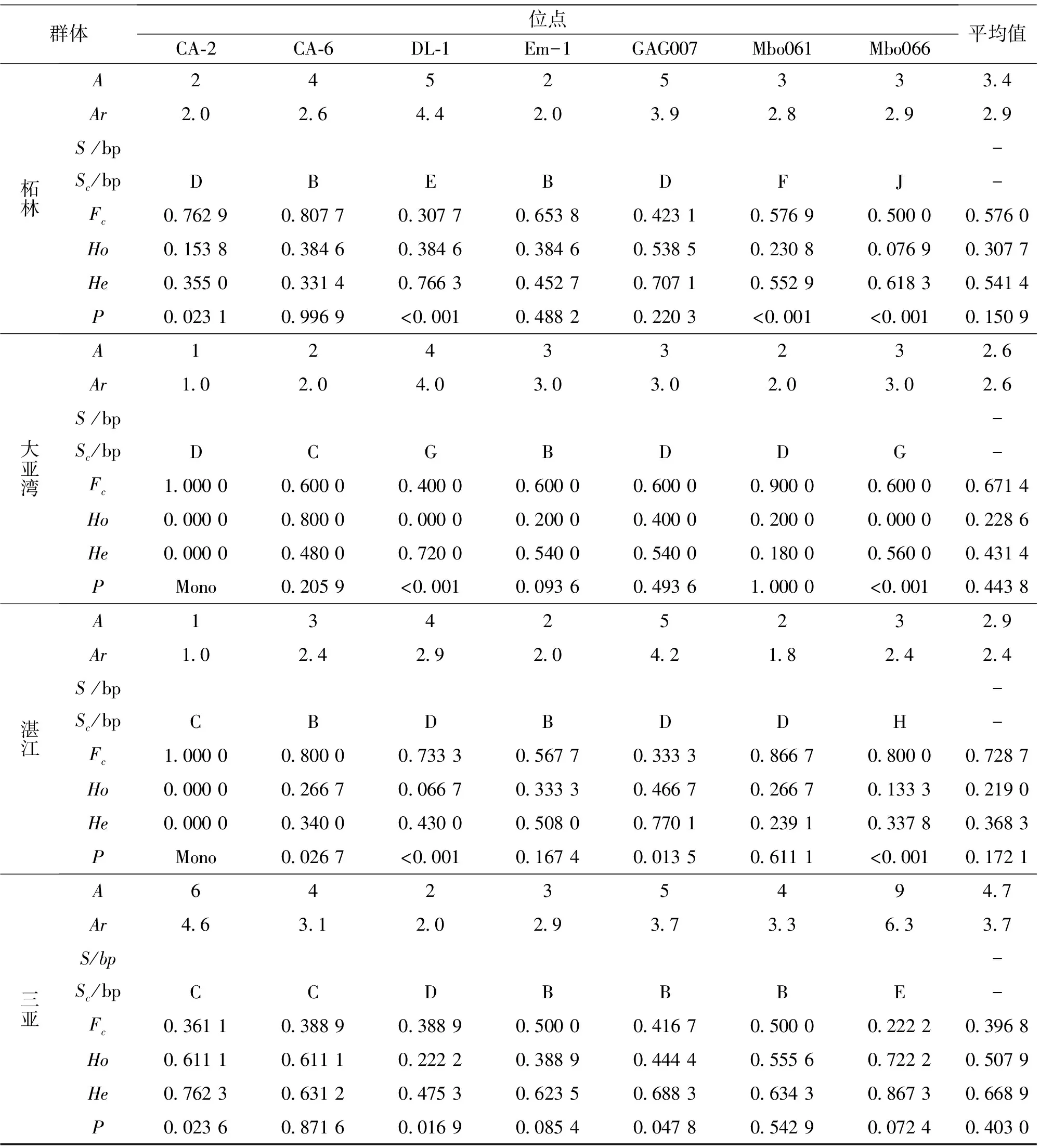
群体位点CA⁃2CA⁃6DL⁃1Em-1GAG007Mbo061Mbo066平均值柘林A24525333 4Ar2 02 64 42 03 92 82 92 9S/bp-Sc/bpDBEBDFJ-Fc0 76290 80770 30770 65380 42310 57690 50000 5760Ho0 15380 38460 38460 38460 53850 23080 07690 3077He0 35500 33140 76630 45270 70710 55290 61830 5414P0 02310 9969<0 0010 48820 2203<0 001<0 0010 1509大亚湾A12433232 6Ar1 02 04 03 03 02 03 02 6S/bp-Sc/bpDCGBDDG-Fc1 00000 60000 40000 60000 60000 90000 60000 6714Ho0 00000 80000 00000 20000 40000 20000 00000 2286He0 00000 48000 72000 54000 54000 18000 56000 4314PMono0 2059<0 0010 09360 49361 0000<0 0010 4438湛江A13425232 9Ar1 02 42 92 04 21 82 42 4S/bp-Sc/bpCBDBDDH-Fc1 00000 80000 73330 56770 33330 86670 80000 7287Ho0 00000 26670 06670 33330 46670 26670 13330 2190He0 00000 34000 43000 50800 77010 23910 33780 3683PMono0 0267<0 0010 16740 01350 6111<0 0010 1721三亚A64235494 7Ar4 63 12 02 93 73 36 33 7S/bp-Sc/bpCCDBBBE-Fc0 36110 38890 38890 50000 41670 50000 22220 3968Ho0 61110 61110 22220 38890 44440 55560 72220 5079He0 76230 63120 47530 62350 68830 63430 86730 6689P0 02360 87160 01690 08540 04780 54290 07240 4030
Ho和He的值在表5列出。除了位点CA- 2,对大亚湾和湛江种群的47对等位基因进行Hardy-Weinberg平衡测试。结果表明,其中16个测试样品经Bonferroni法多重测试校正后具有统计学意义。过高的同源性使得FST值偏离Hardy - Weinberg平衡期望。
表6将7个种群的FST值进行了两两比较,进行遗传分化分析的结果显示,大多数群体间有显著的统计学差异。只有4对没有明显差异:舟山和平潭,大亚湾和平潭,厦门和柘林,柘林和大亚湾。同时,也通过比较等位基因和基因型频率,探讨了野生种群的遗传差异(未列出)。发现只有平潭和舟山,厦门和柘林间无表现出两两之间分化。基于群体间遗传距离(DA)构建的UPGMA树(见图2),分析7个群体之间的遗传关系,结果与AFLP的结论基本一致[22]。
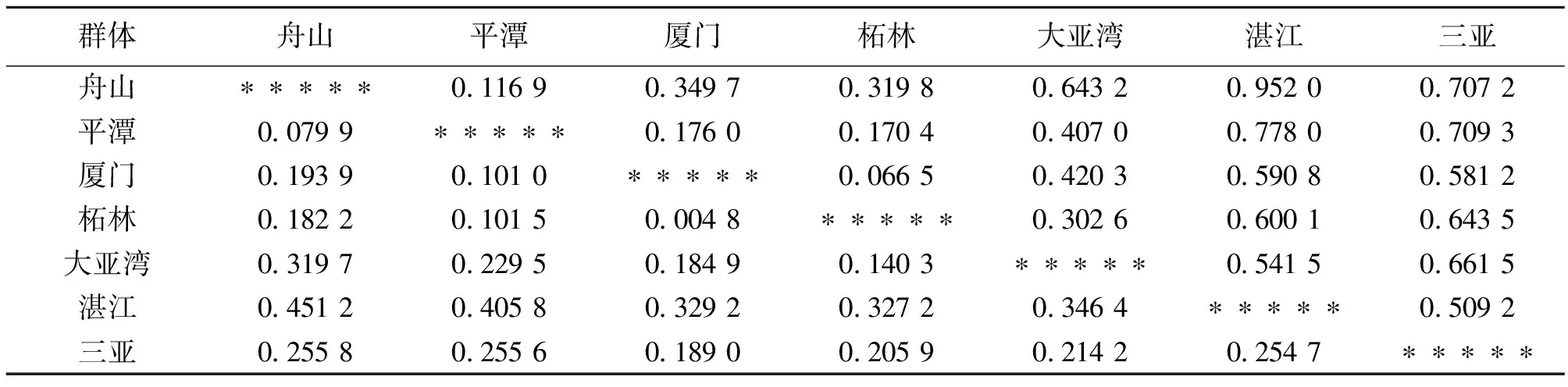
表6 基于7个微卫星位点的群体间的遗传距离(对角线上)和Fst值(对角线下)

图2 赤点石斑鱼7个群体的UPGMA聚类树
3 讨 论
遗传多样性是鱼类生物多样性的主要组成部分,由于鱼类遗传多样性的大小直接反映鱼类对环境适应能力的大小,种内遗传多样性越丰富,物种对环境的适应能力越强,赤点石斑鱼遗传多样性水平较低,赤点石斑鱼生长周期较长,并且该鱼雌雄同体雌性先成熟,低龄鱼为雌鱼,高龄鱼为雄鱼。据统计,石斑鱼群体组成趋向低龄化,这说明自然海区雄鱼数量在减少,雌雄比例失调,反映出资源基础已受到捕捞的影响。
微卫星标记的多态性来源于核心序列重复次数的变化,信息含量丰富,适用于分析群体的遗传结构。微卫星比其他分子标记(如RFLP、RAPD、AFLP等)更能揭示遗传多样性。微卫星标记在鱼类遗传与育种中主要应用于构建分子遗传图谱、种质鉴定及遗传多样性分析等方面。
本研究检测的7个微卫星位点中,大部分群体在位点DL-1上显著偏离Hardy-Weinberg平衡;其他位点上,每个位点至少有两个群体显著偏离Hardy-Weinberg平衡;赤点石斑鱼经济价值高,自然资源捕捞压力很大,受捕捞压力和环境变化的影响,其资源量持续下降。遗传漂变和人工网箱养殖群体对自然种群的影响可能导致了Hardy-Weinberg的偏离。
Wright对Fst的大小与分化程度的关系进行了界定:分化系数在0~0.05之间,群体间分化很弱,0.05~0.15之间表示分化中等,0.15~0.25之间表示分化大,大于0.25表示分化极大[23]。本研究表明,各群体的遗传分化系数在0.004 8~0.255 8之间,大部分群体间有显著分化,其中三亚和湛江两个群体与其余5个群体相比,表现出分化大或极大,并且三亚和湛江群体间呈现极显著差异。
导致赤点石斑鱼不同群体存在明显遗传差异的原因,可能跟赤点石斑鱼生态习性有关,赤点石斑鱼属于定居性鱼类,栖息场所为沿岸岩礁、珊瑚礁[24]。对赤点石斑鱼的标志放流进行研究,发现标志重捕赤点石斑鱼不论是当年或第二年、第三年均在放流处附近不超过2海里的水域重捕到,这说明赤点石斑鱼栖息具有明显的地域性。赤点石斑鱼的这种生态习性,限制了不同群体的基因交流,产生了地理隔离造成了不同群体间存在显著的遗传差异。综合线粒体DNA、AFLP和微卫星结果,赤点石斑鱼7个群体可以明显分成两个种群,舟山、平潭、厦门、柘林和大亚湾群体为北方种群,三亚和湛江群体为南方种群。对于两个种群分化的原因,除了自身生态习性外,温度、盐度、底质和海流等因素可能限制了不同群体的交流,造成了种群的分化。
微卫星标记客观的反映了赤点石斑鱼的群体遗传变异,揭示了我国沿海赤点石斑鱼种群间存在显著差异,存在两个有明显遗传差异的种群,这为赤点石斑鱼的遗传育种工作提供了遗传背景,并为赤点石斑鱼的种质资源保护和网箱养殖的健康发展提供了理论支持。
参考文献:
[1]MASUDA H,AMAOKA K,ARAGA T,et al.The Fishes of the Japanese Archipelago [M].Tokyo:Tokai University Press,1984: 37-38.
[2]UKAWA M,HIGUCHI M,MITO S.Spawning habits and early life history of a serranid fish,Epinephelusakaara(Temminck et Schlegel) (in Japanese with English summary) [J].Jpn J Ichthyol,1966,13: 156-161.
[3]FUKUNAGA K,NOGAMI K,YOSHIDA Y,et al.The latest developments and problems in seed production of the red spotted grouper at Tamano station of Japan Sea-Farming Association [J].Saibai Giken,1990,19: 33-40.
[4]POWELL W,MACHRAY G,PROVAN J.Polymorphism re- vealed by simple sequence repeats [J].Trends Plant Sci,1996,1: 215-222.
[6]RIVERA M A J,GRAHAM G C,RODERICK G K.Isolation and characterization of nine microsatellite loci from the Hawaiian grouper Epinephelus quernus (Serranidae) for population genetic analyses [J].Mar Biotechnol,2003,5: 126-129.
[7]NUGROHO E,TAKAGI M,SUGAMA K,et al.Detection of GT repeats microsatellite loci and their polymorphism for grouper of the genus Epinephelus [J].Fisheries Sci,1998,5: 836-837.
[8]ZATCOFF M S,BALL A O,SEDBERRY G R.Population genetic analysis of red grouper,Epinephelusmorio,and scamp,Mycteropercaphenax,from the southeastern U.S.Atlantic and Gulf of Mexico [J].Mar Biol,2004,144: 769-777.
[9]BASSAM B J,CAETANO-ANOLLES G,GRESSHOF P M.Fast and sensitive silver staining of DNA in polyacrylamide gels [J].Anal Biochem,1991,196: 80-83.
[10]EL MOUSADIK A,PETIT R J.Chloroplast DNA phylogeography of the argan tree of Morocco [J].Mol Ecol,1996,5: 547-555.
[11]GOUDET J.FSTAT,a program to estimate and test gene diversities and fixation indices (version 2.9.3).Available from http://www.unil.ch/izea/softwares/fstat.html.Updated from Goudet,J.(1995).FSTAT v-1.2.A computer program to calculate F-statistics [J].J Heredity,2001,86: 485-486.
[12]NEI M.Molecular Evolutionary Genetics [M].New York:Columbia University Press,1987: 40.
[13]KIMURA M,CROW J F.The number of alleles that can be maintained in a finite population [J].Genetics,1964,49:725-738.
[14]RICE W R.Analyzing tables of statistical tests[J].Evolution,1989,43: 223-225.
[15]WEIR B S,COCKERHAM C C.Estimating F-statistics for the analysis of population structure [J].Evolution,1984,38: 1358-1370.
[16]YUE G H,LI Y,LIM L C,et al.Monitoring the genetic diversity of three Asian arowana (Scleropagesformosus) captive stocks using AFLP and microsatellites [J].Aquaculture,2004,237: 89-102.
[17]SCOTT K D,EGGLER P,SEATON G,et al.Analysis of SSRs derived from grape ESTs [J].Theor Appl Genet,2000,100: 723-726.
[18]CORDEIRO G M,CASU R,MCINTYRE C L,et al.Microsatellite markers fromsugarcane (Saccharumspp.) ESTs cross transferable to erianthus and sorghum [J].Plant Sci,2001,160: 1115-1123.
[19]邵明瑜,张志峰,康庆浩,等.单环刺螠虫消化道组织学和细胞学的初步研究[J].中国水产科学,2003,10(4): 265-270.
[20]ZHANG Z,SHAO M,KANG K H.Classification of haematopoietic cells and haemocytes in Chinese prawnFenneropenaeuschinensis[J].Fish & Shellfish Immunology,2006,21: 159-169.
[21]ZHANG Z,SHAO M,KANG K H.Changes of enzyme activity and hematopoiesis in Chinese prawnFenneropenaeuschinensis(Osbeck) induced by WSSV and zymosan A [J].Aquacultre Research,2005,36: 674-681.
[22]陈省平,胡晓丽,刘涛.赤点石斑鱼7个地理群体的AFLP分析[J].中山大学学报:自然科学版,2009,48: 56-61.
[23]WRIGHT S.The genetical structure of populations[J].Ann Eugen,1951,15: 323-354.
[24]蒙子宁,杨丽萍,吴丰,等.斜带石斑鱼、赤点石斑鱼RAPD和线粒体Cyt b基因序列变异分析 [J].中山大学学报:自然科学版,2007,46: 75-80.
[25]ZATCOFF M S,BALL A O,CHAPMAM R W.Characterization of polymorphic microsatellite loci from black grouper,Mycteropercabonaci(Teleostei: Serranidae) [J].Mol Ecol Notes,2002,2: 217-219.
[26]CHAPMAN R W,SEDBERRY G R,KOENIG C C,et al.Stock identification of gag,Mycteroperca microlepis,along the southeast coast of the United States [J].Mar Biotechnol,1999: 1:137-146.
