4个品系西藏拟溞12S rRNA基因序列及其分子进化研究
2012-02-08赵文李睿
赵文,李睿
(大连海洋大学水产与生命学院,辽宁省水生生物学重点实验室,辽宁大连 116023)
4个品系西藏拟溞12S rRNA基因序列及其分子进化研究
赵文,李睿
(大连海洋大学水产与生命学院,辽宁省水生生物学重点实验室,辽宁大连 116023)
应用线粒体12S rRNA基因序列对4个地理品系的西藏拟溞 (纳木卡错品系、色林错品系、班戈湖品系、赛里木湖品系)进行分子系统发育研究,并结合GenBank中7种相近溞类的12S rRNA基因序列探讨了西藏拟溞的分子系统进化关系。结果表明,在4个品系西藏拟溞的4条12S rRNA基因序列中,A、T、G、C的平均含量分别为31.9%、32.45%、17.33%和17.95%,A+T的含量64.35%,G+C的含量为35.28%,A+T的含量明显高于G+C的含量,符合节肢动物A、T含量高的特点。聚类分析结果表明,西藏拟溞和其他溞属枝角类动物有着明显的遗传差异,说明西藏拟溞在枝角类生物学研究中有其独特的地位。遗传多样性计算结果表明:在4个品系的西藏拟溞中,纳木卡错品系与色林错品系以及纳木卡错品系与班戈湖品系的遗传相似性均较高,达98%左右;班戈湖品系与纳木卡错品系间的遗传距离最小,仅为0.0126,班戈湖与赛里木湖间的遗传距离最大,为0.0199;与其他溞属枝角类相比较,4个品系西藏拟溞种内差异相对不明显,而与其他溞种间的差异显著,西藏拟溞有着较高的遗传多样性水平。研究表明,12S rRNA基因序列包含有可靠的进化信息,可作为西藏拟溞种类鉴定良好的分子标记,同时利用这种分子标记法从分子系统进化方面证明了Daphnia tibetana和Daphniopsis tibetanaSars应分属于不同的种。
西藏拟溞;12S rRNA基因序列;遗传多样性;系统进化
西藏拟溞Daphniopsis tibetana Sars隶属于节肢动物门 Arthropoda、甲壳纲 Crustacea、鳃足亚纲Branchiapoda、枝角目Cladocera、溞科 Daphniidae、拟溞属Daphniopsis。 Sars[1]在 1903年对亚洲中部的枝角类调查中,最早发现了这一种类并对其进行了简单的介绍,因其具有介于溞属和低额溞属之间的中间特征,故将其归为拟溞属并命名为Daphniopsis tibetana。之后,Hedin[2]在1907年对西藏地区中、西部的调查研究时,在色林错也发现了这种体色较黑的西藏拟溞。赵文等[3]在对西藏羌北无人区科学考察中,再次检出此溞,并于2001年12月在实验室内将其进行海水驯化,获得了这一种类实验室内生物学参数的第一手材料。西藏拟溞主要分布于中国西藏、青海、新疆等地,在国外分布于前苏联和印度,为一冷水种和盐水种,在枝角类生物学研究中有其独特的地位[4]。西藏拟溞在中国西北部的很多半咸水湖中为优势种群,能在高寒、贫营养性盐水水体中存活,而且密度和生物量相当可观,经过引种或驯化后,西藏拟溞可作为寒冷季节海水鱼虾苗种高效适口的一种活饵料。目前,有关西藏拟溞遗传多样性的研究报道较少[5]。线粒体12S rRNA基因作为研究动植物相近种及种内群体间亲缘关系的有效标记,也是调查动物群体遗传多样性并了解物种形成及进化历程的有效工具。使用线粒体12S rRNA基因作为分子遗传标记,对于研究西藏拟溞的起源进化、亲缘关系以及遗传结构等方面都具有重要的意义。
1 材料与方法
1.1 材料
1.1.1 西藏拟溞 试验用西藏拟溞有西藏地区的3个品系和新疆地区的1个品系,经形态鉴定,本试验所用的西藏拟溞均为同一个种。西藏品系于2007年5月采自藏北3个湖泊——纳木卡错、色林错和班戈湖,分别记为NMKC、SLC和BG;新疆品系于2007年11月采自新疆赛里木湖,记为SLM。西藏拟溞4个品系的取样资料如表1所示,其中西藏班戈湖品系的样品采集后饥饿48 h后用液氮保存运回实验室;而其他群体均为在实验室内驯化培养的群体。
1.1.2 主要试剂 Wizard Genomic DNA Purification Kit试剂盒 (Promega Corporation USA),TaqDNA聚合酶10×PCR Buffer,引物 (上海生工生物工程技术服务有限公司产品),琼脂糖Bioscience。

表1 西藏拟溞的采样地点、时间和盐度Tab.1 Localities,time and salinity for samples of Daphniopsis tibetana Sars
1.2 方法
1.2.1 西藏拟溞的驯化培养 除班戈湖品系外其余3个西藏拟溞品系均驯化培养于大连海洋大学水生生物学重点实验室,根据每个种群的培养盐度(表1)配制各自的培养用水 (经煮沸消毒的海水),水温为16℃,用折射盐度计 (ATAGOS-10)检测稀释海水的盐度。用盐藻Dunaliella salina、蛋白核小球藻Chlorella pyrenoidosa等单胞藻进行投喂。
1.2.2 DNA的提取 使用Wizard Genomic DNA Purification Kit试剂盒分别对4个品系的西藏拟溞提取单只样品的基因组DNA。每个品系取样30只,除班戈湖品系用液氮保存样品外,其他3个品系取样前均饥饿处理48 h,以消除饵料藻类DNA对西藏拟溞个体DNA的影响。由于溞体微小,提取的单只DNA含量也很少,利用琼脂糖电泳检测很难发现条带,本试验中通过使用紫外分光光度仪测定样本吸收紫外辐射量来确定样品中DNA的含量和纯度。结果显示,绝大部分样品在260 nm波长下的吸光度与280 nm波长下的吸光度的比值为1.8~1.9,符合试验要求。
1.2.3 PCR扩增 引物设计参考 Raay等[6]公布的蚤状溞Daphnia pulex12S rRNA基因特异性引物片段,由宝生物工程 (大连)有限公司合成。

根据试验要求确定最佳PCR反应体系为:模板 DNA 2.0 μL,10×PCR Buffer 2.5 μL,MgCl21.5 μL,dNTP 0.5 μL,上、下游引物各 1 μL,Taq 酶(5 U/μL)0.2 μL,ddH2O 16.3 μL。PCR 反应程序为:72℃下预变性2 min;94℃下变性30 s,56℃下退火30 s,72℃下延伸2 min,共进行30个循环;72℃下再延伸10 min,4℃下保存。PCR扩增产物用15 g/L的琼脂糖凝胶进行电泳检测[6-7]。
1.2.4 PCR扩增产物的纯化及目的片段序列的测定 对PCR扩增产物效果好的样品进行纯化和测序,为提高序列精确度,使用正反链双向测序。
1.2.5 测序结果的比对与分析 使用Mega 4.0软件对测序结果进行同源性比较,并将比对的序列保存为Clustal格式,使用ClustalX 1.83软件进行比对,利用BIOEDIT进行图像处理,结果见图1。
将校准后的序列利用DNAMAN进行碱基组成分析,并计算各序列间未校正的遗传距离 (P距离)。对空位的处理采用完全删除 (complete deletion),同时采用Smith&Waterman(最佳比对)策略进行动态比对,得出西藏拟溞以及7个溞属物种序列的同源性。
用Mega 4.0软件去除非编码位点,用“bootstrap”自举检测1 000次表明各分支的置信度,构建NJ树。
2 结果
2.1 序列同源性的比较
测序结果 (图1)经校对后上传至GenBank,使用Blast软件进行序列相似性搜索,相似度最大的是西藏拟溞线粒体12S rRNA基因序列 (序列号为AY921475.1),相似度为96%;其次为多刺裸腹溞线粒体12S rRNA基因序列 (序列号为AF217136.2),相似度为95%。测序结果显示,4个品系西藏拟溞12S rRNA基因序列具有高度的保守性,每一品系30个个体的相似度分别为:纳木卡错品系99.3%、班戈湖品系98.9%、色林错品系99.1%、赛里木湖品系98.1%。
2.2 西藏拟溞12S rRNA基因序列的组成
对4个品系的西藏拟溞序列进行比对,结果表明 (表2):在西藏拟溞的4条12S rRNA基因序列中,A、T、G、C的平均含量分别为 31.9%、32.45%、17.33%和 17.95%,A+T的含量为64.35%,G+C的含量为35.28%,A+T的含量明显高于G+C的含量,符合节肢动物A、T含量高的特点。

图1 4个品系西藏拟溞线粒体12S rRNA基因片段序列Fig.1 Nucleotide sequence of mitochondrial 12S rRNA region in four populations of Daphniopsis tibetana Sars

表2 4个品系西藏拟溞12S rRNA基因序列核苷酸组成Tab.2 Nucleotide composition of 12S rRNA gene fragment sequences of our populations of Daphniopsis tibetana Sars %
4个品系西藏拟溞12S rRNA基因片段序列碱基转换/颠换数及不同位点碱基对出现的频率见表3。从表3可见,4个品系西藏拟溞12S rRNA基因序列在第2密码子和第3密码子共出现了3次AG转换,仅在第2密码子发生了1次TC转换。4个品系西藏拟溞基因序列中转换高于颠换,其中在第3密码子的转换率、颠换率分别为1.08%和0.54%,R值为2.0;在第2密码子的转换率、颠换率分别为1.59%和0;在第1密码子的转换率、颠换率分别为0.53%和0;所有序列的转换和颠换的比值R为6.0。在核苷酸替换中,颠换很少发生,转换主要发生在A与G之间。总的来说,4个序列之间并未体现出较大的单核苷酸多态性。
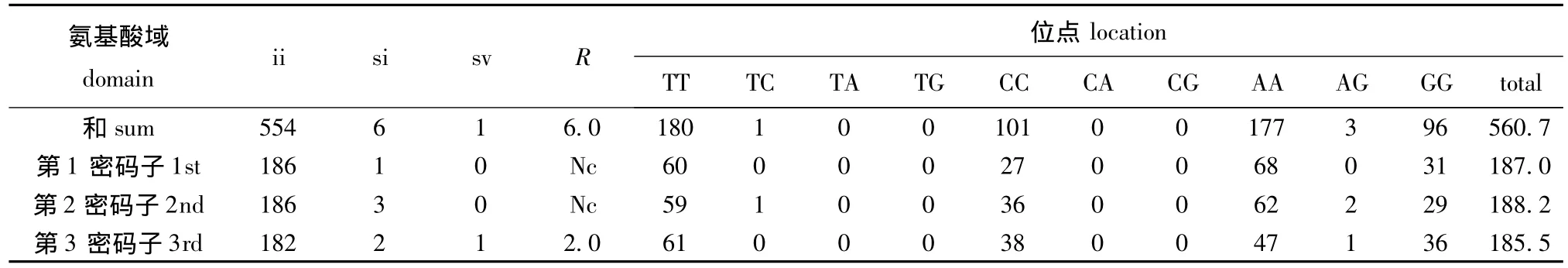
表3 4个品系西藏拟溞12S rRNA基因片段序列碱基转换/颠换数及不同位点碱基对出现频率Tab.3 Nucleotide pair frequencies and transition/transversion ratios of 12S rRNA gene sequences in four strains of Daphniopsis tibetana Sars
2.3 西藏拟溞群体间的遗传多样性
从GenBank数据库中选取不同地理品系的7种相似 序 列 溞 类Daphnialongicephala(AF217136.2)、Daphnia cf.bolivariAP-2005 from Spain(DQ116599.1)、Daphnia atkinsonifrom Israel(DQ116595.1)、Daphnia angulata strainVictoria(AF217137.2)、Daphnia angulata fromAustralia(AY921460.1)、Daphnia salinifera(AF217131.2)、Daphniopsis tibetana(AY921475.1)的12S rRNA基因片段,计算不同种间的遗传距离。从表4可见:属间的遗传距离明显大于属内的遗传距离,西藏拟溞与其他种类之间的遗传距离基本都在0.57以上,说明西藏拟溞与其他溞存在较大的遗传进化差异;而4个品系西藏拟溞中,班戈湖品系与纳木卡错品系间的遗传距离最小,仅为0.0126,班戈湖品系与赛里木湖品系间的遗传距离最大,为0.0199。
对11个种群溞类的12S rRNA进行序列同源性分析,结果表明 (表4):最大遗传相似性依然存在于西藏拟溞4个品系之间,纳木卡错品系与色林错品系以及纳木卡错品系与班戈湖品系的遗传相似性较高,达98%左右;其他7种溞中,属内的遗传相似性也明显大于属间的遗传相似性。
2.4 序列分析及系统树的构建
从图2可见:西藏拟溞品系与其他7种溞基本划分为2大分支,在第Ⅰ大分支中,D.tibetana显然与其他6种溞具有一定的差异;西藏拟溞所在的第Ⅱ大分支中,赛里木湖品系独立于3个西藏品系分化为一支,可以明显看出,由于西藏和新疆地理位置所造成的差异。
由所建的NJ树可以看出,其拓扑结构呈现出与遗传距离和同源性相吻合的信息。
3 讨论
3.1 西藏拟溞12S rRNA序列组成和分子进化分析
在西藏拟溞4个品系的12S rRNA序列中,表现出节肢动物门线粒体DNA序列A、T含量高的特征,其中A+T含量为64.35%,G+C含量为35.28%,A+T含量明显高于G+C含量。线粒体12S rRNA在生物进化过程中很保守,通常被用作属以上分类单位遗传多样性的鉴定指标。从各品系西藏拟溞的种间遗传距离以及相关NJ树可以看出,由于地理隔离所产生的种间差异很明显,本试验中所用的西藏拟溞采自于中国西藏和新疆,从进化树上看,赛里木湖品系和3个西藏品系的距离较远,但是虽然其序列间存在差异,也不能简单地归结于地理隔离的原因,更深入的研究有待于采集更广泛的西藏拟溞品系和开展对线粒体其它部分序列的测序。

表4 西藏拟溞和溞属7种枝角类12S rRNA基因片段序列种间遗传距离(P距离)及遗传相似性Tab.4 P-distance and homology matrix of 12S rRNA gene fragment sequences in 7 species between Daphnia and Daphniopsis tibetana Sars

图2 4个品系西藏拟溞与7种溞Nei's遗传距离的NJ树Fig.2 NJ trees of fours strains of Daphniopsis tibetana and 7 species of Daphnia based on Nei's genetic distance
对西藏地区3个品系的西藏拟溞进行分析,最为接近的是纳木卡错品系和班戈湖品系,它们构成一相近群。从3个湖泊的地理位置看,纳木卡错(30°46'N,90°52'E)至班戈湖 (31°43'N,89°29'E)的直线距离为120.7 km,至色林错 (31°50'N,89°00'E)的直线距离为213.4 km,班戈湖至色林错的直线距离为87.5 km,就纳木卡错品系而言,地理距离与其遗传距离有一定的联系。
西藏拟溞是冷水性广盐种,从盐度方面看,3个湖泊都为内陆盐水湖泊[7],纳木卡错盐度为16,班戈湖为20,而色林错为30,其盐度差异与遗传进化树也是相对应的。从pH方面看,班戈湖和纳木卡错都为9.5,色林错为9.3[8],其 pH差异与遗传进化树相符。赵文等[5]认为,纳木卡错和班戈湖的西藏拟溞可能来源于同一地理种群,分子进化距离及进化树已印证了这一观点,但其更具体的分类有待于对其他方面进一步地分析。从湖泊容积看,3个湖泊之中,色林错面积为1 658 km2,远远大于班戈湖 (80 km2)和纳木卡错 (12 km2),
水体容积大小对溞类的影响是间接的,可能通过不同的因子起作用,3个湖泊水体容积的大小关系与本试验对3个品系西藏拟溞的分子遗传的进化分类关系也基本相符。
3.2 关于Daphnia tibetana和Daphniopsis tibetana Sars
Sars在1903年最早发现西藏拟溞,将其归为拟溞,并命名为Daphniopsis tibetana。1936年Wagler将该溞归属于溞属,命名为西藏溞Daphnia tibetana,拒绝将其归属于拟溞属,自此对这种枝角类的命名便开始了分类上的争论[9]。赵文等[9]的研究表明,西藏溞和西藏拟溞的外部形态具有截然不同的特征,它们应分属于不同的种,最明显的区别为西藏溞的壳瓣后背角有壳刺,而西藏拟溞的壳瓣后背角钝圆不生成壳刺。
从传统意义上来看,本试验中所研究的Daphniopsis tibetanaSars与Daphnia tibetana属于高度相似种,但通过表4和图2的遗传距离以及遗传相似性来看,Daphnia tibetana与4个品系Daphniopsis tibetanaSars之间依然存在着较大的差异:平均遗传距离为0.3746,平均遗传相似性为71.6%。Daphnia tibetana和Daphniopsis tibetanaSars在线粒体12S rRNA上的差异从分子方面证明了Daphnia tibetana和Daphniopsis tibetanaSars应分属于不同的种,这与形态鉴定结果[9]相一致。
[1] Sars G O.On the crustacean fauna of central Asia.Pt.Ⅱ[J].Ann Mus Zool St Petersb,1903(8):171.
[2] Hedin S.Scientific results of a Jurney in Central Asia 1899 to 1902 Central snd West Tibet,3,Stockholm[C].Macmillan and Co,1907.
[3] Zhao W,Zheng M P,Xu X Z,et al.Biological and ecological features of saline lakes in northern Tibet[J]Hydrobiologia,2005,541:189-203.
[4] 赵文,王巧晗,郑绵平,等.西藏拟溞生物学的初步研究[J].大连水产学院学报,2002,17(3):209-214.
[5] 赵文,毕进红,韩婷婷,等.西藏拟溞遗传多样性的初步研究[J].大连海洋大学学报,2011,26(2):108-113.
[6] Raay T J V,Crease T J.Partial mitochondrial DNA sequence of the crustaceanDaphnia pulex[J].Current Genetics,1994,25:66-72.
[7] 郑绵平,向军,魏新俊,等.青藏高原盐湖[M].北京:科学技术出版社,1989.
[8] 赵文,霍元子,高敬.西藏拟溞营养成分的分析与评价[J].中国水产科学,2006,13(3):446-451.
[9] 赵文,王巧晗.西藏拟溞形态结构的再描述[J].大连水产学院学报,2005,20(3):165-173.
Molecular phylogeny of four strains of Daphniopsis tibetana Sars based on mitochondrial 12S rRNA gene sequences
ZHAO Wen,LI Rui
(Key Laboratory of Hydrobiology in Liaoning Province,College of Fisheries and Life Science,Dalian Ocean University,Dalian 116023,China)
The molecular phylogeny of four strains ofDaphniopsis tibetanaSars(NMKC,SLC,BG and SLM)were studied based on mitochondrial 12S rRNA gene sequences.The molecular evolution ofD.tibetanawere dicussed by means of 12S rRNA gene sequence of the corresponding sequences of 7 other species ofDaphniaCladocera from GenBank.The results showed that the mean concentration of A,T,G,C for 4 strainsD.tibetanaof 12 SrRNA gene sequences were 31.9%,32.45%,17.33%and 17.95%,respectively.The base pair concentrations were 64.35%in A+T and 35.28%in G+C.These were accordance with feature of higher A,T concentration in Arthropoda animals.There were significant differences betweenD.tibetanaand other species of cladocera,according to the results of genetic distance and using UPGMA cluster analysis in the Mega 4 software package procedures,indicating thatD.tibetanahas an unique position in the research of biology of Cladocera.The highest genetic similarity were between NMKC strain and SLC strain as well as between NMKC strain and BG strain,the genetic distance between NMKC strain and BG strain was the lowest in 0.0126,and highest was between SLM strain and BG strain in 0.0199.It was indicated that the interspecific difference was very significant,and the intraspecific difference is unconspicuous compared with other species ofDaphnia suchas.All above shows thatD.tibetanaSars has high levels of genetic diversity,and mitochondrial 12S rRNA can provide the reliable evolutionary information,and it can be used as molecular markers for phylogeny events.In addition,Daphniopsis tibetanaSars andDaphnia tibetanawere proved to be different species in molecular aspects by this method.
Daphniopsis tibetanaSars;12S rRNA;genetic diversity;phylogeny
S917.4
A
2095-1388(2012)04-0300-06
2011-12-20
国家自然科学基金资助项目 (31072210)
赵文 (1963-),男,博士,教授,博士生导师。E-mail:zhaowen@dlou.edu.cn
