BraSDG8 RNA干扰载体的构建及其对拟南芥的遗传转化
2012-01-29谢青轩虢婷婷
姜 玲,谢青轩,虢婷婷,阮 颖
(湖南农业大学生物科学技术学院,湖南 长沙 410128)
组蛋白甲基化作为一个重要的表观遗传学标记[1],对植物开花起调控作用[2]。拟南芥SDG8(SET DOMAIN GROUP 8)编码的是一种组蛋白甲基转移酶[3],约含有1 806个氨基酸序列,能特异催化组蛋白H3K36的双、三甲基化,是FLC表达所需的表观遗传记忆密码之一,在抑制植物早花过程中起重要作用[4]。功能缺失的拟南芥sdg8突变体[5-6]36的双、三甲基化水平显著降低,开花调控的关键转录因子FLC的转录激活受到抑制[7],进而植株表现为早花表型[8]。
BraSDG8是湖南农业大学植物发育与表观遗传调控研究室在白菜型油菜中克隆的拟南芥SDG8的同源基因,cDNA全长4 963 bp,编码1 654个氨基酸,结构域的组成与拟南芥SDG8完全一致,包含 AWS(associated with SET)domain、SET domain和Post SET domain 3个结构域,同属于ASH1家族,推测BraSDG8与SDG8具有相似的生物学功能[9]。笔者通过构建pFGC5941-BraSDG8 RNA干扰载体及农杆菌介导的拟南芥遗传转化,旨在为进一步研究白菜型油菜BraSDGG8的功能奠定基础。
1 材料与方法
1.1 材料
拟南芥(Arabidopsis thaliana)哥伦比亚生态型Col,大肠杆菌(Escherichia coli)DH5α,农杆菌(A-grobacterium tumefaciens)GV3101 和 pFGC5941 表达载体由湖南农业大学植物发育与表观遗传调控实验室保存;pMD-19 T Vector购自TAKARA公司。
1.2 方法
1.2.1 BraSDG8-1片段的扩增 比对拟南芥SDG8和BraSDG8 cDNA序列,根据保守区域设计引物,分别在5′端导入2个酶切位点(AscⅠ/BamHⅠ),预计扩增产物大小为443 bp,引物序列如下。
BraSDG8-1 F:5′-GGCGCGCCGGATCCCCAAG CATCAAGTGGCTCT-3′(NcoⅠ/XbaⅠ)
BraSDG8-1 R:5′-CCATGGTCTAGAGCACAGA GTTGATAACTTCTGAATCTGGT-3′
PCR扩增(Tm=57℃),回收片段,连接至pMD19-T载体构建成pMD19-T-BraSDG8-1,热激法转化大肠杆菌DH5α,将阳性菌落经PCR验证后送博尚生物公司测序,所得结果在The Brassica database(BRAD)上进行序列比对分析。
1.2.2 BraSDG8 RNA干扰载体的构建 如图1所示,BamHⅠ和XbaⅠ双酶切载体pMD19-TBraSDG8-1和pFGC5941,将目的片段反向插入至查尔酮内含子与Ocs终止子之间,构建表达载体p-BraSDG8-1;然后AscⅠ和NcoⅠ双酶切载体pMD19-T-BraSDG8-1和p-BraSDG8-1,将目的片段正向插入至35 S启动子与查尔酮内含子之间,构建BraSDG8 RNA干扰载体,记为p-BraSDG8。

将构建好的RNA干扰载体分别导入大肠杆菌DH5α和农杆菌GV3101中,测序验证。
1.2.3 拟南芥的遗传转化 采用浸花法将p-BraSDG8载体转化野生型拟南芥Col生态型。通过卡那霉素(浓度为40 mg/L)筛选获得抗性苗,改良的CTAB法提取叶片总DNA,PCR检测。引物序列如下。
35s825F:5′-ATCCCACTATCCTTCGCAAGACC CTT-3′
Intron825R:5′-TGACTCCATCTTATTCCCTCCG TTTC-3′
2 结果与分析
2.1 BraSDG8-1干扰片段的克隆与检测
克隆得到440 bp左右的目的片段,将目的片段连接至pMD19-T载体上,通过热激法转至大肠杆菌DH5α中,获得阳性菌落,PCR检测片段为440 bp左右,符合预期片段大小(图2)。
将目的片段测序,测序结果与RNA干扰片段进行序列比对,同源性达100%,MegAlign比对(图3),BraSDG8-1基因片段序列完整正确,可用于后

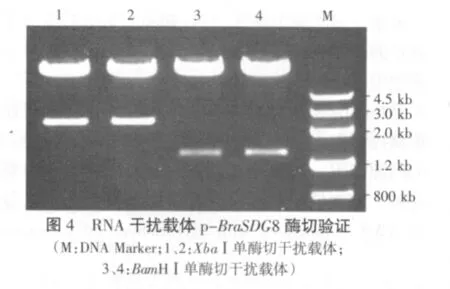
2.2 干扰载体的酶切验证
用BamHⅠ和XbaⅠ分别单酶切p-BraSDG8验证(图4),最终分别得到了1 300 bp和2 500 bp左右的条带,符合预期片段大小,表明BraSDG8 RNA干扰载体p-BraSDG8构建成功。
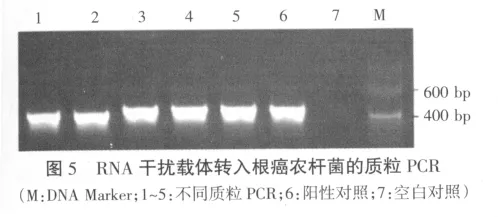
2.3 转化根癌农杆菌的质粒PCR检测
通过冻融法将干扰载体转至根癌农杆菌GV3101,提取阳性菌落质粒,进行质粒PCR检测,扩增得到的条带大小为400 bp左右(图5),与设计的目的片段大小443 bp相符,说明干扰载体已转化至根癌农杆菌中。
2.4 抗性苗的PCR检测
通过改良的CTAB法提取抗性苗总DNA,扩增结果如图6,检测得到1株阳性植株。片段大小与预期片段大小一致,为1 200 bp左右。

2.5 BraSDG8沉默的转基因植株的表型观察
在含有卡那霉素(浓度为40 mg/L)的MS培养基上,抗性苗生长健壮,子叶饱满而呈绿色,根系发育正常,而非抗性苗子叶呈黄色,个体较小,根长相对短小(图7)。与野生型拟南芥Col生态型相比,在营养生长期,转基因植株的莲座叶数目以及生长状态都没有明显区别;在生殖生长期,转基因植株的花期并未缩短,只是花苔数目和果荚数目变少,每个果荚的结实率降低。

3 讨 论
组蛋白甲基化作为一个重要的表观遗传学标记[10-11],是植物发育过程中的重要调节方式,其主要是由组蛋白甲基转移酶(histone methyltransferases,HMTs)来调控的[12-13]。其中,组蛋白赖氨酸甲基转移酶(histone lysine methyltransferase,HLMT)是一类含有 SET(Su(var),Enhancer of zeste,Trithorax)结构域的家族[14]。进化上保守的SET结构域包含约130个氨基酸序列,形成一个结状结构的酶催化中心[15],通过催化组蛋白甲基化来影响染色体的结构进而调控基因的表达[16]。早前的研究显示,在拟南芥中发现了约47个SET DOMAIN GROUP(SDG)基因[17],分别命名为 SDG1~SDG47,其中 SDG8,SDG25,SDG27和SDG30编码的SET结构域蛋白涉及植物开花时间的调控[18]。RNAi是研究基因功能的重要方法[19-20]。BraSDG8是拟南芥SDG8的同源基因,二者的相似性非常高,笔者通过构建干扰载体转化拟南芥,在克隆得到BraSDG8全长cDNA序列的基础上,选取其中一段保守片段构建RNA干扰载体pFGC5941-BraSDG8,并通过农杆菌GV3101介导,浸花法转化野生型拟南芥Col,获得了稳定遗传的转基因植株。但是与野生型拟南芥(Col)相比,该转基因植株开花时间、植株大小、莲座叶数目等表型并未有明显差异,其具体的原因有待进一步研究。
[1]杜婷婷,黄秋花.组蛋白赖氨酸甲基化在表观遗传调控中的作用[J].遗传,2007,29(4):387-392.
[2]夏志强,何奕昆,鲍时来,等.植物开花的组蛋白甲基化调控分子机理[J].植物学通报,2007,24(3):275-283.
[3]谢 萍,田春艳,张令强,等.组蛋白甲基转移酶的研究进展[J].遗传,2007,29(9):1035-1041.
[4]Zhao Z,Yu Y,Meyer D,et al.Prevention of early?owering by expression of FLOWERING LOCUS C requires methylation of histone H3K36[J].Nat.Cell Biol,2005,(7):1256-1260.
[5]孙万刚.拟南芥突变体构建方法及目的基因分离鉴定技术[J].安徽农业科学,2010,38(18):9433-9434.
[6]Xu P L,Zhan J P,Meng J J,et al.A rapid DNA extraction method for PCR detection of Arabidopsis thaliana[J].Agricultural Science&Technology,2010,11(3):41-42,155.
[7]李红梅,任 艳,魏艳丽,等.拟南芥开花时间的分子遗传调控[J].山东科学,2007,20(1):48-53.
[8]Xu L,Zhao Z,DongA,et al.Di-and Tri-but Not Monomethylation on Histone H3 Lysine 36 Marks Active Transcription of Genes Involved in Flowering Time Regulation and Other Processes in Arabidopsis thaliana[J].Mol.Cell.Biol,2008,20(4):1348-1360.
[9]谢青轩,彭 琦,刘春林,等.白菜型油菜BraSDG8基因的克隆与序列分析 [J].湖南农业大学学报(自然科学版),2011,37(4):372-375.
[10]李 想,张飞雄.组蛋白甲基化的研究进展[J].遗传,2004,26(2):244-248.
[11]田筱青,房静远.组蛋白甲基化研究进展[J].生物化学与生物物理进展,2006,33(6):511-516.
[12]He Y H.Control of the transition to flowering by chromatin modifications[J].Mol.Plant,2009,2(4):554-564.
[13]Martin C,Zhang Y.The diverse functions of histone lysine methylation[J].Nat.Rev,2000,6(11):838-849.
[14]Ng D W,Wang T,Chandrasekharan M B,et al.Plant SET domain-containing proteins:structure,function and regulation[J].Biochim.Biophys.Acta,2007,1769:316-329.
[15]Qian C,Zhou M M.SET domain protein lysine methyltransferases:Structure,specificity and catalysis[J].Cell Mol.Life Sci,2006,63(23):2755-2763.
[16]Sims RJ III,Nishioka K,Reinberg D.Histone lysine methylation:a signature for chromatin function [J].Trends Genet,2003,19:629-639.
[17]宋江华,曹家树.植物SET蛋白 [J].细胞生物学杂志,2007,(29):384-388.
[18]Baumbusch L O,Thorstensen T,Krauss V,et al.The Arabidopsis thaliana genome contains at least 29 active genes encoding SET domain proteins that can be assigned to four evolutionarily conserved classes[J].Nucl.Acids Res,2001,29:4319-4333.
[19]李兰岚,饶力群,范 适,等.RNAi技术及其在植物中的应用研究进展 [J].湖南环境生物职业技术学院学报,2005,11(4):313-318.
[20]左 刚,毛建平.转录水平siRNA介导的基因沉默[J].中国生物工程杂志,2005,25(12):78-81.
