大肠癌线粒体DNA诱导NIH3T3细胞D-环突变的作用与机制
2011-06-08姬宏莉姬宏娟马永全
姬宏莉,姬宏娟,马永全,杜 芳,王 辉
肿瘤的发生不仅取决于核内遗传物质,而且还与核外的线粒体DNA(mitochondrial DNA,mtDNA)密切相关[1,2]。mtDNA是细胞内唯一的核外遗传物质,具有可自主复制的DNA基因组。mtDNA是具有编码区和非编码区共16 569个碱基对的双链闭环分子,其中编码区含有37个基因,2种rRNA,22种tRNA和13种与线粒体氧化磷酸化有关的蛋白多肽基因;非编码区由 1120bp(16025-576)的 D环(D-Loop)构成[3]。多项研究发现mtDNA的D环可能为mtDNA突变的热点所在,但不同的实体肿瘤,有关该区突变的频率报道存在明显差异[4]。本实验通过将大肠癌细胞突变的mtDNA片断转染NIH3T3细胞,观察NIH3T3的突变和多态性变化,以便明确变异的mtDNA造成肿瘤发病的机制和作用。
1 材料与方法
1.1 材料 DMEM高糖培养基、0.25%胰蛋白酶购自美国Gibco公司;mtDNA提取试剂盒购自杭州V-gene生物科技公司;2×Pfu PCR MasterMix购自北京TianGene生物科技公司。Lipofection2000TM转染试剂购自Invintrogen公司。
1.2 实验用细胞株及质粒 ①NIH3T3细胞株购于南方医院病理生理教研室;②大肠癌mtDNA的重组表达载体:在 pcDNA3.1(+)载体 BamH I,Xho I位点间插入1200bp的突变的大肠癌mtDNA的D环区,命名为 pcDNA3.1(+)-Sw480 mtDNA,pcDNA3.1(+)-Lovo mtDNA,由本课题组构建。
1.3 方法
1.3.1 细胞培养 NIH3T3细胞在37℃,5%CO2饱和湿度的孵育箱中培养,培养基为含10%进口胎牛血清(GIBCO)的DMEM高糖培养基,隔天换液1次,每4~5 d用0.25%胰蛋白酶消化传代培养。
1.3.2 细胞转染与筛选 转染前24 h将处于对数生长期的NIH3T3细胞消化计数,调整浓度为1×105个/ml,接种于六孔板中,用Lipofection2000TM将pcDNA3.1(+)-Sw480 mtDNA、pcDNA3.1(+)-Lovo mtDNA分别导入NIH3T3细胞,具体转染操作过程参考 Invintrogen公司的Lipofection2000TM说明书及相关文献[5]。置于37℃,5%CO2饱和湿度的培养箱中培养24 h后更换无抗生素的含10%胎牛血清的DMEM 高糖培养液,48 h后用 G418 200 μg/ml筛选,2周后挑选单克隆细胞群落,用含G418 100 μg/ml的培养基继续培养。
1.3.3 mtDNA的提取 用杭州V-gene试剂公司的mtDNA提取试剂盒,采取膜裂解和膜选择结合DNA的技术分离mtDNA。
1.3.4 PCR法扩增mtDNA D-loop 参照mtDNA D-loop的全长设计两段引物并送上海博亚生物技术公司合成。Primer1 5’-tcgaattctttcatggggaagcaga tttgg-3’、Primer2 5’ -atggatccggaggtaagctacataaactg-3’,50 μl PCR 体 系 :mtDNA 4 μl、Primer1 (25 μmol/L)2 μl、Primer2 (25 μmol/L)2 μl、2×MasterMix 25 μl、ddH2O 17 μl。PCR 条件:预变性 94 ℃ 3 min、以下变性 94℃ 30 s、复性 56℃ 30 s、延伸 72℃1 min共33个循环,结束延伸72℃ 5 min。并将PCR产物5 μl行1%琼脂糖凝胶电泳。
1.3.5 PCR产物直接测序 转染不同重组质粒NIH3T3/Sw480-mtDNA、pcDNA3.1(+)-Lovo mtDN的序列与未转染的NIH3T3细胞及基因库数据(http://www.ncbi.nlm.nih.gov/entrez/viewer) 相比对。当转染大肠癌mtDNA重组载体的NIH3T3细胞D环的核苷酸序列与NIH3T3细胞相同,但与基因库记载的不同时,这种改变为多态性变化;当转染大肠癌mtDNA重组载体的NIH3T3细胞D环的核苷酸序列与基因库记载相同,但与NIH3T3细胞不同时,这种改变为多态性变化。发现突变后,为除外系统误差,用 PCR重新扩增线粒体 D环区,用不同的引物正反两个方向再次测序以证实突变确实存在。
2 结 果
2.1 转染细胞mtDNA D-loop的鉴定结果 取上述 PCR产物 5 μl行 1%琼脂糖凝胶电泳,80 V,45 min,可见一条清晰的1.2 kb目的基因条带 (图1)。用Vilber Lourmat凝胶成像图像分析仪分析图像。并送上海博亚生物技术公司正反双向测序。
2.2 转染细胞mtDNA D-loop测序变异结果 转染不同重组质粒 pcDNA3.1(+)-Sw480 mtDNA、pcDNA3.1(+)-Lovo mtDN的序列与未转染的NIH3T3细胞及基因库数据(http://www.ncbi.nlm.nih.gov/entrez/viewer)相比对。分别检测到转染pcDNA3.1(+)-Sw480 mtDNA 的 NIH3T3细胞线粒体D环共有11个位点变化,包括5个突变,6个多态性变化,转染 pcDNA3.1(+)-Lovo mtDN的NIH3T3细胞线粒体D环共有8个位点变化,包括3 个突变,5 个多态性变化(表1、2)。
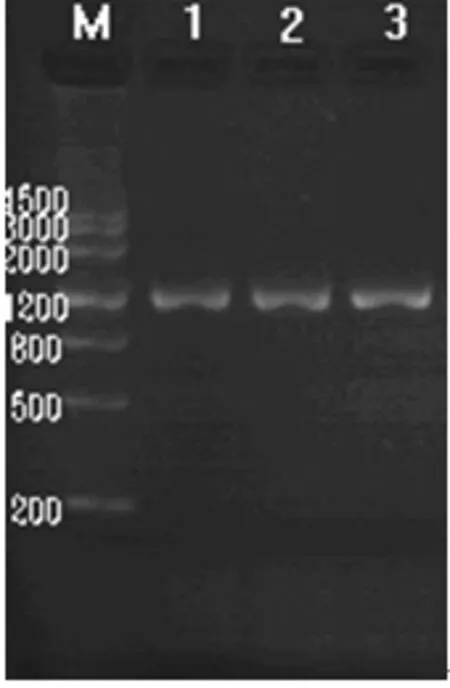
图1 转染前后NIH3T3细胞mtDNA D-loop电泳鉴定PCR结果

表1 转染pcDNA3.1(+)-Sw480 mtDNA的NIH3T3细胞线粒体D环区变化位点
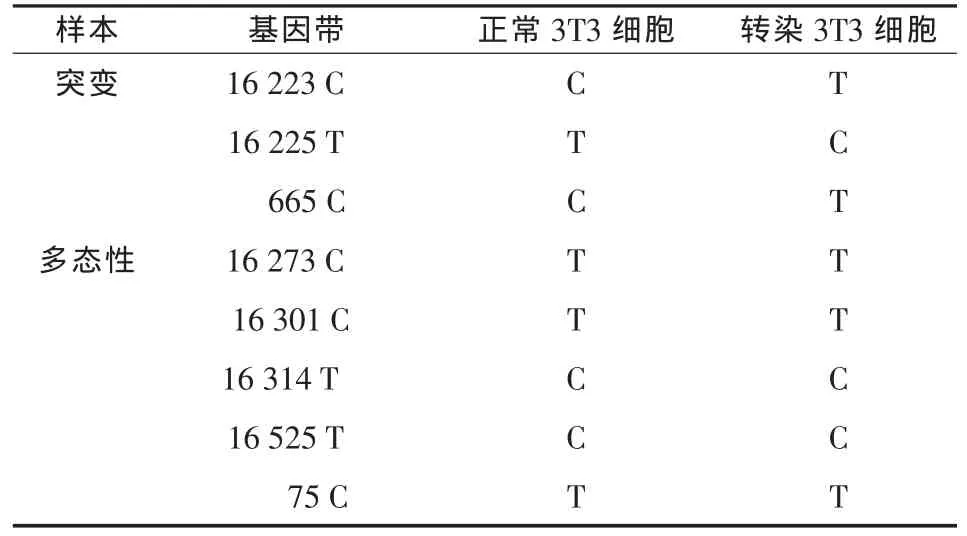
表2 转染pcDNA3.1(+)-lovo mtDNA的NIH3T3细胞线粒体D环区变化位点
3 讨 论
肿瘤的发生都是一个多因素作用,表现为多阶段的复杂过程。线粒体 DNA的突变在肿瘤发病中的作用和机制的研究还处在起步阶段。与cDNA相比,mtDNA更加脆弱,突变率更高。近来研究发现肿瘤的发生与线粒体DNA突变导致功能的改变密切相关。在大肠、肝、肺、胃等脏器以及血液系统的恶性肿瘤中,人们已经发现mtDNA的畸变[6-9]。Habano等[10]也报道了D环区的突变能引起蛋白质合成改变,影响线粒体的功能,进而对细胞的功能产生影响,推测线粒体基因改变将引起其功能异常,释放大量活性氧自由基,而这些活性氧自由基能造成核基因损伤,从而引起各种肿瘤的发生。以往的所有研究均集中在发现肿瘤细胞或组织中存在mtDNA突变的现象,而mtDNA突变对肿瘤发生发展的机制则涉及尚少。为进一步研究mtDNA突变与肿瘤发生发展的内在联系,笔者选取NIH3T3细胞为恶性转化的靶细胞,将突变大肠癌细胞的mtDNA转染NIH3T3细胞,观察转染细胞mtDNA的D环区的突变情况,以进一步明确mtDNA在肿瘤发生发展过程中的作用。
本研究共检测到9个位点变化,其中16 314位T→C,75位C→T,这2个位点变化在转染不同重组质粒的NIH3T3细胞和NIH3T3细胞中均检测到,考虑为多态性变化。在转染不同重组质粒的NIH3T3细胞中均检测到了与正常NIH3T3细胞和基因库记载不同的核苷酸变化,考虑为突变。在 8个突变位点中,除在转染重组质粒pcDNA3.1(+)-Sw480 mtDNA的NIH3T3细胞线粒体DNA D环区的16 437位和16 438位间检测插入序列G,另外的7个突变形式均为碱基置换。在转染pcDNA3.1(+)-Sw480 mtDNA的NIH3T3细胞中检测到16 223位 C→T,16 225位 T→C,16 237位 C→G,16 273位C→T,211位 A→T; 在转染 pcDNA3.1 (+)-Lovo mtDN的NIH3T3细胞中检测到 16 223位 C→T,16 225位C→T,665位C→T。因此,在转染不同重组质粒的NIH3T3细胞中,同时检测到16 223位C→T,16 225位T→C,检测到的相同的突变位点考虑为特征性改变,而这些特征性的改变目前认为可能和肿瘤的易感性有关,但是否致癌有待于进一步研究。
本研究发现,将大肠癌突变的线粒体 DNA导入到NIH3T3中,能够诱导NIH3T3细胞线粒体DNA的D环区发生突变和多态性的变化,从而证实大肠癌线粒体DNA的D环区是一具有高突变性的区域,在一定程度上具有诱导NIH3T3细胞恶性转化的倾向,然而在肿瘤发病中的作用和机制还需进一步研究。
[1]Li Yan,Weibing Song,Liu Hong,et al.Mitochondrial DNA from colotectal cancer cells promotes the malignant phenotype of NIH3T3 cells.Cell Biology International,2008,32(10):979-983.
[2]Astrid Lièvre,Caroline Chapusot,Anne-Marie Bouvier,et al.Clinical value of mitochondrial mutations in colorectal cancer.J Clin Oncol,2005,23:3517-3525.
[3]Daozhen Chen,Huiying Zhan.Study on the mutations in the D-loop region of mitochondrial DNA in cervical carcinoma.J Cancer Res Clin Oncol,2009,135(3):291-295.
[4]Lee HC,Yin PH,Lin JC,et al.Mitochondrial genome instability and mtDNA depletion in human cancers.Ann N Y Acad Sci,2005,1042:109-122.
[5]Suzuki R,Oda Y,Namai E,et al.Development of site specific gene delivery system with sonoporation.Yakugaku Zasshi,2008,128(2):187-92.
[6]Akouchekian M,Houshmand M,Hemati S,et al.High rate of mutation in mitochondrial DNA displacement loop region in human colorectal cancer.Dis Colon Rectum,2009,52(6):526-30.
[7]Daozhen Chen,Huiying Zhan.Study on the mutations in the D-loop region of mitochondrial DNA in cervical carcinoma.J Cancer Res Clin Oncol,2009,135(3):291-295.
[8]Ling-Juan Pang,Jian-Yong Shao,Xiao-Man Liang,et al.Mitochondrial DNA somatic mutations are frequent in nasopharyngeal carcinoma.Cancer Biology&Therapy,2008,7(3):198-207.
[9]Shih-Ching Chang,Pei-Ching Lin,Shung-Haur Yang,et al.Mitochondrial D-loop mutation is a common event in colorectal cancers with p53 mutations.Int J Colorectal Dis,2009,24(7):623-628.
[10]Habano W,Sugai T,Nakamura SI,et al.Microsatellite instability and mutation ofmitochondrial and nuclear DNA in gastric carcinoma.Gastroenterology,2000,118:8352-841.
