家蚕HSP70基因的电子克隆与序列分析*
2011-03-22房守敏伍春莲
房守敏 伍春莲
(西华师范大学生命科学学院,南充 637002)
热休克蛋白(heat shock protein,HSP)是应激后细胞内优先合成的一组蛋白质,在原核和真核生物中普遍存在,参与蛋白质的合成、折叠、装配、跨膜、转运以及变性蛋白质的清除等(Feder and Hofmann,1999;程维杰等,2008)。热激蛋白70(HSP70)是广泛存在且是一组在进化上高度保守的应激蛋白,在原核生物和真核生物体内都存在,并存在于所有的细胞区室和器官中,是当前研究较广泛的HSP家族。HSP70家族的成员数量在不同的生物有所不同,功能亦存在差异由分子量70 kD的热休克蛋白(诱导型)在正常细胞中水平较低,在应激状态下可显著升高(Mc-Millan et al.,2005;Bahrndorff et al.,2009)。大量研究表明机体细胞在受到各种应激,如高热,缺氧等有害应激时,产生的HSP70可以增强细胞对下一次有害损伤的耐受程度,维持细胞的正常功能代谢,提高细胞生存率(杨秉芬等,2009)。因此,HSP70对正常和非正常状态下的生物体的生存作用是非常负责和重要的,深入全面的了解HSP70对机体的作用将对动物的生产,人类生活,生物机体疾病预防起到重大影响。
在大多数生物体中,HSP70家族是由分子量为68、70、72及78 kD的热休克蛋白组成。它们的分子量相近,等电点为pH 5.2~6.3之间,几乎分布于每个细胞器中(Feder and Hofmann,1999;Johnston et al.,1998)。大量研究证明HSP70是生物体中少数最保守蛋白之一,无论是何种物种(原核细胞和真核细胞)的HSP70的氨基酸序列均至少有45%的同源性,越是相近的物种同源性越高(Feder and Hofmann,1999)。HSP70都有“卷线样”构象段,就象其它蛋白一样有绞链部分(如肌球蛋白),是ATP依赖的构像变化,这些丰富的ATP结合蛋白已成为所有热休克蛋白中最具特色和研究最多的之一(任宝波等,2005;王宇萍和蒋建东,2010)。
电子克隆(electronic cloning)是近年来伴随着基因组计划和ESTs计划而发展起来的基因克隆新方法,主要原理是利用日益发展的生物信息学技术,借助电子计算机的巨大运算能力,通过EST或基因组序列组装和拼接,进一步利用RT-PCR的方法快速克隆功能基因(Wang et al.,2007;林生等,2009)。本实验利用电子克隆技术,以黑腹果蝇HSP70蛋白的氨基酸序列在家蚕的数据库中进行查找、比对得到其同源的DNA序列和相应的EST,通过序列组装和拼接来获得家蚕的HSP70的cDNA序列,并利用生物信息学的软件来分析其序列信息,预测和分析其基因结构、蛋白质结构、等电点、分子量等,为进一步克隆和功能研究奠定基础。
1 材料与方法
1.1 数据库
黑腹果蝇Drosophilamelanogaster的HSP70(GenBank检索号AAG22148.2)氨基酸序列下载自美国国立生物技术信息中心NCBI(http://www.ncbi.nlm.nih.gov)。家蚕基因组数据库为西南大学SilkDB(www.silkdb.org/silkdb/)。
1.2 软件
同源性比对采用从NCBI下载的BLAST(basic local alignment Search tool)工具。EST s序列的拼接使用DNASTAR中的SeqMan软件。预测基因的外显子、内含子结构采用Sim4程序:http://gamay.univ-perp.Fr/analyse-seq/sim4。蛋白质序列翻译的在线工具:http://www.expasy.ch/tools/dna.html分子量MW/等电点pI的预测http://www.expasy.ch/tools/pi_tool.html。
1.3 方法
以黑腹果蝇HSP70的氨基酸序列与家蚕基因组预测的基因数据库(http://www.silkdb.org/silkdb/)进行tBlastn同源性检索,挑取相似高的BGIBMGA002381基因作为本研究的候选基因。以家蚕基因组数据库注释的CDS序列与家蚕ESTs数据库进行Blastn比对,获得的EST s序列用DNASTAR中的SeqMan进行拼接,将延伸的序列为种子序列应用Blastn程序检索家蚕EST s库,将检索到的EST s序列再次用DNASTAR中的SeqMan进行电子延伸,延伸步骤直到不能延伸为止。经SeqMan获得的拼接序列与家蚕基因组进行Blastn比对,从而找到其相应的基因组序列。提取该家蚕HSP70所对应的基因组序列,以Sim4软件分析基因结构。利用http://www.expasy.ch/tools/dna.html在线工具预测其氨基酸序列;利用http://www.expasy.ch/tools/pi_tool.htm l在线工具预测其蛋白质的分子量MW/等电点pI。
2 结果与分析
2.1 BmHSP70基因的电子克隆
以黑腹果蝇的HSP70蛋白作为检索序列与家蚕基因组数据库(http://silkworm.swu.edu.cn)进行tBlastn比对搜索,共获得了多个可能的热休克蛋白70同源基因,我们挑取其中与黑腹果蝇的HSP70相似较高的BGIBMGA002381进行了电子克隆。将家蚕基因组数据库中注释的BGIBMGA002381编码区序列(coding sequence,CDS)与NCBI中家蚕表达序列标签(expressed sequence tags,ESTs)数据库作Blastn比对检索,共检索得到20条可靠的ESTs(图1)。利用DNASTAR软件对家蚕HSP70的ESTs序列拼接和延伸,得到其长度为2 057 bp的cDNA序列。将拼接的cDNA在Softberry网站进行在线基因预测,结果表明该基因的编码区全长为1 950 bp,共编码649个氨基酸。
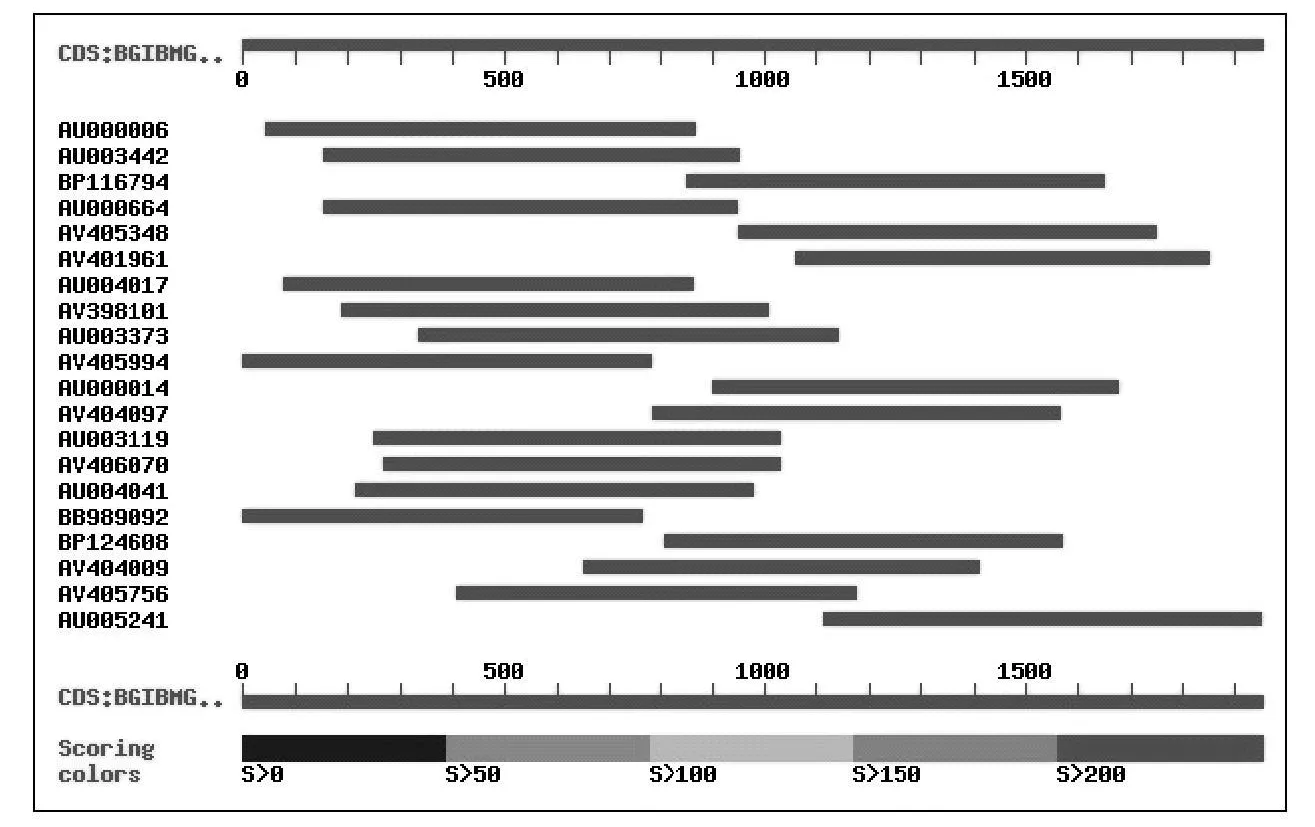
图1 BmHSP70基因的ESTs分析
2.2 BmHSP70基因的序列分析
将BmHSP70基因的ESTs拼接序列与家蚕基因组序列进行Blastn比对,获取BmHSP70基因的基因组序列,利用Sim4程序分析基因结构,结果如图2所示。BmHSP70基因的ESTs拼接序列含有2个外显子和3个外显子,内含子边界均符合标准的GT/AG规则。

图2 家蚕BmHSP70基因的结构简图
BmHSP70基因的核苷酸序列和推到的氨基酸序列如图3所示。经预测BmHSP70蛋白的分子量为71.18 kD,等电点pI为5.33。用蛋白质软件Antheprot分析家蚕HSP70蛋白,发现3个HSP家族的签名序列:IDLGTT YS(9-16残基),IFDLGGGTFDVSIL(197-210残基)和IVLVGGST RIPKVQK(334-348残基);Dank特征基序DLGTT-S-V(10-18残基),非细胞器基序RARFEEL(298-304残基),胞质HSP70特征基序GPTIEEVD(645-652残基)以及靠近C端的GGMP 4肽序列;2个糖基化位点NKSI和NVSA。根据所获得的序列符合HSP70家族特有的氨基酸序列特征,因此确认该序列是家蚕HSP70基因完整编码区cDNA序列(崔亚东等,2010)。
通过应用Clustral X和MEGA4.0软件,以家蚕HSP70与其它昆虫HSP70氨基酸序列建立建邻近进化树(Neighbor-joining method)。结果显示,BmHSP70与粉茎螟和二化螟HSP70的亲缘关系较近,在进化树中聚为一支,而且有很高的置信值(图4)。通过氨基酸相似性计算,表明家蚕BmHSP70与其他HSP70高度相似,特别是与粉茎螟和二化螟HSP70的相似性高达98.0%和97.2%,即使在进化树中表现出与其亲缘关系较远的美洲斑潜蝇、黑腹果蝇、埃及伊蚊、淡色库蚊和致倦库蚊的HSP70间相似性也高达75%左右。

图3 BmHSP70基因的核苷酸序列和推到的氨基酸序列

图4 家蚕和其他昆虫HSPs的NJ树
3 讨论
本研究在生物信息学分析的基础上,采用电子克隆技术,得到了家蚕HSP70的基因BGIBMGA002381。分析表明该基因的cDNA序列长2 057 bp,其编码区长1 950 bp,共编码649个氨基酸。该基因由2个内含子和3个外显子组成,其内含子边界均符合GT/AG规则。BmHSP70蛋白的分子质量为71.18 kD,等电点为5.33。序列分析表明BmHSP70蛋白具有HSP70保守的特征序列,而且具有大量的ESTs证据,表明该基因可能为家蚕体内有功能HSP70基因。BmHSP70与其他昆虫HSP70的氨基酸序列非常保守,特别是与粉茎螟和二化螟HSP70的相似性高达98.0%和97.2%。
本实验主要介绍了基于Internet网上生物信息资源对新基因全长cDNA的电子克隆策略,对于那些需要经常进行序列分析,或者分析规模较大的实验室,可以构建本地的生物信息学数据分析平台,把一些重复性的、可程序化的过程直接交由计算机完成,用户的主要精力就可转移到对分析结果进行后续分析及实验设计上,能节约大量的人力和物力。
生物信息学方法的应用使新基因全长cDNA克隆和分析的方法不断更新,朝着快速、经济、准确的方向发展,但鉴于生物大分子结构和功能的复杂性,许多分析软件的输出结果存在较大的偏差,因此利用生物信息学进行的“虚拟”克隆的结果尚需回到实验室进行验证。但是,这种分析方法为实验研究提供了重要的线索,对随后的研究起到了“事半功倍”的作用,避免走弯路,极大地提高了工作效率。可以相信随着基因组序列信息的日益丰富,计算方法和数据库的不断完善,生物信息学将在基因全长cDNA克隆和分析中扮演更加重要的角色。
[1]Bahrndorff S,Marien J,Loeschcke V,et al.Dynamics of heat-induced thermal stress resistance and hsp70 expression in the springtail,Orchesella cincta[J].Funct.Ecol.,2009,23(2):233-239.
[2]Feder M E,Hofmann GE.Heat-shock proteins,molecular chaperones,and the stress response:evolutionary and ecological physiology[J].Annu Rev Physiol,1999,61:243-282.
[3]Johnston J A,Ward C L,Kopito R R.Aggresomes:a cellular response to misfolded proteins[J].J Cell Biol,1998,143(7):1883-1898.
[4]McMillan D M,Fearnley S L,Rank N E,et al.Natural temperature variation affects larval survival,development and Hsp70 expression in a leaf beetle[J].Funct Ecol,2005,19(5):844-852.
[5]Wang H T,Chang J W,Guo Z,et al.In silico-initiated cloning and molecular characterization of cortexin 3,a novel specifically expressed in the kidney and brain,and well conserved in vertebrates[J].Int J Mol Med,2007,20(4):501-510.
[6]程维杰,李秋玲,孙延鸣,等.热休克蛋70(HSP70)研究进展[J].畜牧兽医杂志,2008,27(6):55-57.
[7]崔亚东,陆明星,杜予州.二化螟热休克蛋白70基因的克隆及热胁迫下的表达分析[J].昆虫学报,2010,53(8):841-848.
[8]林生,潘大仁,周以飞,等.果蔗Hsp90基因的电子克隆及序列分析[J].热带作物学报,2009,30(12):1824-1830.
[9]任宝波,王玉艳,王纯净,等.HSP70家族的分类及基因结构与功能[J].动物医学进展,2005,26(1):98-101.
[10]杨秉芬,孙启,鸿曹诚.热激蛋白70研究进展[J].生物技术通讯,2009,20(5):716-718.
[11]王宇萍,蒋建东.热休克蛋白70的结构和功能[J].中国细胞生物学学报,2010,32(2):305-313.
