羊鲍野生群体遗传多样性与分化的研究
2011-03-14
(集美大学 水产学院,水产生物技术研究所,福建 厦门 361021)
羊鲍(Haliotis ovina)属鲍科。其壳与肉皆可入药,且肉质细嫩、营养价值高,是中国重要的经济鲍类之一。羊鲍在中国主要出产于海南及西沙群岛海域。近年来由于酷渔滥捕及海区生境不断恶化等原因,羊鲍的自然资源受到严重破坏,这对原本就不大的羊鲍天然捕获量来说犹如雪上加霜。对羊鲍自然资源的保护已经刻不容缓。而对于羊鲍遗传背景的深入了解是制定科学有效的保护措施的必要前提。关于羊鲍遗传学方面的研究已有相关报道[1-4],但迄今尚未发现采用随机扩增片段长度多态性 DNA 标记(RAPD)技术,对产于中国的羊鲍野生群体进行遗传多样性分析的报道。
RAPD标记技术诞生于1990年,由于其具有操作简单、成本低廉且无需专门设计扩增引物等特点而被广泛应用于遗传图谱的构建、基因定位、遗传多样性分析、物种或品种鉴定、亲缘关系确定及良种选育等诸多遗传学研究领域[5-9]。在经济贝类的群体遗传学分析方面,RAPD技术已成功运用于缢蛏(Sinonovacula constricta)、皱纹盘鲍(Haliotis discus hannai)、杂色鲍(Haliotis diversicolorReeve)、大连湾牡蛎(Crassostrea talienwhanensis)、马氏珠母贝(Pinctada martensii)、文蛤(Meretrix meretrix)等物种的研究[10-15]。表明 RAPD技术既能够获得足够多的分子标记来用于分析不同群体的遗传多样性,又可通过数据处理来判断不同群体间的遗传分化程度[13]。
本研究采用 RAPD技术,对中国羊鲍野生群体的遗传多样性与遗传分化进行分析探讨,以期为中国羊鲍种质资源的保护、自然资源的可持续利用及良种选育提供科学理论依据。
1 材料与方法
1.1 材料
实验所用野生羊鲍分别采自中国海南省的英州、安游及亚龙湾,每个群体随机取样 30个,取得的新鲜样品加冰保存,运至实验室后放入-40℃冰箱保存至分析。
1.2 基因组DAN的制备
DNA提取主要参照传统的酚/氯仿抽提法,略做修改。具体步骤如下:每个个体取足部肌肉组织50~100 mg,剪碎放入1.5 mL离心管中。加入700 μL由 2×CTAB(10 mmol/L Tris-HCl,pH8.0;0.5 mol/L EDTA,pH8.0;1.4 mol/L NaCl;0.02 g/mLCTAB),0.2 μL β-2 巯基乙醇及 15μL 蛋白酶 K(20 g/L)组成的CTAB消化液于振荡器上振荡均匀,55℃消化过夜。待肌肉组织完全消化后加入 700 μL氯仿 :异戊醇(24 :1)混合液,颠倒混匀15 min,离心 10 min(12 000 r/min)。离心后吸取上清液至另一个新管。再加入600 μL 苯酚 :氯仿 :异戊醇(25 :24 :1)混合液进行抽提,提取后的上清液再加入 600 μL 氯仿 :异戊醇(24 :1)混合液抽提一次,加入 800 μL经-20℃预冷的异丙醇,颠倒混匀,-20℃存放 2 h后离心 10 min(10 000 r/min),产生白色沉淀,弃上层液体,保留离心管底部沉淀,再先后用经-20℃预冷的 1mL70%与1mL 100%的乙醇缓慢颠倒洗涤 DNA沉淀,各自离心 10min(10 000 r/min),弃上层液,保留沉淀。将DNA样品置于空气中晾干;干燥后的 DNA样品加入100 μL ddH2O,放入4℃冰箱充分溶解。用0.8%琼脂糖电泳检测溶解后的DNA的质量。
1.3 RAPD扩增反应
RAPD反应条件参照Williams 等的方法[16]。反应总体积为 25 μL,其中 1×Buffer 2.5 μL,MgCl2(25 mmol/L)2.5 μL,dNTP(2.5 mmol/L)1 μL,Taq 酶(5U/μL)0.3μL,引物(5 mg/L)3μL,模板 DNA 50 ng,其余的部分用ddH2O补足。于PCR仪上进行扩增反应。PCR反应条件:首先经94℃预变性4 min,后进入45个PCR循环:即94℃变性1min,35℃退火1min,72℃延伸1min。最后再经72℃继续延伸10 min。扩增产物用2.0%琼脂糖凝胶电泳分离,EB染色,紫外投射仪检测并拍照记录。
1.4 数据处理
对电泳后凝胶上清晰可见的扩增条带进行记录,在同一迁移位置有带的记为“1”,无带则记为“0”,列出“0”,“1”矩阵,输入统计软件 POPGENE 1.32 进行统计分析,获得羊鲍 3个群体的多态位点百分数(P)、Shannon’s 信息指数(I)、Nei’s 基因多样性指数(H)、群体间的遗传距离(D)、群体间基因分化系数(GST)等遗传学参数。并由程序 NEIGHBOR中的 Neighborjoining构建羊鲍3个群体的聚类分析图。
2 结果
2.1 引物筛选与RAPD扩增结果
本研究采用40个随机引物进行RAPD扩增实验,从中优化出14个能扩增出清晰条带、并可重复的有效引物(表1,引物S97对3个羊鲍野生群体的RAPD扩增图谱见图1)。用这14个引物在羊鲍3个野生群体 90个个体中共检测到72个位点。其中英州群体的多态位点数最多为65个,安游和亚龙湾群体的多态位点数则分别为61和62个。

表1 14个多态随机引物的DNA序列及其扩增结果Tab.1 Sequences and amplification results of 14 random primers
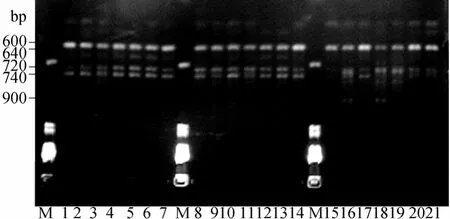
图1 引物S97对3个羊鲍野生群体的RAPD扩增图谱Fig.1 RAPD amplification bands of three H.ovina stocks with primer S97
2.2 遗传多样性
羊鲍 3个野生群体的多态位点百分数(P)为84.72%~90.28%,平均为 87.04%;Shannon’s信息指数(I)为 0.400~0.456,平均为 0.433;Nei’s 基因多样性指数(H)为0.262~0.305,平均为0.286 (详见表2)。
2.3 遗传分化与聚类分析
羊鲍3个野生群体间的遗传距离(D)为0.0504~0.0731。其中英州群体与安游群体之间的遗传距离最大为 0.0731,英州群体与亚龙湾群体的遗传距离最小为 0.0504,而安游群体与亚龙湾群体的遗传距离居中为 0.0577。群体间的遗传分化系数(GST)为0.0976。根据各群体间的遗传距离应用 NJ法得到 3个群体的聚类分析图(图2)。

表2 羊鲍3个群体的遗传多样性参数Tab.2 Indexes of genetic diversity of three H.ovina stocks by RAPD
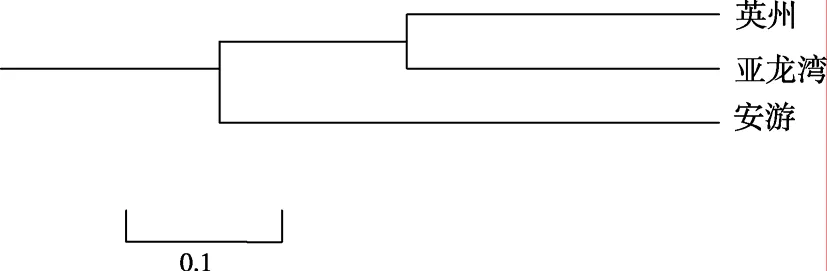
图2 用NJ法得到的羊鲍3个野生群体的聚类分析图树Fig.2 NJ cluster analysis dendrogram of three H.ovina stocks
3 讨论
遗传多样性是生物多样性的核心与基础,是生物种群内和种群间可遗传的变异。种群遗传变异的重要性在于它能够使进化的变化得以发生,而这些变化的速率与现有的遗传多样性的数量成正比[17]。遗传变异作为物种自身生存和未来进化的资源,不但能够给种群带来适合度方面的优势,而且同样有利于人工育种。多态位点百分数(P)、Shannon’s信息指数(I)、Nei’s基因多样性指数是群体遗传多样性度量的主要参数。在本研究中,3个羊鲍野生群体的多态位点百分数(P)为 87.04%,Shannon’s信息指数(I)为0.433,Nei’s基因多样性指数(H)为0.286。将此结果与同样采用 RAPD技术所获得的一些经济贝类野生群体的遗传多样性参数均值进行比较,羊鲍群体的遗传多样性与文蛤(P= 86.5%,H=0.1935)[15]、栉孔扇贝(Chlamys farreri)(P= 85.46%,I=0.243)[18]、西施舌(Coelomactra antiquata)(P=78.97%,I=0.349,H=0.309)[19]等物种的遗传多样性相当;远远高于大珠母贝(Pinctada maxima)(P= 50.0%,I=0.1170)[20]、皱纹盘鲍(P=50.0%,I=0.258)[21]等物种的遗传多样性;而仅略低于青蛤(Cyclina sinensis)(P= 92.07%,I=0.558,H=0.377)[22]的遗传多样性。由此可以判断,羊鲍在海洋经济贝类中具有较高的遗传多样性水平,种质资源状况良好。这一结论与采用 AFLP技术所获得的研究结果一致[4]。根据以往的研究报道,在经过几个世代的人工养殖后,物种的遗传多样性会有所降低[23-25],羊鲍虽为中国的经济鲍类之一,但至今未开展大规模的人工养殖,这就避免了羊鲍养殖群体对野生群体的遗传污染,从而间接地保护了羊鲍自然群体的遗传多样性。
Nei[26]认为群体间遗传分化系数GST的值可以从0到1,当各群体间几乎没有分化时,GST的值接近0,因为各群体的所有等位基因频率几乎相同。在本研究中,羊鲍群体间的遗传分化系数GST为0.0976,显示其中90.24%的遗传分化存在于群体之内,9.76%的遗传分化存在于群体之间,因此羊鲍的群体内变异为主要变异,群体间的遗传分化较小。遗传距离是判断遗传变异的另一尺度。本研究的 3个羊鲍群体间的遗传距离介于0.0504~0.0731之间,远小于大珠母贝[20]、马氏珠母贝[14]等物种群体间的遗传距离,而与文蛤[15]、缢蛏[10]等物种群体间的遗传距离相当。物种分化实际上是生物自身对异质化环境适应的结果。对于水产动物来说,发生遗传分化的原因主要包括地理隔离与水文状况差异以及人工干涉等[12-14]。具体到本研究的羊鲍群体来说,三个群体的采集地之间的地理距离较小,水文状况差异不大。此外,虽然羊鲍为营附着生活的贝类,成体的活动范围较小,但在羊鲍的生活史中,有营浮游生活的幼体阶段,这就为群体之间发生基因交流创造了可能。刘必谦等[13]也认为,虽然对于贝类来说,基因流动的范围是有限的,但由于进化是一个漫长的过程,在此过程中相距不远的群体之间的基因也有可能慢慢扩散,从而降低了各群体间的遗传差异。从本研究的聚类分析结果来看,英州群体与亚龙湾群体之间的亲缘关系相对较近,而亚龙湾群体与安游群体之间的亲缘关系相对较远,这可能是由于安游群体与亚龙湾群体的采集地点都是较为封闭的海湾,从而在一定程度上阻碍了两群体之间的基因交流所致。
一般认为,RAPD技术的多态性检测程度要高于等位酶技术。这是因为从理论上讲等位酶技术只能分析部分功能基因(外显子)的情况,而对其余部分的基因无法分析,而 RAPD技术可以分析研究对象的整个基因组的情况,从而能够获得更多的遗传信息。在本次采用RAPD技术的研究中3个羊鲍野生群体的多态位点百分数(84.72%~90.28%),远高于采用等位酶技术获得的 3个羊鲍野生群体的多态位点百分数(27.27%~54.55%)[2],这一比较结果再次验证了上述理论。很多实验证实,RAPD标记在单位分析中(每引物或引物对)检测到的多态位点数显著低于AFLP标记[27-28]。而从本研究中可以看出,优化较多数量的引物用于实验检测,便能获得足够的用于分析的分子标记,从而弥补 RAPD标记在该方面的不足。虽然影响RAPD反应的因素较多,但如果在实验中采用标准化的反应条件,注重个体之间、群体之间实验操作的同步性与对比性,保证DNA的提取、浓度检测及PCR反应各成分的一致性和电泳条件的统一性,并通过多次重复反应,就可以获得稳定而真实的实验分析结果。此次 RAPD技术的分析结果与之前的 AFLP分析在羊鲍 3个群体的遗传多样性水平、群体间的遗传分化程度的判断及群体间的聚类分析结果等都显示出高度的一致性[4]。由此不难看出,RAPD分析技术在遗传学研究领域仍不失为一种简便有效的分子标记。
[1]Klinbunga S,Pripue P,KhamnamtongN,et al.Genetic diversity and molecular markers of the tropical abalone(Haliotis asinina)in Thailand[J].Mar Biotechnol,2003,5:505-517.
[2]Li Z B.The genetic diversity and differentiation ofH.ovinapopulations by allozyme analysis[C]//13th International Congress on Genes,Gene Families and Isozymes-ICGGFI.Bologna:Medimond,2006:201-207.
[3]黎中宝,Appleyard Sharon A,Elliott Nicholas G.羊鲍(Haliotis ovina)和耳鲍(H.asinina)MtDNACOⅠ和COⅡ基因片段序列的比较研究[J].海洋与湖沼,2008,39(2):168-173.
[4]Li Z B.The genetic diversity and differentiation ofHaliotis ovinaby AFLP [C]//2009 International Conference on Environmental Science and Information Application Technology.Hong Kong:Engineering Technology Press,2009:7:39-42.
[5]Bassam B J,Castano-Anolles G.DNA amplification fingerprinting of bacteria[J].Appl Microbiol Biotech,1992,77:321-341.
[6]Comincini S,Sironi M,Bandi C,et al.RAPD analysis of systematic relationships among the Cerridae[J].Heredity,1996,76:215-221.
[7]Wilkie S E,Isaac P G,Slater R J.Random amplified polymorphic DNA (RAPD) markers for genetic analysis inAllium[J].Thero Appl Genet,1993,86:497-504.
[8]黎中宝.应用RAPD技术研究4种鲍的亲缘关系[J].中国生态农业学报,2004,12(4):60-63.
[9]谢浩,陆仁后,项超美,等.利用 RAPD技术对三种绒螯蟹亲缘关系的研究[J].水生生物学报,1999,23(2):120-126.
[10]李成华,李太武,宋林生,等.4个缢蛏群体遗传结构的RAPD分析[J].水产科学,2004,23(12):26-28.
[11]陈静,刘志刚,王伟,等.马氏珠母贝 4种壳色选育系 RAPD反应体系优化[J].现代农业科学.2009,16(3):1-4.
[12]李太武,杨文新,宋林生,等.皱纹盘鲍(Haliotis discus hannai Ino)和杂色鲍(Haliotis diversicolorReeve)遗多样性的研究[J].海洋与湖沼,2003,34(4):445-448.
[13]刘必谦,戴继勋,喻子牛.RAPD标记在大连湾牡蛎种群研究中的应用[J].青岛海洋大学学报,1998,28(1):82-87.
[14]王爱民,阎冰,叶力,等.三个野生种群马氏珠母贝遗传多样性的 RAPD分析[J].农业生物技术学报,2003,11(2):163-168.
[15]阎冰,邓岳文,杜晓东,等.广西地区文蛤的遗传多样性研究[J].海洋科学,2002,26(5):5-7.
[16]Williams J G K,Kubelik A R,Livak J,et al.DNA polymorphisms identified by arbitrary primers are useful as genetic markers[J].Nucleic AcidsRes,1990,18(22):6 531-6 535.
[17]克里施纳默西K V.生物多样性教程[M].张正旺,主译.北京:化学工业出版社,2006:9-10.
[18]刘亚军,孔晓瑜,喻子牛.栉孔扇贝(Chlamys farreri)自然群体遗传多样性的RAPD分析[J].海洋与湖沼,2006,37(4):289-295.
[19]尤仲杰,包永波,张爱菊.中国沿海西施舌5个自然群体形态差异和 RAPD分析[J].海洋学报,2007,29(3):99-103.
[20]苏天凤,朱彩艳,江世贵.海南岛大珠母贝遗传多样性分析[J].海洋科学,2005,29(8):20-22.
[21]李莉,常林瑞,孙振兴,等.皱纹盘鲍遗传多样性的RAPD 分析[J].海洋水产研究,2006,27(1):59-63.
[22]白胡木吉力图,高悦勉,姚红伟.青蛤北方3个群体遗传多样性分析[J].水产科学,2008,27(9):487-489.
[23]宋林生,李俊强,李红蕾,等.用 RAPD技术对中国栉孔扇贝野生种群与养殖群体的遗传结构及其遗传分化的研究[J].高技术通讯,2002,7:83-86.
[24]方展强,陈军,郑文彪,等.鳜野生群体与养殖群体的RAPD 分析[J].大连水产学院学报,2005,20(1):16-19.
[25]宋林生,相建海,李晨曦,等.日本对虾野生种群与养殖种群遗传结构的 RAPD标记研究[J].海洋与湖沼,1999,30(3):261-266.
[26]Nei M.F-statistics and analysis of gene diversity in subdivided populations[J].Ann Human Genet,1977,41:225-233.
[27]Thierry M L,Panaud O,Toupance B,et al.Assessment of genetic relationships betweenSetaria italicaandits wild relativeS.viridisusing AFLP markers[J].Theor Appl Genet,2000,100:1 061-1 066.
[28]Nakajima Y,Oeda K,Yamamoto T.Characterization of genetic diversity of nuclear and mitochondrial genomes inDaucusvarieties by RAPD and AFLP[J].Plant Cell Reports,1998,17:848-853.
