不同基因型大豆GS1基因的克隆与分析
2011-02-20陈丽华刘丽君刘页丽裴宇峰
陈丽华,刘丽君,刘页丽,裴宇峰,祖 伟*
(1.国家农业标准化监测与研究中心,哈尔滨 150036;2.东北农业大学农学院,哈尔滨 150030;3.黑龙江农垦科研育种中心,哈尔滨 150090)
就植物的生长和发育而言,氮素的同化是个十分重要的生理过程。其中,无机氮必须同化为谷氨酰胺和谷氨酸等有机氮才能被植物体所吸收和利用。谷氨酰胺合成酶(Glutamine synthetase,GS)是高等植物氮代谢中十分重要的酶,它和谷氨酸合成酶联合作用,催化氨的同化,使其转变成Gln和Glu,在高等植物体内含氮有机物的生物合成中作为氮的供体[1]。高等植物的种子、叶、根、根瘤和果实等器官中分布着多种GS的同工酶。在植物叶片中存在两种GS同工酶:一种定位于细胞质部分,称为胞液(胞质)型GS-GS1;另一种定位于叶绿体部分,称为叶绿体型GS-GS2[2]。在此前氮代谢规律的研究中大多将GS酶活性作为主要的测定指标[3-4],而缺乏不同基因型大豆在GS基因结构和转录水平的研究。本试验通过对不同基因型大豆GS1基因的克隆和分析,为不同基因型大豆GS1基因的功能分析和转录水平的定量分析奠定基础。
1 材料与方法
1.1 不同基因型大豆
高油大豆品种:垦丰 9(KF9)、东农 46(DN46);高蛋白大豆品种:东农42(DN42)、黑农35(HN35);秣食豆(MSD);野生大豆(WS)。其中KF9、DN46、HN35、DN42由东北农业大学农学院提供;秣食豆由东北农业大学动物科学技术学院提供;野生大豆由吉林农业科学院提供。
1.2 总RNA的提取
取不同基因型大豆幼苗幼嫩叶片,采用上海生工UNIQ柱式Trizol总RNA提取试剂盒提取总RNA。
1.3 DNA提取
取不同基因型大豆幼苗幼嫩叶片,采用CTAB法提取DNA[5]。
1.4 反转录
采用上海生工AMV第一链cDNA合成试剂盒。
1.5 目的基因的PCR扩增
根据GenBank上的大豆GS1基因序列,应用Primer Premier 5.0软件设计基因特异引物。以基因组DNA或反转录产物为模板,使用LA Taq酶扩增基因序列。
GS1基因引物:
Sense:5′AAGGGTCTTTGCTTGATTTTG 3′
Antisense:5′AACGAGGGAAAGGAATAGAAG3′
1.6 PCR产物的电泳回收
参照上海华舜生物工程有限公司回收试剂盒说明书完成。
1.7 PCR产物的克隆
参照pMD18-T vector试剂盒说明进行连接。大肠杆菌感受态的制备、大肠杆菌的转化、质粒DNA提取均采用常规方法[6]。
1.8 序列分析
应用Clustal W软件对所获得的基因序列进行比较分析,分析不同基因型大豆GS1基因的差异,提取共有序列,并构建分子进化树。应用DNAMan软件的Translation功能将基因序列转变成蛋白质序列。应用NetGene2软件预测基因外显子和内含子。应用NCBI在线spidey软件和DNAMan软件分析基因外显子和内含子。
2 结果与分析
2.1 不同基因型大豆GS1基因cDNA序列的克隆
通过RT-PCR扩增,在不同基因型大豆中均可扩增出约1300bp的目的大小GS1基因片段(见图1)。回收1 300 bp的目的大小GS1基因片段,与pUCm-Tvector连接,转化大肠杆菌JM109感受态细胞。将筛选到的阳性克隆送交测序。
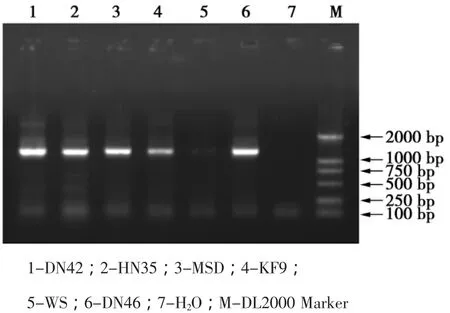
图1 GS1基因的扩增Fig.1 Amplification of GS1 gene
2.2 不同基因型大豆GS1基因序列分析
经测序获得了6个不同基因型大豆的GS1基因序列,应用Clustal W对比程序对这6个基因序列进行比较分析,分析结果见图2。从图2可以看出,来自不同基因型大豆的GS1基因序列相似性很高,因此能够从中提取共有序列,并据此设计Real-time PCR引物,检测不同基因型大豆GS1基因的表达规律。但是这些GS1基因序列间仍存在显著的差异,有许多单碱基突变和插入缺失突变。其中非编码区的差异显著高于编码区,并且全部的插入缺失突变都位于非编码区,不会造成移码突变。这说明非编码区在进化过程中的选择压力小于编码区,更易于积累基因突变。编码区内的碱基置换可能改变其所编码的氨基酸序列或影响翻译效率,进而造成酶活性和酶量的差异。由此导致不同基因型大豆氮代谢的差异,最终导致蛋白品质上的差异。值得关注的是,在突变集中的3′端非编码区,东农42和半野生大豆GS1基因序列表现出极高的相似性,暗示着二者之间可能有着较近的亲缘关系。

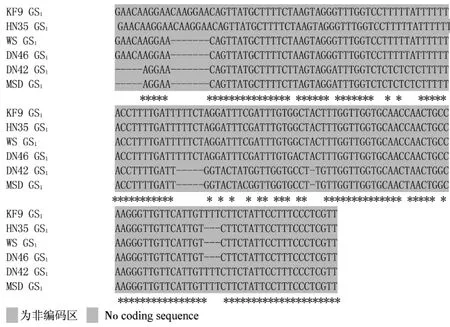
图2 不同基因型大豆GS1基因序列比对结果Fig.2 Alignment of GS1 genes sequences from different genotype soybeans
根据不同基因型大豆GS1基因序列,应用Clustal W软件构建了GS1基因分子进化树(见图3)。从分子进化树中可以看出,东农42、半野生大豆、东农46 GS1基因序列进化距离较近。
2.3 不同基因型大豆GS1 蛋白质序列分析
提取不同基因型大豆GS1基因的编码区序列(CDS),应用DNAMan软件的Translation功能将其转变成蛋白质序列,应用ClustalW对比程序对这6个蛋白质序列进行比较分析,分析结果表明不同基因型大豆GS1蛋白质序列具有很高的相似性(比对结果图略)。并且NCBI保守结构域数据库(CDD)检索结果表明,不同基因型大豆GS1蛋白质序列都具有两个保守的结构域,催化结构域(Gln-synt_N)和结合结构域(Gln-synt_C),说明它们都具有催化功能(见图4)。
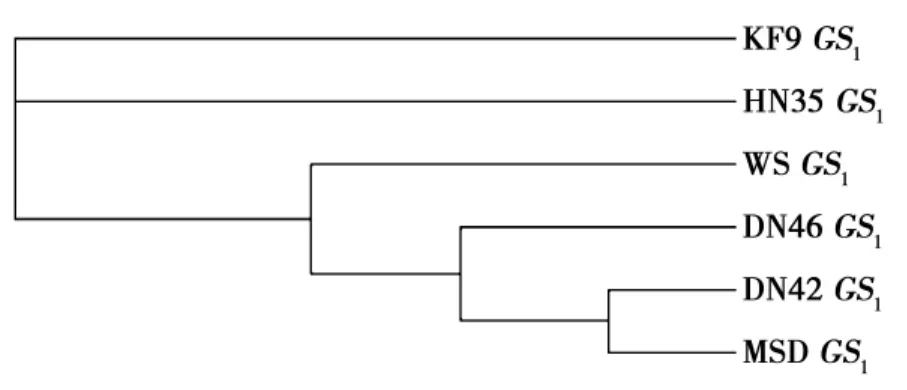
图3 不同基因型大豆GS1基因分子进化树Fig.3 Evolutionary tree of GS1 genes from different genotype soybeans
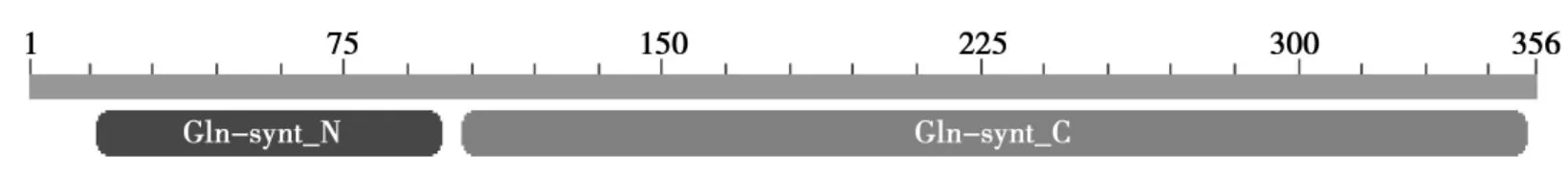
图4 GS1 蛋白质保守结构域分析Fig.4 Analysis of conserved domains for GS1 protein
应用Clustal W软件构建了这6个蛋白质序列的分子进化树(见图5),从中可以看出东农46和东农42 GS1蛋白质序列进化距离较近,垦丰9和黑农35 GS1蛋白质序列进化距离较近。尽管东农42和半野生大豆GS1基因序列进化距离较近,但是在蛋白质序列的分子进化树中二者相距较远,导致这一差异的原因是二者在GS1基因非编码区的高度相似性。
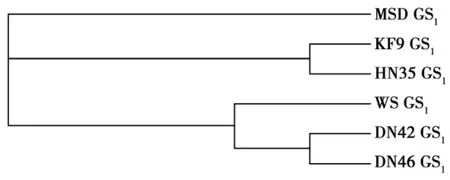
图5 不同基因型大豆GS1 蛋白质分子进化树Fig.5 Evolutionary tree of GS1 protein from different genotype soybeans

图6 GS1基因基因组序列的PCR扩增Fig.6 PCR amplification of genomic sequence of GS1 genes
2.4 东农42 GS1基因结构分析
以东农42基因组DNA为模板,用GS1基因特异引物进行PCR扩增,电泳结果如图6所示,在49.8和52.8℃均可扩增出约4 kb的特异条带。
经测序获得了东农42 GS1基因的基因组序列,应用NetGene2软件预测基因外显子和内含子,预测结果显示该基因具有12个外显子和11个内含子(见图7)。应用DNAMan软件的Dot metrix功能对东农42 GS1基因基因组序列和cDNA序列进行比对,可以很直观地看出东农42 GS1基因cDNA序列完全包含在其基因组序列中,二者形成12个匹配区段,说明东农42 GS1基因基因组序列具有12个外显子(图略)。应用NCBI在线spidey软件对东农42 GS1基因基因组序列和cDNA序列进行比对,同样可以看出,东农42 GS1基因cDNA序列具有12个外显子,基因外显子及其侧翼序列的特征都符合GT、AG规则(图略)。以上分析结果充分证明了东农42 GS1基因具有12个外显子和11个内含子。
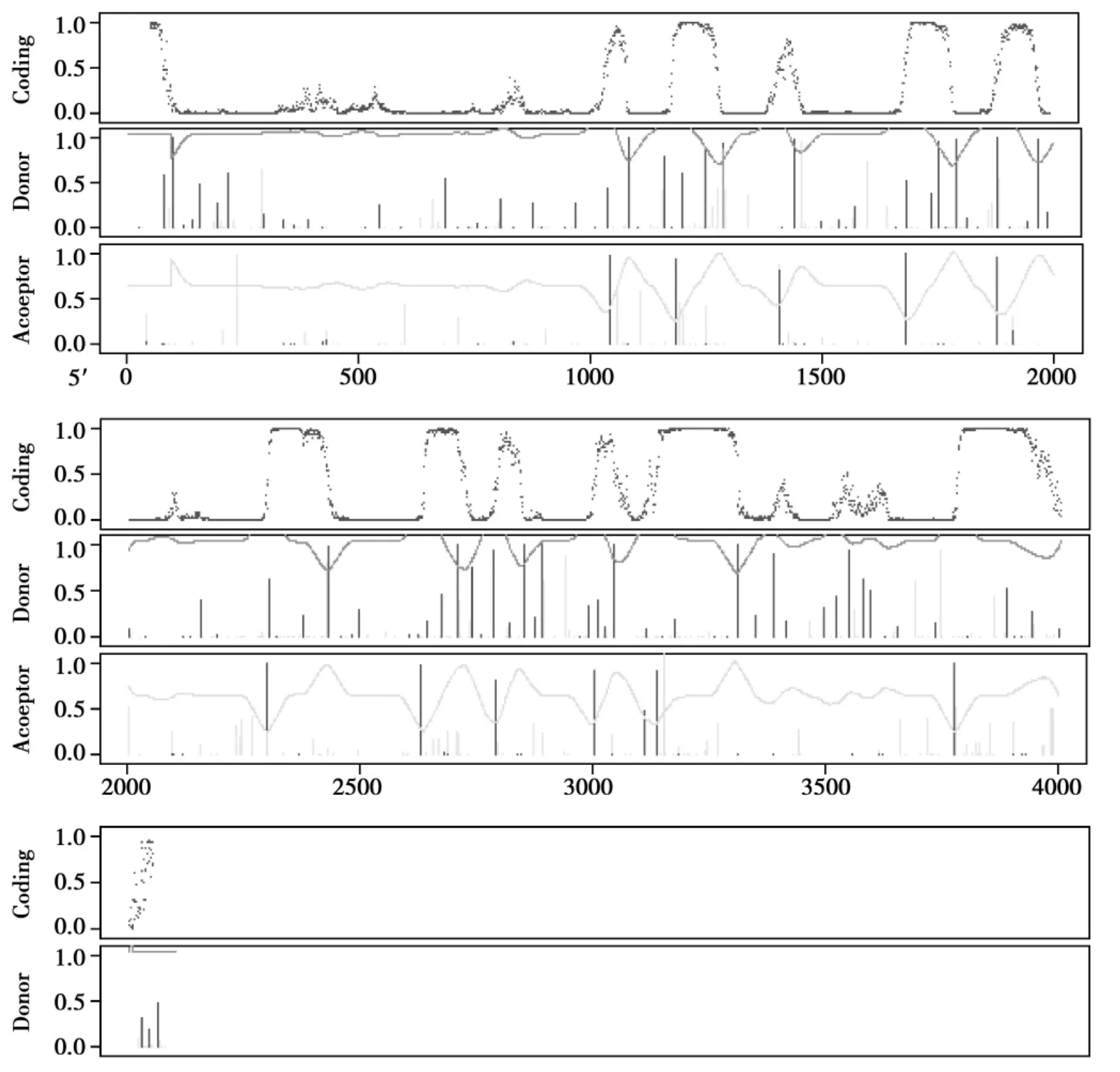
图7 NetGene2软件预测GS1基因外显子和内含子结果Fig.7 Graphics showing the prediction output of GS1 gene using NetGene2 software
3 讨论
基因序列的差异影响蛋白质序列,并最终导致酶分子结构和功能的差异,因此研究不同基因型大豆关键酶基因序列的差异是阐明大豆品质差异分子基础的关键之一。目前关于氮代谢关键酶基因的研究主要集中在GS,大麦、玉米、水稻、豌豆、大豆、苜蓿等许多植物的GS cDNA已被克隆,但做不同基因型间序列比较以及基因组序列克隆和分析的很少。
本研究克隆了不同基因型大豆GS1基因的cDNA,比较了基因序列及其编码的蛋白质序列的差异,为阐明不同基因型大豆氮代谢差异的分子机理奠定了基础。但是关于酶分子结构与功能之间的相关性还有待于深入研究。为阐明GS1基因的结构,本研究克隆了东农42 GS1基因的基因组片段。生物信息学分析表明该基因包括12个外显子和11个内含子,这与Cock等在苜蓿中的研究结果一致,反映了不同物种间同源基因结构的保守性[7]。
酶基因的表达调控是在不同水平上进行的,主要包括:①基因的结构;②mRNA的转录和加工;③翻译;④多肽的亚细胞定位、加工和修饰;⑤酶蛋白多聚体的组装;⑥酶活性的调控;⑦酶的降解。其中转录水平和酶活性水平的调控是关键环节[8]。由于不同基因型、不同生育期以及不同部位酶基因mRNA丰度的差异有限,因此对mRNA的精确定量是转录水平调控研究的关键。以前这方面的研究主要采用Northern杂交方法,不能实现精确定量。Real-time PCR技术的建立和发展使mRNA的精确定量成为可能。本研究比较了不同基因型大豆GS1基因的cDNA序列,发现了其共有序列,为设计通用Real-time PCR引物和探针,在转录水平研究不同基因型大豆GS1基因的表达规律奠定了基础。
4 结论
垦丰9、东农46、东农42、黑农35、秣食豆、野生大豆的GS1基因序列相似性较高,不同基因型大豆GS1基因序列间的差异主要集中在非编码区,东农42、半野生大豆GS1基因序列进化距离较近。不同基因型大豆GS1蛋白质序列具有很高的相似性,并且都具有两个保守的结构域,催化结构域(Gln-synt_N)和结合结构域(Gln-synt_C),东农46和东农42 GS1蛋白质序列进化距离较近,垦丰9和黑农35 GS1蛋白质序列进化距离较近。东农42 GS1基因具有12个外显子和11个内含子。
[1]Paula M M,Ligia M L,Isabel M S,et al.Expression of the plastidlocated glutamine synthetase of Medicago truncatula[J].Plant Physiology,2003,132(1):390-399.
[2]李常健,林清华,张楚富.高等植物谷氨酰胺合成酶研究进展[J].生物学杂志,2001,18(4):1-3.
[3]刘丽君,孙聪姝.氮肥对大豆结瘤及叶片氮素积累的影响[J].东北农业大学学报,2005,36(2):133-137.
[4]孙太靖,龚振平,马春梅.大豆植株氮素积累与转运动态的研究[J].东北农业大学学报,2004,35(5):517-521.
[5]代翠红,李杰,朱延明,等.不同提取方法对4种重要作物提取效率的比较[J].东北农业大学学报,2005,36(3):329-332.
[6]萨姆布鲁克.分子克隆实验指南[M].3版,北京:科学出版社,2002.
[7]Cock J M,Brock I W,Watson A T,et al.Regulation of glutamine synthetase genes in leaves of Phaseolus vulgaris[J].Plant Mol Biol,1991,17:761-771.
[8]Suzuki A,Knaff D B.Glutamate synthase:Structural,mechanistic and regulatory properties,and role in the amino acid metabolism[J].Photosynthesis Research,2005,83:191-217.
