亚胺培南耐药鲍曼不动杆菌的耐药性及耐药基因型分析
2011-02-09李小风苏小燕
熊 樱,李小风,苏小燕,夏 云
(重庆医科大学附属第一医院检验科,重庆 400016)
鲍曼不动杆菌是革兰阴性非发酵条件菌,是医院获得性感染的重要致病菌之一,可引起呼吸机相关性肺炎、菌血症等多种院内感染。该菌分布广泛、生存力强、定植率高、耐药率高、传播性强,极易造成院内感染的爆发流行。碳青霉烯类抗生素对其有较好的抗菌活性,通常作为治疗重症鲍曼不动杆菌感染的最后手段。随着临床碳青霉烯类抗生素的广泛应用,对其耐药的鲍曼不动杆菌的报道日益增多,给临床治疗带来了极大困难[1-2]。本研究旨在了解本院临床分离的亚胺培南耐药鲍曼不动杆菌耐药性特点、碳青霉烯酶和16S rRNA甲基化酶耐药基因型分析。
1 材料与方法
1.1 材料 菌株来源:收集本院2009年11月至2010年 1月临床分离的非重复的、对于亚胺培南耐药的鲍曼不动杆菌(imipenem resistant Acinetobacter baumannii,IRAB)54株。细菌来自不同科室患者的痰、咽拭子、血液、分泌物等标本,细菌分离培养按《全国临床检验操作规程进行》,所有菌株经Vitek-2 Compact(BioMerieux,France)全自动微生物分析仪鉴定确认。
1.2 主要试剂与仪器 Taq酶、基因扩增仪(PCR)相关试剂(大连 TaKaRa公司)、限制性内切酶 ApaI(美国 Promega公司)、药敏试条(法国 BioMerieux公司)、Vitek-2全自动微生物分析仪(法国 BioMerieux)、PCR扩增仪(美国 PE公司)、CHEF-M apper DRⅡ型脉冲场电泳仪(美国BioRad公司)。
1.3 方法
1.3.1 药敏和最小抑菌浓度(MIC)值 采用Vitek-2 Compact系统(BioMerieux,France)测定抗菌药物待测菌株的MIC值,按照美国临床实验室标准化协会(CLSI)标准配合AES高级专家系统进行结果判定和分析修正。
17种抗菌药物分别为:亚胺培南(IPM)、氨苄西林(AM)、氨苄西林/舒巴坦(AMS)、氨曲南(AZM)、头孢替坦(CTE)、头孢曲松(CTR)、头孢唑啉(CZ)、头孢吡肟等(FEP)、头孢他啶(TAZ)、呋喃妥因(FTN)、庆大霉素(GM)、妥布霉素(TOB)、丁胺卡那霉素(AN)、环丙沙星(CIP)、左旋氧氟沙星(LEV)、甲氧苄啶/磺胺甲恶唑(SXT)、哌拉西林/他唑巴坦(TZP)。质控菌株为大肠埃希菌ATCC25922,铜绿假单胞菌ATCC27853。
1.3.2 碳青霉烯酶基因及其插入序列的筛选 包括OXA-23组、OXA-24 组、OXA-51组、OXA-58组、IMP 型、VIM 型、ISAbal F/OXA-23R,采用煮沸法提取细菌DNA。引物合成、PCR反应体系及反应条件参照文献[3-6],引物由上海Invitrogen公司合成。PCR产物在含 0.5 μ g/mL溴化乙锭的 1%琼脂糖凝胶上电泳,在紫外光线下观察结果,在凝胶成像系统上成像。引物序列,见表1。
1.3.3 16S rRNA甲基化酶耐药基因筛选 PCR扩增4种主要的 16S rRNA甲基化酶耐药基因(armA、rmtA、rmtB、rmtC),由上海Invitrogen公司合成引物。引物合成、反应体系及反应条件参照文献[7-10]。引物序列,见表1。

表1 PCR引物序列
1.3.4 基因测序 PCR扩增阳性产物经纯化并送重庆医科大学感染病学重点实验室测序,结果在网上GenBank基因库中比对确定。
2 结 果
2.1 亚胺培南耐药鲍曼不动杆菌的耐药性 54株亚胺培南耐药菌株均表现为多重耐药,其中21株为全部耐药,其余菌株对青霉素类、广谱头孢菌素类、单酰胺类、碳青霉烯类、氨基糖苷类和喹诺酮类抗生素也都表现为高度耐药。亚胺培南、头孢菌素类、氨曲南等100%耐药,氨苄西林/舒巴坦、妥布霉素、丁胺卡那霉素等其它药物耐药率也几乎都在90%以上,见表2。
2.2 碳青霉烯酶编码基因PCR扩增结果 54株亚胺培南耐药的鲍曼不动杆菌中OXA-23引物扩增阳性53株,序列分析证实为OXA-23基因(GenBank登录号:CP001182),OXA-51引物扩增均阳性,序列分析证实为OX A-66基因(GenBank登录号:GQ914991),ISAbalF/OXA-23R基因(GenBank登录号:DQ923478)。PCR结果见图1。各株菌均未检测到OXA-24、OXA-58碳青霉烯酶基因,也没有检测到 IMP、VIM 型金属酶基因。
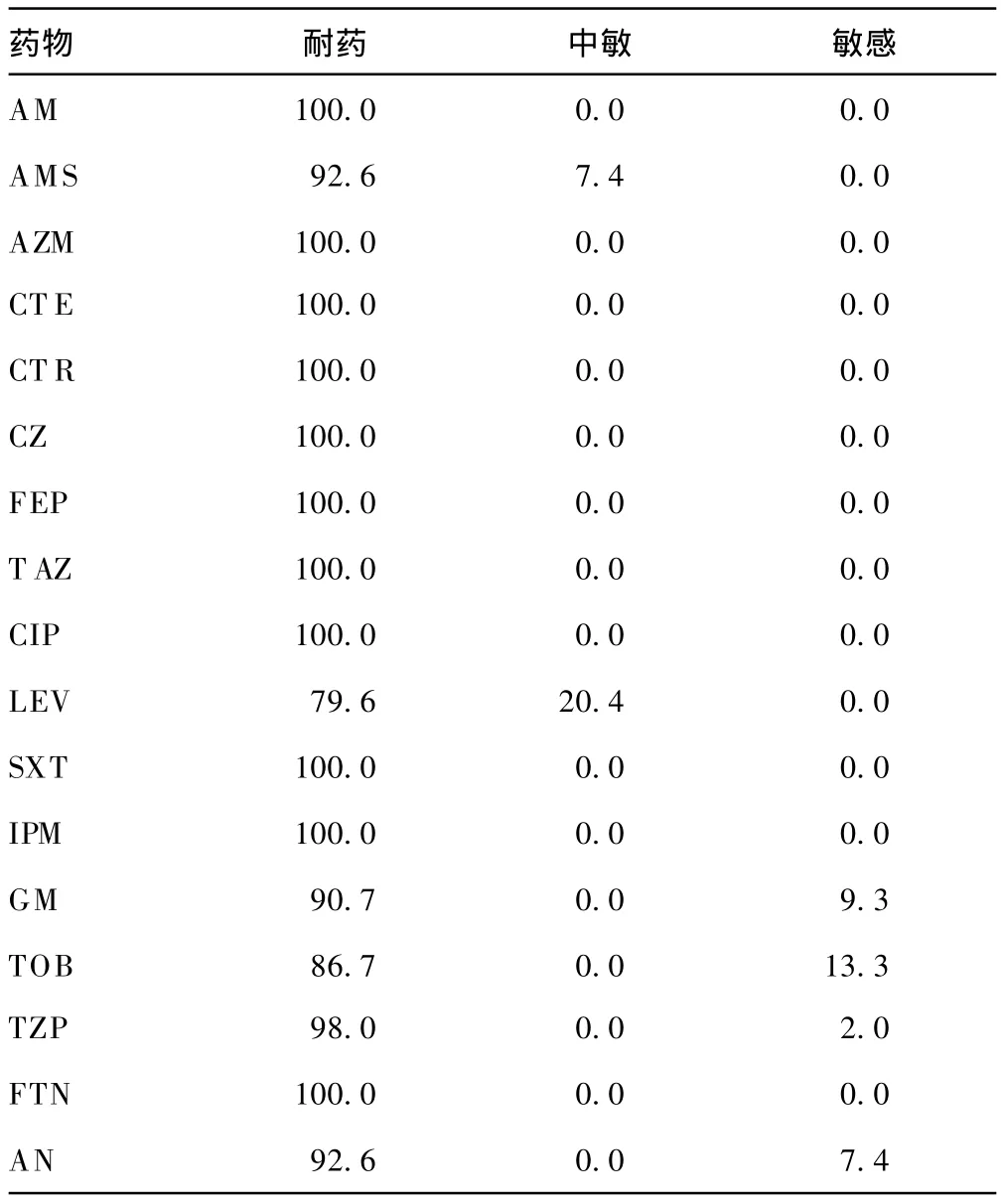
表2 亚胺培南耐药鲍曼不动杆菌对抗菌药物的耐药率(%)
2.3 16S rRNA甲基化酶基因PCR扩增结果 54株亚胺培南耐药的鲍曼不动杆菌中扩增到armA基因阳性的菌株有41株,皆对测定的5种氨基糖苷类抗生素耐药。PCR扩增产物测序与GenBank上armA的序列比对完全一样(GenBank登录号:GU437214)。其余3种16S rRNA甲基化酶基因rmtA、rmtB、rmtC引物扩增均为阴性。PCR结果见图1。
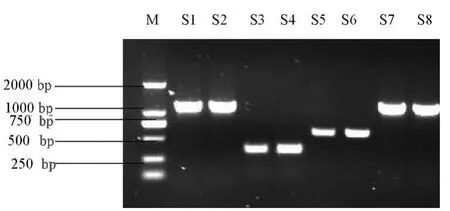
图1 PCR扩增结果
3 讨 论
碳青霉烯类抗生素有广谱的抗菌活性,尤其是能抵抗革兰阴性菌产生的超广谱β-内酰胺酶(ESBLS)和头孢菌素酶(AmpC)的水解,而被作为最后的治疗手段广泛应用于临床不动杆菌重症感染的治疗。但碳青霉烯类耐药鲍曼不动杆菌的出现,就意味着对现有的常用广谱抗菌药物均耐药,出现泛耐株(pan-resistance PDRA)[11]。PDRA引起的感染、爆发流行,常无药可用,病死率高。本院鲍曼不动杆菌对亚胺培南的耐药率由2005年的2.6%上升至2008年的25.5%,耐药率明显增加,而同期本院对碳青霉烯类抗生素人均使用量由2005年的0.074 g上升至2008年的0.19 g,使用量大量增加,表明耐药性增加与近年来临床上大量使用碳青霉烯类抗生素密切相关。应严格限制碳青霉烯类抗生素的使用以有效控制耐药性增长。本组细菌主要来自各科室的重症监护室,这可能与患者本身免疫力低,各种手术、介入治疗等侵入性操作有关。医护人员勤洗手、严格无菌操作、对感染或定植患者的及时发现和隔离等对控制流行十分重要。
大量研究显示,鲍曼不动杆菌对碳青霉烯类抗生素耐药的分子机制可能涉及外膜孔蛋白的缺失、外排泵的过度表达、青霉素结合蛋白的改变,而最重要的是碳青霉烯酶的产生。目前鲍曼不动杆菌中已发现的碳青霉烯酶主要有B类(VIM、IM P)金属酶和D类酶(即OXA酶),B类金属酶水解碳青霉烯的活力较强;D类酶只有微弱水解亚氨培南的能力,其耐药性的介导常合并外膜通透性的降低和/或外排泵的激活。
本研究PCR扩增、产物测序比对,表明本组细菌均携带blaOXA-66基因,53株菌产blaOXA-23型碳青霉烯酶,这与学者Wang[12]报道的blaOXA-23型碳青霉烯酶是介导我国鲍曼不动杆菌对亚氨培南耐药最主要的基因型基本一致。未检测到blaOXA-24/-58型碳青霉烯酶和IMP、VIM金属酶基因。blaOXA-23组基因阳性的菌株上游都携带插入序列ISAbal。ISAbal可能与OXA类碳青霉烯酶编码基因的同源重组以及表达调控有关。
16S rRNA甲基化酶最早是在产生氨基糖苷类抗生素的微生物体中发现,欧洲和日本学者先后在一系列肠杆菌中发现该种基因。它能使氨基糖苷类抗生素的作用靶位甲基化,药物无法结合到细菌16S rRNA,而导致高水平的氨基糖苷类耐药。研究表明16S rRNA甲基化酶大多位于可结合质粒以及整合子、转座子中,造成这些耐药基因在临床致病菌不同种属间传播,全球性的流行越来越引起关注[13]。armA、rmtA、rmtB、rmtC是目前发现的4种主要的16S rRNA甲基化酶基因[7-9]。本研究发现本组亚胺培南耐药的鲍曼不动杆菌都是多重耐药菌株,且对所有氨基糖苷类药物耐药率都在85%以上。54株亚胺培南耐药的鲍曼不动杆菌中筛选到41株菌携带armA基因,阳性率高达76%,远高于其他国家和地区。说明armA 16S rRNA甲基化酶基因在本院临床分离的亚胺培南耐药鲍曼不动杆菌中普遍存在,其介导的全部氨基糖苷类药物高水平耐药,使原本多重耐药问题变得更加严峻,甚至出现泛耐株,使临床治疗陷入困境。因此应合理使用抗生素,加强实验室对临床细菌耐药的监控,主张联合治疗[14],防止多重耐药菌株的播散,预防医院感染的爆发。
[1]Jain R,Danziger LH.Multidrug-resistant acinetobacter infections:an emerging challenge to clinicians[J].Ann Pharmacother,2004,38(9):1449-1459.
[2]何秀丽,夏云,张莉萍.2008年重庆医科大学附属第一医院临床病原菌分布和耐药性分析[J].重庆医学,2009,38(19):2428-2432.
[3]Jeon BC,Jeong SH,Bae IK,et al.Investigation of a nosocomial outbreak of imipenem-resistant Acinetobacter baumannii producing the OXA-23 beta-lactamase in Korea[J].Clin Microbiol,2005,43(5):2241-2245.
[4]Woodford N,Ellington MJ,Coelho JM,et al.Multiplex PCR for genes encoding prevalent OXA carbapenemases in Acinetobacter spp[J].Int J Antimicmb Agents,2006,27(4):351-353.
[5]Brown S,Young HK,Amyes SG.Characterisation of OXA-51,a novel class D carbapenemase found in genetically unrelated clinical strains of Acinetobacter baumannii from Argentina[J].Clin Microbiol Infect,2005,11(1):15-23.
[6]Jane FT,Elaina MW,Neil W,et al.The role of ISAbal in expression of OXA carbapenemase genes in Acinetobacter baumannii[J].FEMS Microbiol Lett,2006,258(1):72-77.
[7]Yokoyama K,Doi Y,Yamane K,et al.Acquisition of 16S rRNA methylase gene in Pseudomonas aeruginosa[J].Lancet,2003,362(9399):1888-1893.
[8]Doi Y,Yokoyama K,Yamane K,et al.P1asmid-mediated 16S rRNA methylase in Serratia marcescens conferring high-level resistance to aminoglycosides[J].Antimicmb Agents Chemother,2004,48(2):491-496.
[9]Galimand M,Courvalin P,Lambert T.Plasmid-mediated high-level resistance to aminoglycosides in Enterobacteriaceae due to 16S rRNA methylation[J].Antimicrob A-gents chemother,2003,47(8):2565-2571.
[10]Zhou H,Pi BR,Yang Q,et al.Dissemination of imipenemresistant Acinetobacter baumannii strains carrying the ISAba1 balOXA-23 genes in a chineses hospital[J].J M ed Microbiol,2007,56(8):1076-1080.
[11]王政,刘丁,陈萍.2008年重庆大坪医院细菌耐药性监测[J].重庆医学,2009,38(19):2400-2403.
[12]Wang H,Guo P,Sun H,et al.M olecular epidemiology of clinical isolates of carbapenem-resistant Acinetobacter spp.from Chinese hospitals[J].Antimicrob Agents Chemother,2007,51(11):4022-4028.
[13]Yamane K,Wachino J,Doi Y,et al.Global spread of multiple aminodlycoside resistance gene[J].Emer Infec Dis,2005,11(6):951-953.
[14]Levin AS,Levy CE,Manrique AE,et al.Severe nosocomial infections with imipenem-resistant Acinetobacter baumannii treated with Ampicillin/sulbactam[J].Int J Antimicrob Agents,2003,21(1):58-62.
