红茶菌中优势微生物的分离鉴定及系统发育分析
2011-01-11乔宏萍沙大年金泰廙杭晓敏
乔宏萍, 沙大年, 金泰廙, 杭晓敏
(1.上海交大昂立股份有限公司生物医药研究所,上海 200233;2.复旦大学公共卫生学院,上海,200032)
红茶菌中优势微生物的分离鉴定及系统发育分析
乔宏萍1,2, 沙大年*1, 金泰廙2, 杭晓敏1
(1.上海交大昂立股份有限公司生物医药研究所,上海 200233;2.复旦大学公共卫生学院,上海,200032)
采用不同的培养基对红茶菌优势微生物进行了分离,共得到酵母菌8.3×106个/mL,醋酸菌1.4×107个/m L,选取不同的菌落进行纯化后得到2株醋酸菌和4株酵母菌。经过分子生物学鉴定和系统发育树分析后,初步确定AC1为醋酸杆菌Acetobacter senegalensis,AC2为葡糖醋杆菌Gluconacetobacter saccharivorans;Y1为膜璞毕赤酵母Pichia membranifaciens,Y2为毕赤酵母Pichia galeiform is,Y3为异型德克酵母Dekkera anomala,Y4为拜耳接合酵母Zygosaccharom yces bailii。
红茶菌;醋酸菌;酵母菌;系统发育树
红茶菌是一种在国内外具有悠久历史的功能 性饮料,它是由醋酸菌、酵母菌等有益微生物共同自然发酵而成。其保健功能主要表现在清理肠胃,帮助消化,抑制有害菌,防治胃肠道疾病,预防结肠癌的发生,防治高血压和动脉硬化以及增强机体免疫力等。目前,对红茶菌的培养依然是以传统的家庭培养为主,实现工业化生产依然存在很多问题。首先,混合菌种的接种条件、接种量以及优势菌群等还没有系统全面的研究报道;其次,每一个菌种的代谢特征、培养条件和功能效果等方面还没有深入细致的研究;另外,抑菌和保健功能的作用机理还没有完全搞清楚[1-5]。因此,菌种的分离鉴定是解决所有问题的基础,作者从红茶菌培养液中分离了其优势菌群并用分子生物学方法对其进行鉴定,为其进一步研究发酵条件、代谢产物以及保健作用等奠定基础。
1 材料与方法
1.1 试验材料
红茶菌菌液:作者所在公司保存;醋酸菌分离培养基:葡萄糖 20 g、酵母膏10 g、碳酸钙20 g、琼脂20 g、p H 6.8水1 000 m L;酵母菌分离培养基:麦芽汁琼脂培养基;醋酸菌培养液:LB;酵母菌培养液:PB培养液。
1.2 试验方法
1.2.1 菌株的分离 平板稀释分离法。
1)分别取5 mL培养好的红茶菌菌液和一小块菌膜加入45 m L无菌水稀释,加入无菌玻璃珠,充分振荡,使溶液混匀,然后逐次稀释一直到10-3、10-4和 10-5。
2)分别取100μL稀释好的菌液,涂布在醋酸菌和酵母菌分离平板上,每个浓度重复3皿。
3)酵母菌平板放在28℃下恒温培养,醋酸菌在30℃下恒温培养,待醋酸菌分离平板上看到透明圈,然后纯化保存菌种,酵母菌分离平板上长出菌落后,挑取不一样的菌落进行纯化保存。
1.2.2 菌株的分子生物学鉴定
1)DNA的提取 分别挑取平板上生长旺盛的各个菌落接种在酵母菌或醋酸菌培养液中,待生长至对数生长期,离心取少量菌体,用 CTAB法[6]提取DNA,经琼脂糖凝胶电泳检测,纯化后-20℃冰箱保存待用。
2)PCR扩增和测序 酵母采用26srDNA近5端D1/D2区域通用引物[7,8]。PCR扩增体系(50 μL):10×PCR缓冲液5μL,M gCl2(25 mmol/L)3 μL;dN TP(10 mmol/L)1μL;Tag(2 U/μ L)1 μL;引物(10 pmol)1μL;DNA 1μL 加超纯水至50μL反应条件:95℃5 min,94℃1 min,52℃1 m in,72℃1 min 20 s 36个循环72℃8 min。
醋酸菌采用细菌16SrDNA通用引物[9,11],PCR反应体系(25μL):10×PCR缓冲液 2.5μL;M gCl2(25 mmol/L)2.0μL;dN TP(2.5 mmol/L)1.5μL;dN TP(2.5 mmol/L)1.5μL;TagDNA 聚合酶(2 U/μL)1μL引物(10μmol/L)各2μL超纯水14μL。PCR反应条件为:94℃5 min,94℃30 s,55℃1 min,72℃2 min,35个循环,最后72℃保温5 min。
所得PCR产物送上海鼎安生物科技有限公司纯化,测序。将所测序列登陆 GenBank数据库,进行BLAST比对,同时将所测序列通过Bankit提交GenBank,获取每个序列的登录号。
1.2.3 构建系统发育树 将所测序列在 GenBank核酸序列数据库中进行同源序列搜索,下载相似菌株序列,并与分离菌株的序列放在一起,用ClustalX软件进行多序列匹配排列,通过 Neighbo r-Joining分析方法用M ega4软件构建系统进化树。
2 结果与讨论
2.1 菌株的分离结果
采用两种不同的培养基分别分离醋酸菌和酵母菌,结果为,在醋酸菌分离平板上共分离到1.4×107个/m L菌株,挑取周围有透明圈并且菌落形态不一样的菌落进行分离纯化,共得到2株不同的菌株。在麦芽汁琼脂培养基上共分离到8.3×106个/m L菌株。挑取菌落形态不一样的菌株进行纯化保存,纯化得到4株不同的酵母菌。
2.2 菌株的分子生物学鉴定
将分离获得的2株醋酸菌进行DNA提取,PCR扩增均得到 16SrDNA序列,经比对后为A.senegalensis和G.saccharivorans。在麦芽汁琼脂平板上分离得到的酵母菌,进行DNA提取,PCR扩增均得到600 bp左右的一段序列。登陆 Genbank比对后,结果见表1。
2.3 系统发育分析
从红茶菌菌液中共鉴定分离到4株不同的酵母菌,2株不同的醋酸菌,通过Bankit提交 Genbank获取登录号(表1)。将4株酵母菌的26S rDNAD1/D2区域序列与 GeneBank中的序列进行比较,选取相近的典型菌株的26S rDNAD1/D2区域序列,用 ClustalX1.8与Mega4软件构建系统发育树见图1。同样的方法2株醋酸菌构建16SrDNA序列系统发育树见图2。
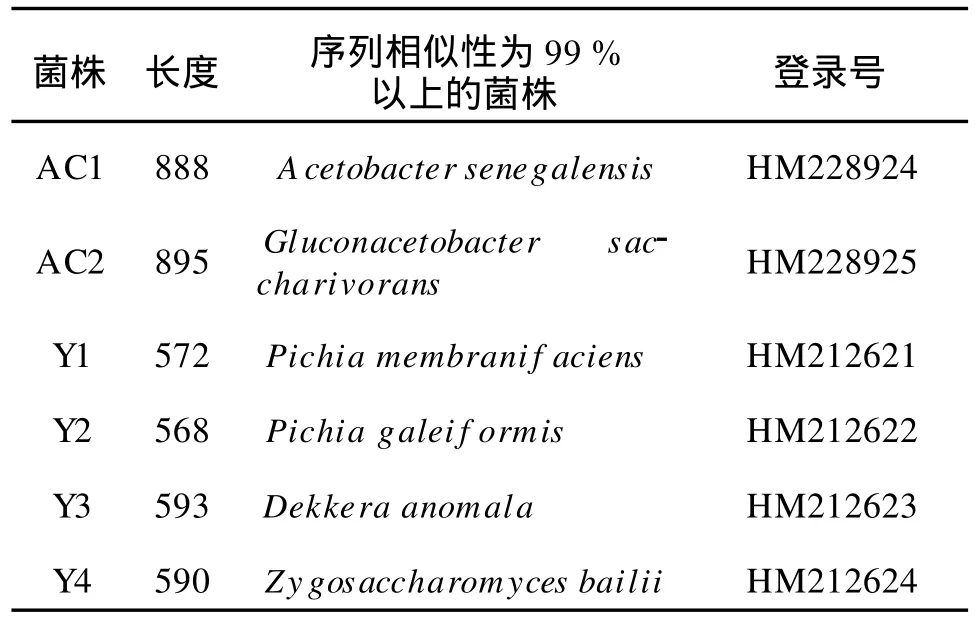
表1 红茶菌分离菌株的分子鉴定Tab.1 Molecular identification of strains isolated from Kombucha
从酵母菌26S rDNAD1/D2区域序列构建的系统发育树来看,Y1和 Y2亲缘关系较近,与P.membranifaciens和P.galeiform is聚在一起,在登 陆 Genbank比 对 后,Y1与P.membranifaciens相似性为 99%,同样 Y2与P.galeiform is相似性为99%,因此,初步将 Y1鉴定为P.membranifaciens,Y2鉴 定 为P.galeiform is。同样,Y3与D.anom ala在亲缘关系上最近,Y4与Z.bailii相似性最高,将二者在分类学地位上归于D.anom ala和Z.bailii。
从醋酸菌16SrDNA序列构建的系统发育树可以看出,AC2与G.saccharivorans聚在一起,亲缘关系较近,在分类上也将其归为G.saccharivorans。AC1单独形成一个分支,与A.senegalensis的遗传距离最近,在 Genebank中比对,与他的相似性也最高为99%,因此,也将其归为A.senegalensis。

图1 4株酵母菌的26S rDNA区域序列系统发育树Fig.1 Phylogenic tree of 4 strains yeasts based on 26S rDNA sequences

图2 2株醋酸菌的16S rDNA区域序列系统发育树Fig.2 Phylogenic tree of 2 strainsacetic bacteria based on16S rDNA
3 结 语
不同来源的红茶菌,其菌种存在差异,作者分离得到的醋酸菌和酵母菌在属间与报道的大体一致[3],种间略有不同。
用分子生物学方法鉴定微生物是一种快速、简单、有效地方法。在细菌的鉴定中,16SrDNA因其在漫长的进化中具有高度的保守性,因而近几年来被广泛的应用于细菌的分类学研究[9-12]。同样,酵母菌的26SrDNA中的D1/D2区域序列,可以很方便的把酵母菌区分开来。
(References):
[1]廖卢艳,蒋立文.红茶菌的研究进展[J].中国食品与营养,2006,9:22-23.
L IAO Lu-yan,JIANG Li-wen.The research development of Kombucha[J].Food and nutrition in China,2006,9:22-23(in Chinese)
[2]Dufresne,C and Farnworth,E.Tea,kombucha and health:A review[J].Food Research International,2000,33:409-421.
[3]吴薇,籍保平.红茶菌国内外研究应用概况[J].食品科技,2003,12:9-11.
WU Wei,JI Bao-ping.A survey of kombucha’s study and application in the world[J].Food Science and Technology,2003,12:9-11(in Chinese).
[4]吴薇,盖宝川.红茶菌混合菌种的分离与鉴定[J].食品科学,2004,25(4):55-58.
WU Wei,GAI Bao-Chuan.Study on the isolation and identification of microbes of Kombucha[J].Food Science,2004,25(4):55-58(in Chinese).
[5]Ai Leng Teoh,Gillian Heard,Julian Cox.Yeast ecology of kombucha fermentation[J].International Journal of Food Microbiology,2004,95:119-126.
[6]林加涵,魏文玲,彭宣宪.现代生物学实验[M].北京:高等教育出版社,2001.
[7]褚学英,惠丰立,李晓辉.大曲中主要酵母菌的分子鉴定[J].中国酿造,2008,5:27-29.
CHU Xue-ying,HU IFeng-li,L IXiao-hui.Molecular analysison diversity of culturable yeast isolated from Daqu[J].China brewing,2008,5:27-29(in Chinese).
[8]Kurtzman C P,Robnett C J.Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit(26S)ribosomal DNA partial sequences[J].Antonie van Leeuwenhoek,1998,73(4):331-371.
[9]汪川,张朝武,裴晓方,等.一株从酸奶中分离的德氏乳杆菌保加利亚亚种的鉴定和系统发育分析[J].卫生研究,2007,36(6):713-718.
WANG Chuan,ZHANG Chao-wu,PEI Xiao-fang,et al.Identification and phylogeneti c analysis of one strain of Lactobacillus delbrueckii subsp.Bulgaricus separated from yoghourt[J].Journal of hygiene Research,2007,36(6):713-718(in Chinese).
[10]姜海荣,倪永清,宋丽军,等.新鲜哈密瓜汁中可培养细菌的分离鉴定及系统发育分析[J].食品与生物技术学报,2010,29(3):426-431.
JIANG Hai-rong,N I Yong-qing,SONG Li-jun,et al.Identification and phylogenesis analysis of cultivable microo rganisim isolated from fresh hami-melon juice[J].Journal of food science and biotechnology,2010,29(3):426-431(in Chinese).
[11]张会敏,冯友军.一株野生细菌的16SrDNA序列分析与系统发育树的构建[J].生物信息学,2005,3(1):1-4.
ZHANG Hui-min,FENG You-jun.16SrDNA sequence analysis and phylogenic tree construction of a strain of field bacterium[J].China Journal of Bioinformatics,2005,3(1):1-4(in Chinese).
[12]龙雯,陈存社.16SrRNA测序在细菌鉴定中的应用[J].北京工商大学学报:自然科学版,2006,24(5):10-12.
LONG Wen,CHEN Cun-she.Application of 16SrRNA sequence in bacteria indication[J].Journal of Beijing technology and business university:Natural Science Edition,2006,24(5):10-12(in Chinese).
Isolation and Identification of Predominant Microbes from Kombucha and Phylogenic Analysis
QIAO Hong-ping1,2, SHA Da-nian*1,JIN Tai-yi2, HANG Xiao-min1
(1.Bio-Pharmaceutical Research Institution,Shanghai JIAO DA ONLL Y CO.,L td.Shanghai 200233,China;2.School of Public Health,Fudan University,Shanghai200032,China)
In this manuscript,,the predominant microbes were isolated from Kombucha by different medium,8.3 106cfu/m L of yeasts and 1.4 107cfu/m L of acetic bacteria were obtained.There were 4 strains of yeasts and 2 strains of acetic bacteria through purified from different colonies.By molecular identified and phylogenic analyzed,the isolate AC1 was identified asAcetobactersenegalensis,AC2 asGluconacetobactersaccharivoran, Y1 asPichia membranifaciens, Y2 asPichiagaleiform is, Y3 asDekkeraanom ala, Y4 asZygosaccharom yces bailii.
Komucha,acetic bacteria,yeast,phylogenic analysis
Q 93.331
A
1673-1689(2011)04-0609-04
2010-08-17
国家863计划项目(2007AA 10Z355)。
乔宏萍(1978-),女,山西孝义人,农学博士,主要从事应用微生物研究。Email:xiaoqiao2001@hotmail.com
*通信作者:沙大年(1961-),男,上海人,高级工程师,主要从事保健品的开发与应用研究。Email:xxsha166@hotmail.com
book=612,ebook=262
