广西茅尾海常见牡蛎的分子鉴定
2010-10-23宋忠魁蔡小辉杨家林
宋忠魁, 蔡小辉, 童 潼, 杨家林
(广西海洋研究所 广西海洋生物技术重点实验室, 广西 北海 536000)
广西茅尾海常见牡蛎的分子鉴定
宋忠魁, 蔡小辉, 童 潼, 杨家林
(广西海洋研究所 广西海洋生物技术重点实验室, 广西 北海 536000)
应用COⅠ条形码技术鉴定广西茅尾海的3种常见牡蛎, 其俗名分别是“白眼蚝”、“红眼蚝”和“蚝砺”。研究结果表明, 茅尾海3种牡蛎应归属牡蛎科(Ostreidae)、巨蛎属(Crassostrea), 其中“白眼蚝”是香港巨牡蛎(C.hongkongensis)、“红眼蚝”是有明巨牡蛎(C. ariakensis)、“蚝砺”是 Kumamoto牡蛎(C.sikamea)。研究验证了已开发的多重PCR技术能对3种牡蛎进行快速鉴定, 并揭示香港巨牡蛎是茅尾海优势种, 基本上澄清了与“近江牡蛎”相关的俗名问题。
茅尾海; 牡蛎; COⅠ条形码; 分子鉴定
牡蛎(Ostreidae)属软体动物门Mollusca、双壳纲Lamellibranchiata、珍珠贝目 Pterioida, 为世界性广布种。牡蛎广栖性, 贝壳可塑性强, 因此牡蛎的多数种类单纯依靠贝壳的外部形态很难精确区分。牡蛎异地引种频繁, 不仅打破了种间地理隔离, 也人为地扩大了种的生存空间范围, 进一步加剧牡蛎基于形态特征鉴定的难度。为之, 牡蛎分类学家倡导牡蛎物种的准确鉴定应结合传统的形态分类学方法与分子标记技术[1,2]。
应用分子标记识别牡蛎物种身份是当前牡蛎分类学研究热点之一。测序目的基因并结合系统发育分析能有效地鉴定牡蛎物种。例如, 王海艳[3]应用线粒体基因(16S和COⅠ)和核基因(28S)并结合形态学特征对中国近海常见牡蛎进行的分类研究。Reece等[4]认为分子系统发育分析有助于澄清牡蛎分类的争议问题, 利于异地引种。某些目的基因譬如ITS区在一些牡蛎种间的长度本身差异显著, 所以其本身的长度多态性可以应用于牡蛎种的鉴定。Wang和Guo[5]认为ITS1和ITS2的长度多态性能有效地鉴定4种牡蛎和 4组牡蛎种对。基于序列分析发展的 PCRRFLP和多重 PCR技术能对一些牡蛎种类进行快速鉴定。ITS1-RFLP能有效地区分巨蛎属的7个物种,COⅠ-RFLP能有效地区分巨蛎属的 9个物种[6]。Wang和Guo[7]开发的多重PCR技术能有效区分巨蛎属的5个物种, 无须酶切鉴定。
广西钦州茅尾海是一个口袋形状的内海, 位于钦州湾的顶端, 地处东经 108°28′33″~108°55′33″, 北纬 21°33′15″~21°54′40″, 面积 135 km2, 是我国最大的“近江牡蛎”(Ostrea rivularis)天然采苗基地。茅尾海常见牡蛎主要有3种, 俗称“白眼蚝”、“红眼蚝”和“蚝砺”(亦称蚝蜊、黄蚝), 其中“白眼蚝”和“红眼蚝”正是茅尾海蚝民眼中的“近江牡蛎”。
“近江牡蛎”的分类问题是牡蛎物种分类学争议的焦点之一。多数牡蛎分类学家认为“近江牡蛎”这一学名不应该存在, 先前所称的“近江牡蛎”可区分为两个物种, 即香港巨牡蛎(Crassostrea hongkongensis)和有明巨牡蛎(C. ariakensis)[8]。再者, “近江牡蛎”与多组俗名发生联系[3,8,9], 如“白肉”和“红肉”, “白蚝”和“赤蚝”, “白眼蚝”和“红眼蚝”等, 进一步升级了“近江牡蛎”的分类问题。那么, 这些俗名到底是怎样一种等同关系, 所有俗名的正式学名又是什么, 都有待于给出明确的答案。
鉴于牡蛎COⅠ的数据不断累积, 因此应用COⅠ条形码鉴定中国近海常见牡蛎已成为可能。利用COⅠ条形码技术鉴定茅尾海 3种牡蛎, 探讨“近江牡蛎”相关俗名之间的关系, 为“近江牡蛎”的分类问题提供佐证。更进一步验证多重PCR技术的快速鉴定作用, 分析茅尾海的牡蛎优势种, 为茅尾海牡蛎资源的保护和合理开发利用提供依据。
1 材料与方法
1.1 实验材料
实验用牡蛎于2008年12月20日采自茅尾海增养殖区, 随机采样, 收集60个个体, 来自吊养、立养两种养殖方式。牡蛎物种身份经蚝民初步判别后, 取闭壳肌, 95%乙醇固定3次, -20℃储存备用。
1.2 方法
1.2.1 DNA 提取
取乙醇固定后的少量闭壳肌肌肉组织(约50 mg)于1.5 mL EP管, 其余肌肉组织留作凭证, 加600 µL STE缓冲液, 使用眼科剪剪碎肌肉组织, 再加入 60µL 10%SDS和10 µL 20%蛋白酶K, 55 ℃水浴消化完全(约 2~3 h), 然后采用常规的酚-氯仿-异戊醇方法依次抽提, 使用异丙醇于-20℃沉淀DNA过夜, 70%乙醇洗涤, 空气晾干, 50~100 µL灭菌双蒸蒸馏水溶解, -20℃保存备用。
1.2.2 PCR扩增与测序
合成 COⅠ条形码的通用引物 LCO-1490(5'-GGTCAACAAATCATAAAGATATTGG-3')和HCO-2198(5'-TAAACTTCAGGGTGACCAAAAAATCA-3')进行 PCR扩增。在 AG 22331型 PCR 扩增仪(Eppendorf, Germeny) 上进行, 反应总体积 25 µL,其中含10 ×缓冲液2.5 µL, 2.5 mmol/LdNTP 混合液1.0 µL, 2.5 mmol/L MgCl21.5 µL, DNA 模板 1 µL(< 50 ng), 5U/µL Taq 酶 0. 5 µL(以上试剂均为大连宝生物工程有限公司产品), 引物终浓度为0.4 µmol/L,补足灭菌双蒸蒸馏水至25 µL。优化后PCR 扩增条件为: 94℃预变性5 min; 94℃变性60 s, 52℃退火60 s, 72℃延伸 60 s, 30个循环; 最后在 72℃延伸 10 min。PCR 产物经 1.0%琼脂糖凝胶电泳, 溴化乙锭(EB)染色, 用凝胶图像及分析系统(Alpha innotech,USA)观察、照相。阴性对照组用灭菌双蒸蒸馏水代替模板 DNA。所有 PCR反应均获得单一扩增条带,且大小与预期一致(600~700 bp之间)。PCR扩增产物直接送至上海生物工程有限公司测序, 扩增引物用作测序引物。
1.2.3 序列整理与邻接(neighbour-joining, NJ)分析
使用DNAStar软件包中的SeqMan程序拼接序列并结合 Chromas程序判别可疑位点, 执行软件包中的EditSeq程序识别引物位置并剪切引物序列。使用 Blastn搜索同源序列并确定下载序列的登陆号。拟下载的同源序列须满足的首要条件是: 同源序列的长度至少不低于目的片段的长度。使用 Blastx确定编码位点的位置。利用软件ClustalX1.81进行序列比对, 使用软件MEGA3.1中的邻接法进行聚类分析,系统发育树分支置信度使用自引导检验(Bootstrap test)估计, 1 000次重复。
1.2.4 多重PCR基因分型
基于序列信息可以推定物种身份, 但为了再次印证物种身份, 主要为了验证多重 PCR是否能快速鉴定物种身份, 参考文献[4], 合成5条引物, 建立多重 PCR的反应体系和反应条件。引物信息如下:COCar183r(5'-AAAAAAGATTATAACTAATGCATGTCGG-3'), COChk 387r(5'-GGAGTAAGTGGATAAGGGTGGATAG-3'), COCsi546r(5'-AAGTAACCTTAATAGATCAGGGAACC-3'),其中 LCO-1490 和 HCO-2198 同上(示 1.2.2); 反应体系是: 总体积 25 µL, 其中含 10 ×缓冲液 2.5 µL, 2.5 mmol/LdNTP 混合液 1.0µL, 2.5 mmol/L MgCl21.5 µL, DNA 模板 1 µL (< 50 ng), 5U/µL Taq 酶 0. 2 µL(以上试剂均为大连宝生物工程有限公司产品), 前 3条引物终浓度为 0. 24µmol/L , 后两条引物终浓度为0. 36 µmol/L , 补足灭菌双蒸蒸馏水至25 µL; 反应条件是: 94 ℃预变性5 min; 94 ℃变性60 s, 52℃退火60 s, 72 ℃延伸60 s,30个循环; 最后在72 ℃延伸10 min。PCR 产物经1.5 %琼脂糖凝胶电泳, 溴化乙锭(EB)染色, 用凝胶图像及分析系统(Alpha innotech,USA)观察、照相。
2 结果与讨论
2.1 分子鉴定
2.1.1 COⅠ条形码
COⅠ条形码技术的出现和发展应归功于Folmer等[10]开发的一对引物LCO-1490和HCO-2198, 该引物对的通用性极强, 已在诸多物种类群如鳞翅目、鸟类、双翅目等得到成功扩增[11~13], 尽管扩增的目的片段大小在不同物种类群间有少许差异, 譬如, 昆虫为658 bp, 鸟类是648 bp。
已有的研究表明, COⅠ条形码技术可用于物种鉴定和新种的发现[14]。本研究利用引物对LCO-1490和HCO-2198在3种牡蛎8个个体中成功扩增出单一的COⅠ目的条带。经测序、序列拼接、引物剪切后, 得到的目的片段大小均为 649bp, “白眼蚝”和“红眼蚝”个体间无序列差异, “蚝砺”两个个体之间在序列位置 178(T→C)、343(C→A)和 373(T→C)发生碱基替换。Genbank 登录号是 GQ214236,GQ214237, GQ214238, GQ214239, GQ214240,GQ214241, GQ214242。
利用 Blastn进行同源搜索, 基于序列相似性(Identities)可以初步推定, “白眼蚝”是香港巨牡蛎(Crassostrea hongkongensis), “红眼蚝”是有明巨牡蛎(C. ariakensis), “蚝砺”是Crassostrea sikamea(即Kumamoto 牡蛎)。
下载在线序列46条, 其中6条来自完整线粒体基因组, 它们的 Genbank 登录号及其在线粒体基因组中的对应位置分别是 EU672834(126~774)、FJ593172(126~774)、FJ593173(126~774)、EU266073(129~777)、EU672831(126~774)、AF177226(15624~16272)。超过649 bp的序列有2条, Genbank 登录号是AF280608和AF300616。除此以外的其余序列的长度均为649 bp。序列AF300616长度为663 bp, 但其同源序列的长度只有645 bp, 鉴于AF300616与香港巨牡蛎有很近的亲缘关系, 因此也纳入邻接分析。
应用软件MEGA3.1对53条序列执行邻接分析,结果示图 1a。 图 1a是一棵无根树, 反应的种间关系是 ((C.ariakensis, C.hongkongensis, C.nippona),(C.gigas, C.sikamea))与Reece等[4]的研究结果高度吻合。Reece等基于COⅠ片段重建的巨蛎属种间关系覆盖了现有巨蛎属已知种类, 为巨蛎属种身份的判定提供了重要参考, 即存在一个或多个未知种与已知种有更近的亲缘关系。由图 1a可以看出, “白眼蚝 ”序 列 (GQ214238, GQ214239, GQ214240)与C.hongkongensis的相关序列聚成一支, Bootstrap值高达100%, 且与参考序列中的大部分(60%, 9/15)无序列差异。结合已知的巨蛎属种间关系可以推定,“白眼蚝”与 C.hongkongensis是同一物种。“红眼蚝”序列(GQ214236, GQ214237)与在线序列EU007493、EU007494比较, 存在 1个碱基差异, 即在同源序列的位置46发生了T→C的转换。从树的拓扑结构来看, “红眼蚝”与C. ariakensis是同一物种。“蚝砺”个体之间存在的序列差异并没有让它们跨越种的界限。首先, 两个个体的序列聚在一个进化支, 并没有分布在两个进化支; 其次, 两个个体的序列只与C.sikamea的相应序列聚成一支, Bootstrap值高达100%。凭此两点以及已知的巨蛎属种间关系可以推断, “蚝砺”是 C.sikamea。不过, 同域分布的“蚝砺”个体间存在较大的序列差异值得进一步研究。
2.1.2 多重PCR
简单改动Wang和Guo[7]开发的多重PCR技术,用于鉴定上述3个物种, 结果示图1b。由图1b可见,3个物种产生了不同的基因分型, 其中外引物(outer primers, LCO-1490和HCO-2198)在3个物种产生的内参照带约700 bp清晰、可辨, “红眼蚝”的特征带位于100~250 bp之间, “白眼蚝”的特征带位于250~500 bp之间, “蚝砺”的特征带位于500~700 bp之间。依据Wang和Guo的研究结果, 可以推定“红眼蚝”是有明巨牡蛎, “白眼蚝”是香港巨牡蛎, “蚝砺”是Kumamoto牡蛎。不可否认, 当前扩增体系的确产生了非特异扩增, 非特异扩增带主要出现在1 000~2 000 bp之间, 但并不影响本研究中3个物种的鉴定。随机取样采苗区3龄蚝60个个体, 随机取样的方式就是随机选择吊养绳和蚝柱, 应用目前扩增体系进行鉴定。鉴定结果表明: (1)60个个体中,“红眼蚝”约占 5%, “白眼蚝”占 90%左右, “蚝砺”约占5%; (2)蚝民对种的判断与当前分子鉴定结果 100%吻合; (3)红眼蚝主要出现于吊养区(即盐度较高海域), 蚝砺主要出现于立养区(即盐度较低海域)。研究结果揭示, “白眼蚝”(香港巨牡蛎)是茅尾海采苗区优势种, 为茅尾海“近江牡蛎”原种场建设提供了科学依据。
2.2 “近江牡蛎”的俗名问题
王海艳等[3,8]在研究“近江牡蛎”的分类问题时,曾使用过两套俗名“白肉和红肉”以及“白蚝和赤蚝”。从其研究结果来看, “白肉”等同于“白蚝”, 学名是香港巨牡蛎, “红肉”等同于“赤蚝”, 学名是有明巨牡蛎(又订名为近江牡蛎)。结合本研究可以得出如下结果:俗名“白眼蚝”、“白肉”、“白蚝”是同一物种, 即香港巨牡蛎; 俗名“红眼蚝”、“红肉”、“赤蚝”是同一物种,即有明巨牡蛎。但问题远不止于此, 苏天凤[9]在研究华南沿海增养殖区“近江牡蛎”时, 也有“白蚝和赤蚝”的提法, 认为“白蚝”是香港巨牡蛎和有明巨牡蛎两个种的混合体, “赤蚝”可能是一个新种。由此看来,苏天凤认为“近江牡蛎”应该包含3个种。显然, 苏天凤提及的“白蚝和赤蚝”与王海艳等谈及的“白蚝和赤蚝”不能等同, 进一步言, 也不能等同于“白肉”和“红肉”, 也不能等同于“白眼蚝”和“红眼蚝”。
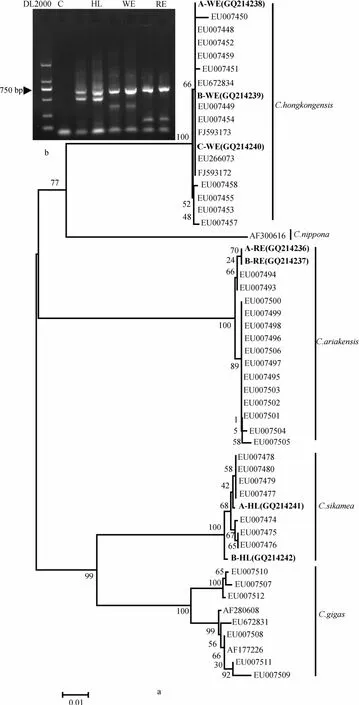
图1 邻接分析(a)和多重PCR(b)结果Fig. 1 Results of neibour-joining analysis and multi-plex PCR图中WE, RE, HL指的是白眼蚝, 红眼蚝和蚝砺。图1a中枝上数值指的是自展值, 鉴定样本的标本号和Genbank登陆号使用加粗字体显示。 图1b中的“C”暗示阴性对照WE, RE, and HL represent white-eye oyster, red-eye oyster and Haoli, respectively. The numerical values on the branches denote the bootstrap values in the Fig.1a. The bold fonts are used to mark the specimen numbers and Genbank accession numbers of the detected samples. The letter“C” in the Fig.1b indicates the negative control
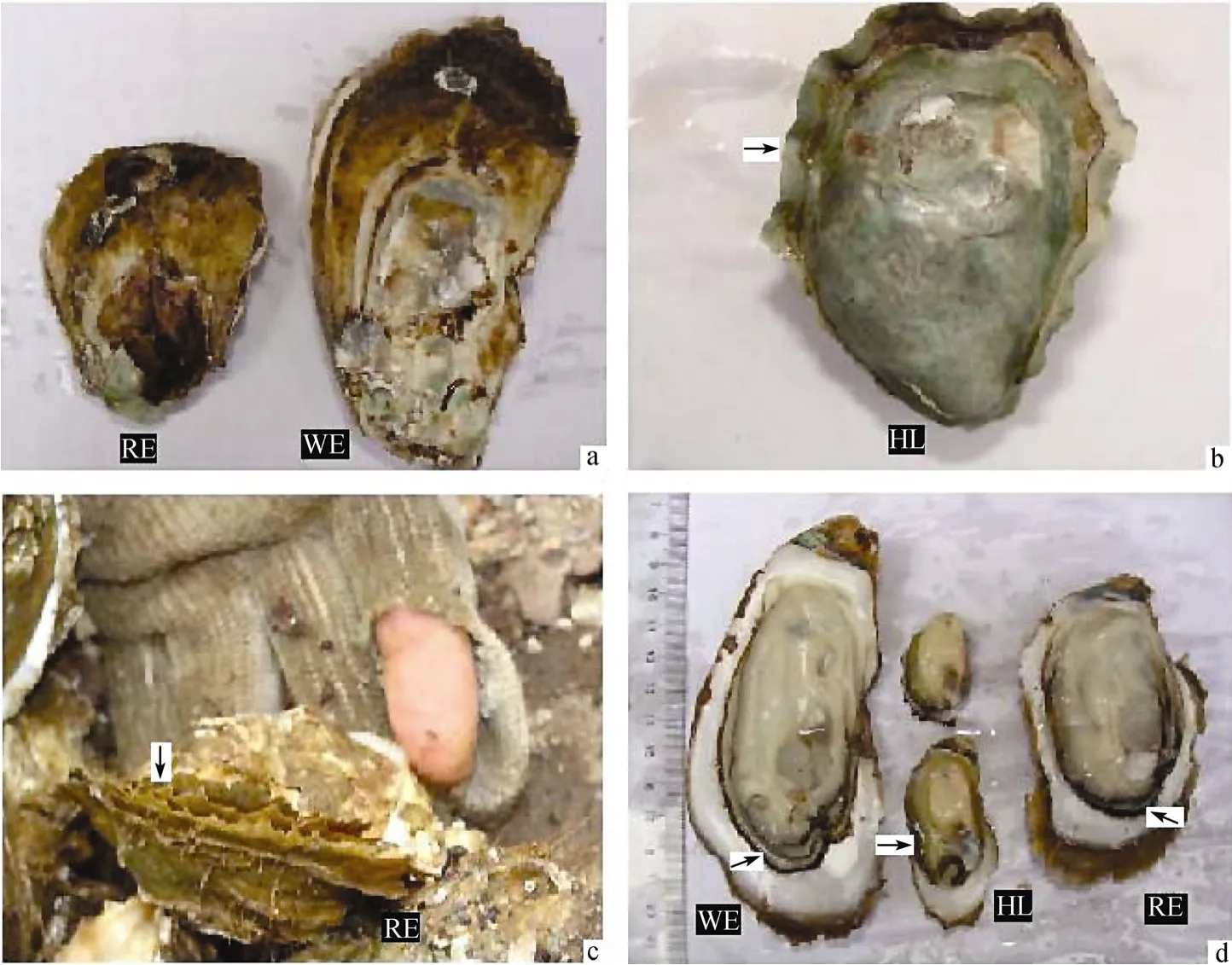
图2 白眼蚝(WE), 红眼蚝(RE)和蚝砺(HL)的形态学特征Fig. 2 Morphological characteristics of white-eye oyster, red-eye oyster and “Haoli”箭头指出种识别的形态特征和可比较的外套膜特征The positions pointed by the arrow heads can be used for the recognition of the species identities.
蚝民一般依据如下特征辨别“白眼蚝”、“红眼蚝”和“蚝砺”: (1)壳的形态。“白眼蚝”长形或长圆形, 右壳鳞片少; “红眼蚝”偏于圆形或卵圆形, 右壳鳞片多,典型的右壳鳞片呈“菊花”状(示图2a和图2c); “蚝砺”左壳边缘起波浪形(图2b)。(2)软体部和外套膜颜色。“蚝砺”软体部的颜色呈黄色或黄褐色, 且外套膜的颜色亦呈黄色, 所以“蚝砺”又有“黄蚝”之称。“白眼蚝”和“红眼蚝”的软体部分呈白色或灰白色, 前者外套膜颜色偏白, 后者外套膜颜色偏黑或灰色(图2d)。苏天凤描述“白蚝”和“赤蚝”软体部颜色如下: 前者呈乳白色或灰白色, 后者呈黄褐色, 广东阳江一带蚝民也称“赤蚝”为“黄蚝”。王海艳等谈及的“白蚝和赤蚝”软体部颜色如下: “白蚝”的软体部颜色为雪白色, “赤蚝”的颜色接近褐色。单凭肉质颜色可以推定: 苏天凤提及的“白蚝”对应的可能是“白眼蚝”和“红眼蚝”, 提及的“赤蚝”对应的可能是“蚝砺”。但据此作出推断毕竟偏颇, 仍需采集标本, 重新审视形态学特征并结合分子鉴定以进一步证实。
致谢: 感谢海南大学王爱民教授和广西红树林研究中心阎冰研究员在技术和理论上给予的建议。感谢钦州市钦南区沙井养蚝专业合作社钟应桥先生在标本采集方面给予的大力支持。
3 结论
本研究应用COⅠ条形码技术对茅尾海3种牡蛎进行了有效鉴定, 印证了 Wang和 Guo开发的多重PCR技术能用于茅尾海 3种牡蛎的快速鉴定, 且推定茅尾海采苗区优势种是香港巨牡蛎, 基本上澄清了“近江牡蛎”的俗名问题。
[1] 阙华勇, 刘晓, 王海艳, 等. 中国近海牡蛎系统分类研究的现状和对策[J]. 动物学杂志, 2003, 38(4):110-113.
[2] 王海艳, 郭希明, 刘晓, 等. 中国北方沿海“褶牡蛎”的分类和订名[J]. 海洋科学, 2009, 33(10): 104-106.
[3] 王海艳. 中国近海常见牡蛎分子系统演化和分类的研究[D]. 青岛: 中国科学院海洋研究所, 2004.
[4] Reece K S, Cordes J F, Stubbs J B,et al.Molecular phylogenies help resolve taxonomic confusion with AsianCrassostreaoyster species[J]. Mar Biol, 2008,153: 709-721.
[5] Wang Y P, Guo X M. ITS length Polymorphism in oysters and its use in species identification [J]. J. Shellfish Res, 2008, 27(3): 489-493.
[6] Cordes J F, Jie X, Reece K S. Discrimination of nineCrassostreaoyster species based upon restriction fragment-length polymorphism analysis of nuclear and mitochondrial DNA markers [J]. J Shellfish Res, 2008,27(5): 1155-1162.
[7] Wang H Y, Guo X M. Identification of Crassostrea ariakensis and related oysters by multiplex species-specific PCR[J]. J Shellfish Res, 2008, 27(3):481-487.
[8] 王海艳, 郭希明, 刘晓, 等. 中国近海“近江牡蛎”的分类和订名[J]. 海洋科学, 2007, 31(9): 85-86.
[9] 苏天凤. 华南沿海养殖近江牡蝠的分类研究[J]. 南方水产, 2006, 2(6): 72-75.
[10] Folmer O, Black M, Hoeh W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Mol Mar Biol Biotechnol, 1994, 3: 294-299.
[11] Hebert P D N, Stoeckle M Y, Zemlak T S, et al. Identification of Birds through DNA Barcodes[J]. PLoS Biology, 2004, 2(10): 1-7.
[12] Smith M A, Woodley N E, Janzen D H, et al. DNA barcodes reveal cryptic host-specificity within the presumed polyphagous members of a genus of parasitoid flies (Diptera: Tachinidae) [J]. PNAS, 2006, 103(10):3657-3662.
[13] Hajibabaei M, Janzen D H, Burns J M, et al. DNA barcodes distinguish species of tropical Lepidoptera[J].PNAS, 2006, 103(4): 968-971.
[14] Savolainen V, Cowan R S, Vogler A P, et al. Towards writing the encyclopaedia of life: an introduction to DNA barcoding[J]. Phil Trans RSoc B, 2005, 360:1805-1811.
Molecular identification of the common oysters from the Maowei sea in Guangxi, China
SONG Zhong-kui, CAI Xiao-hui, TONG Tong, YANG Jia-lin
(Guangxi Key Laboratory for Marine Biotechnology, Guangxi Institute of Oceanology, Beihai 536000, China)
Sep., 30, 2009
Maowei sea; oyster; COⅠbarcodes; molecular identification
The COⅠbarcodes were used to identify three common oysters from the Maowei sea in Guangxi. Their ethnic names are white-eye oyster, red-eye oyster, and “Haoli”. Our results suggested that the three oysters should be classified as genus Crassostrea within the family Ostreidae, with the scientific names being C. hongkongensis, C.ariakensis, and C. sikamea, respectively. The exploited multiplex PCR assays were able to identify the three oysters fast and reliably, and proved to us that C. hongkongensis was the predominant species of the Maowei sea. Moreover,the combination of the molecular and morphological evidences clarified the confusion of the ethinic names concerned with Ostrea rivularis.
Q179
A
1000-3096(2010)08-0011-06
2009-09-30;
2009-11-02
广西海洋生物技术重点实验室主任基金(GKLMBTD0801)
宋忠魁(1973-), 男, 副教授, 主要从事海洋经济动物分子标 记 辅 助 选 育 研 究 , 电 话 : 0779-2057372, E-mail: songsir2003@yahoo.com.cn
(本文编辑: 张培新)
