靶向HIV-1 pol的高效人工miRNA的构建与体外抗病毒能力评价
2010-10-16程通张涛张雅丽魏丽华夏德镇王颖彬张军夏宁邵
程通,张涛,张雅丽,魏丽华,夏德镇,王颖彬,张军,夏宁邵
厦门大学生命科学学院 国家传染病诊断试剂与疫苗工程技术研究中心,厦门 361005
靶向HIV-1 pol的高效人工miRNA的构建与体外抗病毒能力评价
程通,张涛,张雅丽,魏丽华,夏德镇,王颖彬,张军,夏宁邵
厦门大学生命科学学院 国家传染病诊断试剂与疫苗工程技术研究中心,厦门 361005
RNAi技术在抗HIV-1治疗研究中已显示出巨大潜力,获得可高效特异抑制HIV-1的RNAi元件是进行相关研究的重要基础。miRNA在抑制和表达方式上相比siRNA具有更多的优势。本研究即探讨构建可高效特异靶向HIV-1的人工miRNA元件。选择以保守性较好的HIV-1 pol基因为靶区筛选高效保守的RNAi序列,设计了16个可靶向pol区高保守区段的RNAi靶点,构建表达载体与HIV-1感染性克隆进行共转染抑制实验,筛选显示pol1026序列兼具高保守性及高抑制效率特点。以天然miR-30a为基础骨架构建了靶向pol1026靶点的人工miRNA元件,通过与HIV-1感染性克隆质粒的共转染抑制实验验证获得了可有效抑制HIV-1表达的人工miRNA元件(miR-1026E)。通过与携带靶序列的报告质粒的共转染实验证明miR-1026E具有良好的靶点特异性。本研究进一步构建了携带miR-1026E表达元件的重组慢病毒,转导MT-4细胞并对转导后细胞进行克隆化筛选,获得稳定整合miR-1026E表达元件的MT-4-miR1026E细胞克隆,该细胞在体外攻毒实验中可高效抑制HIV-1的复制,具有显著的抑制HIV-1的能力。同时应用实时RT-PCR方法检测显示,miR-1026E在细胞中不会影响内源性代表miRNA(miR-181与miR-16)的表达水平和干扰素效应相关基因stat1的表达水平,具有良好的特异性。所获得的可特异高效抑制HIV-1复制的人工miRNA元件可为抗HIV-1研究提供重要参考。
RNA干扰,HIV-1,pol,人工miRNA
Abstract:RNA interference(RNAi)has exhibited huge potentials on anti-HIV-1 therapy research.The obtainment of RNAi element targeting to HIV-1 highly effectively and specifically was crucial for relevant research.Recent reports had described that microRNAs(miRNAs)posses more characteristics of inhibition and expression mechanisms than small interfering RNAs(siRNAs).In this study we explored the construction of artificial miRNA targeting to HIV-1 effectively and specifically.Sixteen siRNAs sequences were selected based on the conserved regions in the HIV-1 pol gene.ShRNA expression vectors were co-transfected withHIV-1 clone pNL4-3 to evaluate the abilities of siRNAs to inhibit HIV-1 expression.The pol1026 sequence was selected from candidates.The target sequence in the stem-loop structure of the well-characterized native miR-30a was replaced with pol1026 sequences, and the artificial miRNA expression vectors were co-transfected with the HIV-1 clone pNL4-3, results showed that HIV-1 can be effectively inhibited by miR-1026E.Target specificity of miR-1026E was confirmed by co-transfection assay with reporter plasmids containing different target sequences.The miR-1026E expression element was then inserted into Lentivirus which was used as a vector to transduce the MT-4 cells, MT-4-miR1026E expressing miR-1026E stably was cloned from transduced cells.The MT-4-miR1026E cell effectively inhibited HIV-1 replication in vitro.And the intracellular miR-181 and miR-16 expression levels and stat1 mRNA levels were not affected by the expression of miR-1026E in MT-4-miR1026E cells.miR-1026E is a promising candidate for future research.
Keywords:RNA interference, HIV-1, pol, artificial miRNA
RNA干扰(RNAi)是细胞内序列特异性抑制靶基因表达的机制,RNAi技术应用于抗HIV-1治疗研究是目前的研究热点[1-2]。获得对HIV-1具有高效抑制能力和高特异性的 RNAi元件是进行相关研究的重要基础。miRNA是细胞内源性调节基因表达的RNA分子,在细胞生长、分化和凋亡等功能中起着重要作用[3]。miRNA基因可由RNA聚合酶II或III启动转录,前体经RNaseIII家族的Drasha和Dicer酶加工形成成熟miRNA,天然miRNA主要通过与靶基因mRNA的3'端非编码区的不完全匹配以翻译抑制方式负调控靶基因的表达[4]。近来研究显示,对天然miRNA进行靶序列区替换可改变miRNA的抑制靶点和抑制方式[5],显示了构建人工miRNA的可行性。由于 miRNA在表达和抑制方式上相比siRNA具有其独特的优势,因此可高效靶向 HIV-1的人工miRNA元件具有更好的应用潜力。HIV-1 pol基因编码病毒复制必需的蛋白酶、逆转录酶和整合酶,序列保守性高。本研究即以HIV-1 pol基因为靶区域,通过分析选择设计可靶向 pol区高保守区段的RNAi靶位点。通过与HIV-1感染性克隆的共转染抑制实验筛选兼具高保守性及高抑制率的 RNAi靶点,并尝试以天然 miRNA为基础骨架构建可靶向该靶点的人工 miRNA元件,并进一步通过脱靶效应检测、抑制特异性分析和体外攻毒实验对本研究构建的人工 miRNA元件的抑制效率和特异性进行分析。本研究获得的可特异高效抑制HIV-1复制的人工miRNA元件为进一步的抗HIV-1研究提供了基础。
1 材料与方法
1.1 质粒和细胞株
MT-4细胞株、E.coli DH5α菌株为本实验室保存。pSUPER质粒购自 Oligoengine公司。携带luciferase基因的报告质粒 pGL3-control购自Promega公司。慢病毒包装质粒(包含pLP1、pLP2和pVSVG)以及293FT细胞购自Invitrogen公司,表达质粒pMACSKK.II购自Miltenyi公司,慢病毒转移质粒pLL3.7由厦门大学生命科学学院韩家淮教授[6]惠赠。
1.2 生化试剂
DMEM、RPMI 1640基础培养基购自Gibco公司。细胞培养用胎牛血清(FBS)购自Hyclone公司。引物由上海生工生物工程有限公司合成。Luciferase Reporter Assay Kit购自Promega公司。DEAE Dextran购自Sigma公司。Trizol、脂质体Lipofectamine 2000、NCod miRNA First-Strand cDNA Synthesis Kit和Platinum® SYBR® Green qPCR SuperMix UDG Kit购自Invitrogen公司。IFN Response Watcher Kit购自TaKaRa公司。限制性内切酶、T4 DNA连接酶、Taq酶购自TaKaRa公司。HIV Elisa检测试剂盒来自北京万泰公司。β-半乳糖苷酶原位染色试剂盒购自碧云天生物技术研究所。核酸提取试剂盒购自北京天根公司。
1.3 siRNA表达载体的构建
以HIV-1NL4-3序列为模板序列,应用Invitrogen和 Genscript公司提供的辅助设计软件设计靶向HIV-1 pol基因的siRNA序列。靶向luciferase基因的 siRNA-luc[7]和靶向 HIV-1 gag基因的 siRNA-gag1607[8]作为实验对照。合成表 1各靶序列所对应的引物,分别经退火处理后连接到经 Bgl II和Hind III双酶切的pSUPER载体,经酶切和测序鉴定获得所需克隆。
1.4 miRNA表达载体的构建
pLLKK表达载体的构建:通过 PCR方法扩增pMACSKK.II质粒上的H-2KK表达框(PCR引物序列分别为:5'-TTTACTAGTCATGTTTGACAGCTT ATCATCG-3'和5'-TTTCTCGAGATACAAGGATCC ATCTACCCTCCTTTTCCACC-3'),PCR产物以Spe I和Xho I双酶切处理后连接到经Xba I和Xho I双酶切处理的慢病毒转移载体pLL3.7,经酶切鉴定后获得pLLKK表达载体。

表1 用于siRNA合成的引物序列Table 1 Primers used for siRNA construction
miRNA表达载体的构建:合成各靶点miRNA序列所对应的引物(表 2),以 ddH2O溶解至终浓度为60 mmol,每对互补引物各3 μL加入24 μL退火缓冲液中,95℃处理 4 min,70 ℃ 1 0 min 后逐步冷却至4℃。退火产物进行 3.0%琼脂糖凝胶电泳,以单正链引物为对照检测是否退火成功。将退火成功的双链片段通过T4 DNA连接酶连接至经BamH I和Xho I双酶切处理的pLLKK表达载体,转化E.coli DH5α感受态菌株,经酶切鉴定后获得目标miRNA表达载体。
1.5 报告质粒的构建
以 HIV-1感染性克隆质粒 pNL4-3为模板扩增pol基因片段(1279~1442 nt),5'端添加 Xba I位点,3'端添加Fse I位点,PCR产物以XbaI和Fse I双酶切后连接到相同酶切处理的 pGL3-control质粒上,经酶切鉴定后获得的质粒命名为pGL3-pol1402。采用相同的方法将带有pol1026靶点序列的pol基因片段(890~1055 nt)插入到pGL3-control获得pGL3-pol1026。采用基因合成方法合成pol基因片段(890~1055 nt),其中的pol1026靶点序列“GTATGTAGGATCTGACTTA”采用同义密码子突变为“GTATGTAGGCAGCGAT TTA”,序列两端分别添加Xba I位点与Fse I位点,采用上述相同方法插入到pGL3-control获得pGL3-pol1026EM。
1.6 共转染实验检测miRNA对HIV-1的抑制效率
24孔细胞培养板中每孔铺入2.0×105个293FT细胞,12 h后进行转染。将miRNA表达质粒(0.3 μg)与HIV-1感染性克隆质粒pNL4-3(0.3 μg)混合后用脂质体Lipofectamine 2000进行转染,转染后72 h收集细胞培养上清进行检测,样品经10倍梯度稀释后用HIV ELISA检测试剂盒检测p24蛋白的活性。
1.7 荧光素酶(Luciferase)表达活性的检测
24孔细胞培养板中每孔铺入2.0×105个293FT细胞,12 h后进行转染。将0.5 μg 待测miRNA表达质粒与0.1μg报告质粒(pGL3-control、pGL3-pol1026或 pGL3-pol1402)混匀后用脂质体Lipofectamine 2000进行转染,转染48 h后收集细胞裂解液用Luciferase Reporter Assay Kit检测荧光素酶活性。
1.8 IFN效应相关基因stat1的检测
用Trizol试剂提取细胞的总RNA,采用TaKaRa公司的 IFN Response Watcher Kit及 Platinum®SYBR® Green qPCR SuperMix UDG Kit进行检测,检测过程参照产品说明书进行。
1.9 RT-PCR检测miRNA水平
用 Trizol试剂提取细胞的总 RNA,用 NCod miRNA First-Strand cDNA Synthesis Kit和Platinum®SYBR® Green qPCR SuperMix UDG Kit检测样品中的成熟miRNA分子的水平。以内源性U6B RNA分子为PCR内参,miRNA特异性检测引物序列如表3所示。

表2 用于miRNA构建的引物序列Table 2 Primers used for miRNA construction
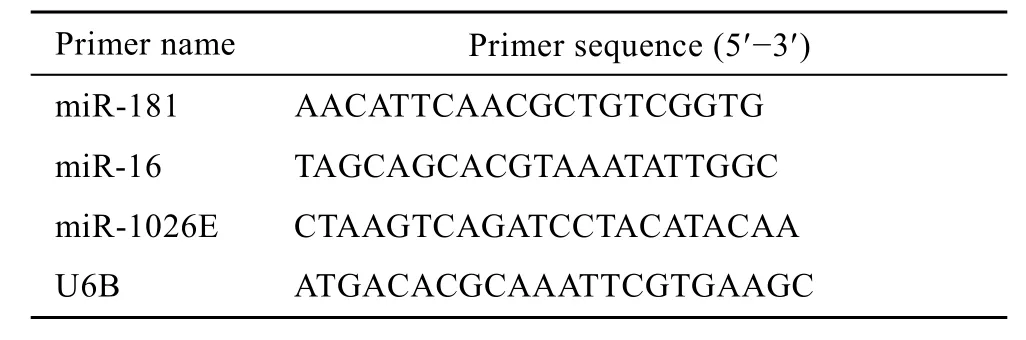
表3 用于real-time PCR检测的引物序列Table 3 Primers used for real-time PCR
1.10 重组慢病毒的构建和鉴定
将 293FT细胞铺于 10 cm细胞培养平皿中(5×106个/皿),6 h后用脂质体 Lipofectamine 2000将18 µg包装质粒混合物、6 µg的慢病毒转移质粒pLLKK-miRNA和6 μg的pSUPER-Drosha(携带靶向细胞Drosha基因的siRNA表达元件,siRNA靶序列为AACGAGUAGGCUUCGUGACUU[9])混合转染,转染12 h后更换为完全培养基,72 h后收集含重组慢病毒的细胞培养上清。重组慢病毒效价鉴定:通过293FT细胞转导实验鉴定感染效价,293FT细胞铺于24孔细胞培养板(5×104个/孔),12 h后分别加入100 µL经10倍梯度系列稀释后的待测病毒样品。培养 48 h后用流式细胞仪(Beckman Coulter EPICS XL)检测孔中表达EGFP的细胞数量,测定重组病毒的滴度(TU/mL)。
1.11 重组慢病毒转导MT-4细胞
将 1×105个 MT-4细胞铺于 24孔细胞培养板中,加入polybrene至终浓度为6 µg/mL,慢病毒用量为MOI=40,室温600×g水平离心60 min,12 h后换液。
1.12 HIV-1NL4-3的制备与鉴定
将 293FT细胞铺于 6孔细胞培养板中(8×105个/孔),6 h后脂质体转染2.4 µg pNL4-3质粒,12 h后更换为完全培养基,72 h后收集含有 HIV-1NL4-3的细胞培养上清。通过梯度稀释后对HIV-1感染报告细胞株 TZM-b1的感染实验鉴定其效价。将TZM-b1细胞铺于96孔细胞培养板(1×104个/孔),6 h后分别加入50 µL经10倍梯度系列稀释后的待测病毒,加入DEAE Dextran至终浓度为20 µg/mL。培养48 h后用β-半乳糖苷酶原位染色试剂盒显色,通过检测样品孔中的蓝斑数量,测定病毒滴度(IU/mL)=蓝斑数量×稀释倍数×20。
1.13 HIV-1NL4-3的体外攻毒实验
将5×105个MT-4细胞或改造后的MT-4细胞铺于24孔细胞培养板中,总培养体积为2 mL,加入HIV-1NL4-3感染12 h后用无血清培养基洗涤2次,继续维持培养,在不同时间点收集细胞培养上清检测p24蛋白的活性。其中,Day1只采集200 µL细胞培养上清用于检测,并补充 200 µL完全培养上清;Day3开始每个时间点均进行1:1细胞传代,具体方法是:在各时间点,用移液器将孔中的MT-4细胞均匀吹散,吸出一半细胞悬液(1 mL),部分取样进行台盼蓝染色后计数获得活细胞浓度 M,其余低速离心收集培养上清用于检测 p24蛋白活性;样品孔中补入1 mL新鲜的完全培养基继续培养。各时间点细胞总数的计算公式为:各时间点细胞理论增殖总数=(样品孔活细胞浓度M×培养体积2 mL)×2N。(其中N为累计传代次数,Day1 未进行传代,Day1和Day3的 N均为 0;Day3及之后各时间点均进行了传代, Day5、Day8、Day11、Day14和Day17的N分别为:1、2、3、4和5)。
2 结果
2.1 靶向HIV-1 pol高保守区的高效RNAi靶点的筛选
本研究对Los Alamos HIV Sequence Database中的625株HIV-1的pol基因序列进行了保守性分析,获得 16个可靶向 pol区高保守区段的 RNAi靶位点。并构建了可靶向上述靶点的siRNA表达载体,分别通过与HIV-1感染性克隆质粒pNL4-3在 293FT细胞中进行共转染实验检测抑制 HIV-1表达的能力。结果如图1所示,本研究设计的靶向上述靶点的 siRNA可显示出不同程度的抑制HIV-1表达的能力,其中靶向pol1026的克隆可显示出较好的抑制效果。同时保守性分析数据也显示,pol1026靶点也具有较好的保守性特征,其在数据库中无点突变和仅有一个突变的序列数占总序列数的百分比分别为45%和40%,二者累计为85%。因此,本研究选择以pol1026为靶点构建人工miRNA元件。
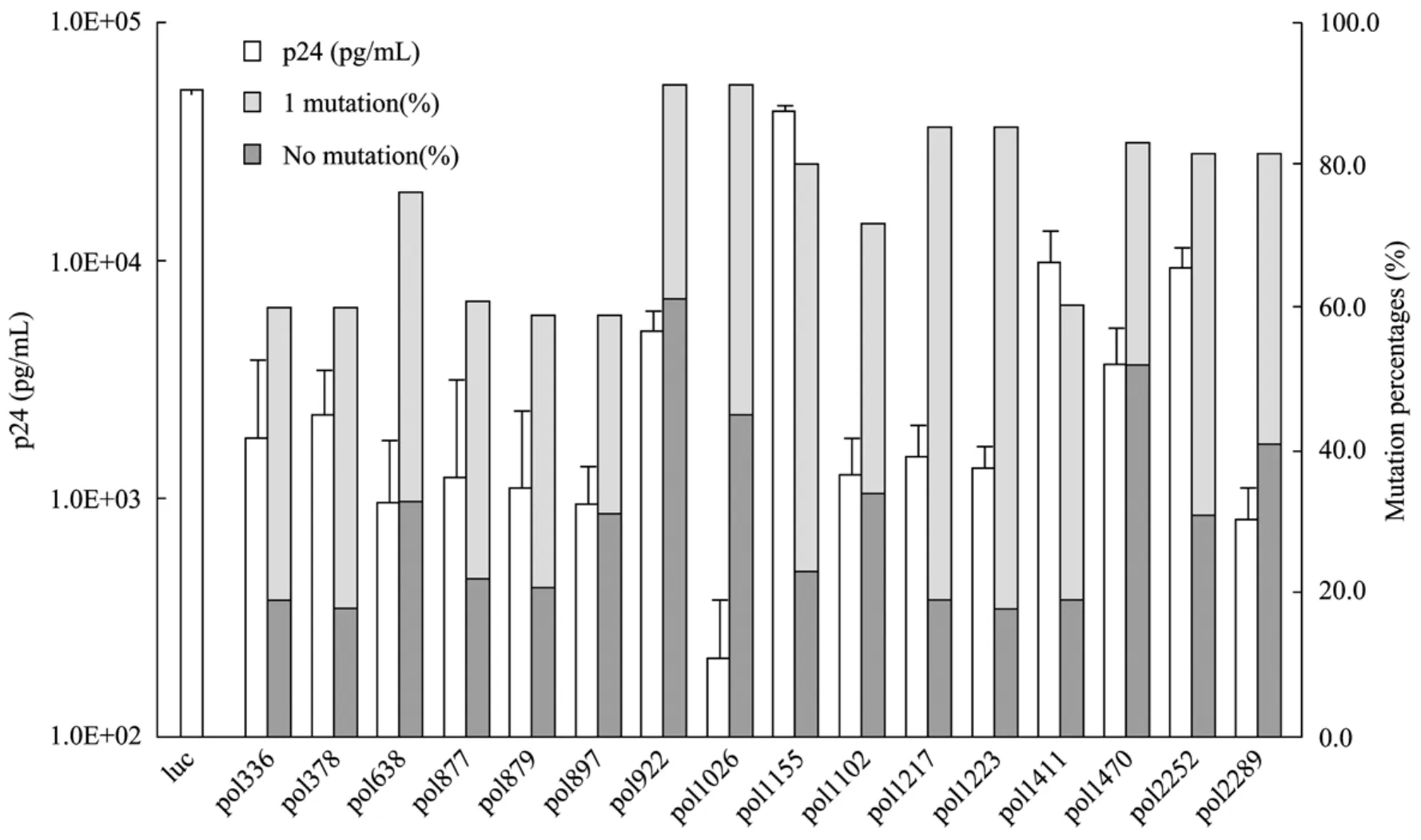
图1 不同RNAi靶点的抑制效率与保守性分析Fig.1 Analysis of inhibition efficiencies and conservation of different RNAi targeting sequences.
2.2 靶向HIV-1 pol1026的人工miRNA的构建与鉴定
miR-30a是目前剪切位点研究较为清楚的天然miRNA[5],因此本研究选择以 miR-30a作为基础模板进行靶向HIV-1 pol1026靶点的人工miRNA的构建。有研究显示靶点序列仅移位1 nt的siRNA即可在抑制效率上表现出较大差别[10],因此本研究设计了2个可靶向HIV-1 pol1026靶点的仅移动1 nt的人工miRNA(miR-1026E、miR-1026F)(图2),分别替换入天然miR-30a的靶序列区构建获得人工miRNA表达载体pLLKK-miR-1026E和pLLKK-miR-1026F。通过与 HIV-1感染性克隆质粒的共转染实验检验本研究设计的人工miRNA是否保持对HIV-1的抑制能力。共转染48 h后检测细胞培养上清中的p24活性,以靶向 LacZ基因的 pLLKK-miR-LacZ作为阴性对照。结果如图3所示,miR-1026E具有较好的抑制HIV-1表达的能力,而 miR-1026F未表现出抑制效果,因此本研究选择miR-1026E进行进一步的分析评价。该结果也说明了靶位区序列的选择对miRNA抑制效果存在较大影响,采用步移方式对筛选针对特定靶位的高效miRNA是必要的。
2.3 miR-p1026E抑制特异性分析
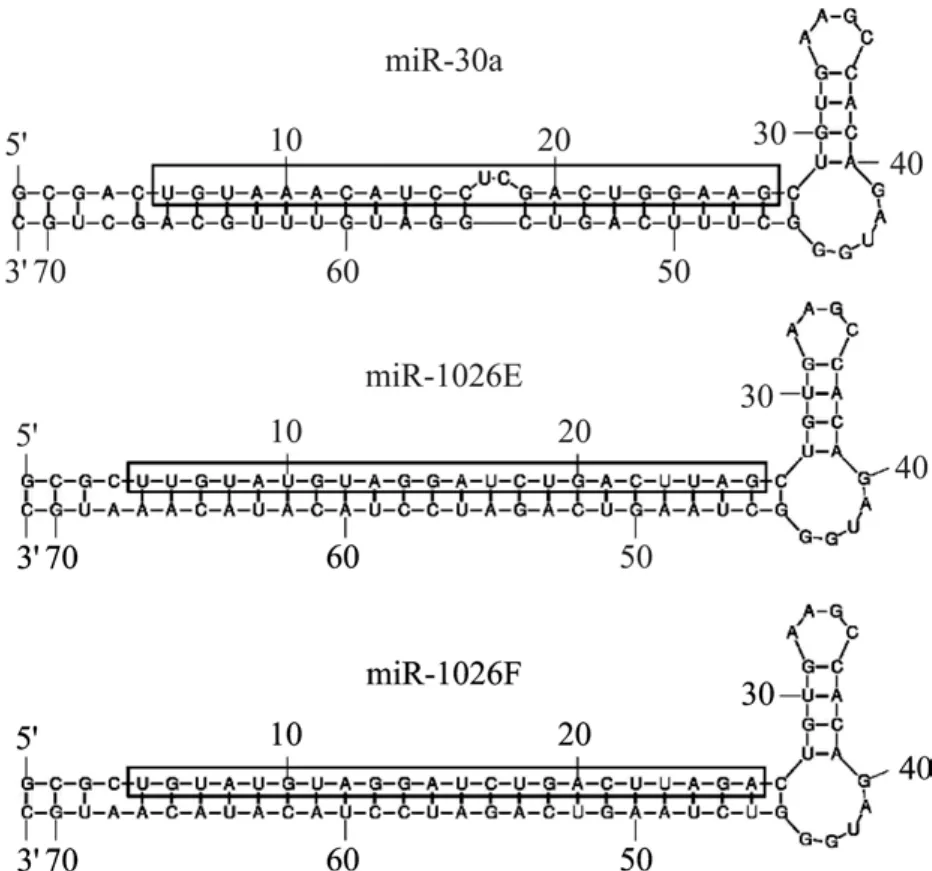
图2 人工miRNA元件的二级结构预测Fig.2 Predicted secondary structures of artificial miRNAs.
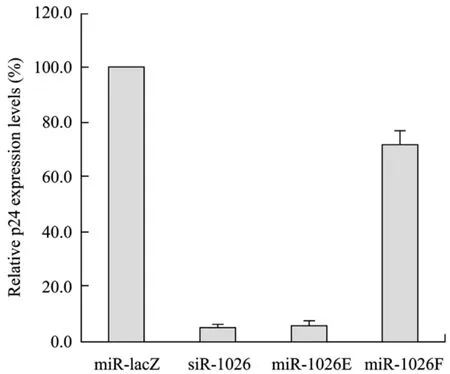
图3 共转染实验检测人工miRNA对HIV-1表达的抑制效率Fig.3 Analysis of HIV-1 p24 protein expression in 293FT cells cotransfected with different artificial miRNA expression vectors and pNL4-3.
本研究进一步对miR-1026E的抑制特异性进行分析。目前较常用的分析特定siRNA或miRNA抑制效率的方法是通过与在报告基因终止密码子后携带 RNAi元件靶序列的报告质粒进行共转染[11]。本研究将miR-1026E靶序列及其相邻序列克隆入荧光素酶表达质粒pGL3-control的终止密码子和ployA之间获得报告质粒pGL3-pol1026,同时也构建了位于HIV-1 pol区但不包含miR-1026E靶序列的对照报告质粒 pGL3-pol1402以及包含经序列突变后的miR-1026E靶序列的对照报告质粒pGL3-pol1026EM。将 miR-1026E表达载体 pLLKK-miR-1026E分别与各报告质粒进行共转染实验,以靶向luc基因的 pSUPER-luc和靶向 LacZ的pLLKK-miR-LacZ载体作为载体对照。共转染 48 h后收集细胞裂解上清并检测荧光素酶活性。结果如图4所示,miR-1026E可有效抑制pGL3-pol1026的表达,不能抑制pGL3-control和pGL3-pol1402以及靶位点区序列突变的 pGL3-pol1026EM表达,表现出较好的抑制特异性。
2.4 重组慢病毒载体的构建与稳定表达miR-1026E的MT-4细胞的获得
来源于HIV-1的重组慢病毒是一种新型基因转移载体,其可有效感染造血系统来源细胞并具有基因整合能力,目前已在 HIV-1的抗病毒研究中得到较为广泛的应用[12]。本研究即应用第3代慢病毒载体系统在Drosha活性抑制的293FT细胞中构建了携带 miR-1026E表达元件的重组慢病毒载体Lenti-miR1026E。本研究使用的重组慢病毒载体同时带有 EGFP表达元件,可用于快速检测重组病毒效价对靶细胞的基因转移效率,并便于检测慢病毒转导后细胞的克隆化效果[13]。MT-4细胞是一种HIV-1受体细胞,可在体外支持T嗜性HIV-1的复制[14]。重组慢病毒载体Lenti-miR1026E对MT-4细胞进行转导,应用有限稀释法进行克隆化筛选,通过筛选检测获得一株可稳定表达报告基因的细胞克隆 MT-4-miR1026E,流式细胞仪检测显示MT-4-miR1026E细胞中 EGFP的表达阳性率接近100%(图5)。本研究进一步通过实时RT-PCR方法检测了miR-1026E在MT-4-miR1026E细胞中的表达水平,检测结果(图 6)显示 miR-1026E 在MT-4-miR1026E细胞中获得了有效表达。
2.5 miR-1026E在MT-4细胞中的脱靶效应检测
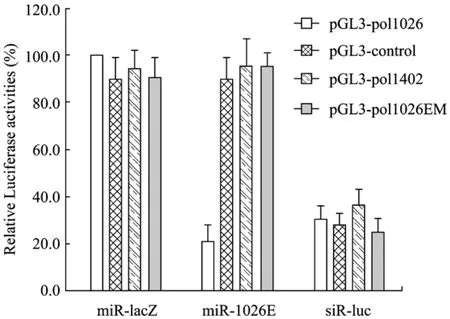
图4 miR-1026E抑制特异性分析Fig.4 Analysis of target specificity of miR-1026E.
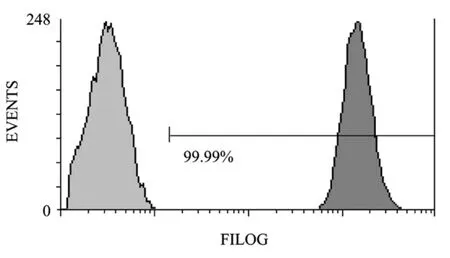
图5 报告基因在细胞克隆MT-4-miR1026E中的表达效率Fig.5 Expession efficiency of reporter gene EGFP in MT-4-miR1026E cells.
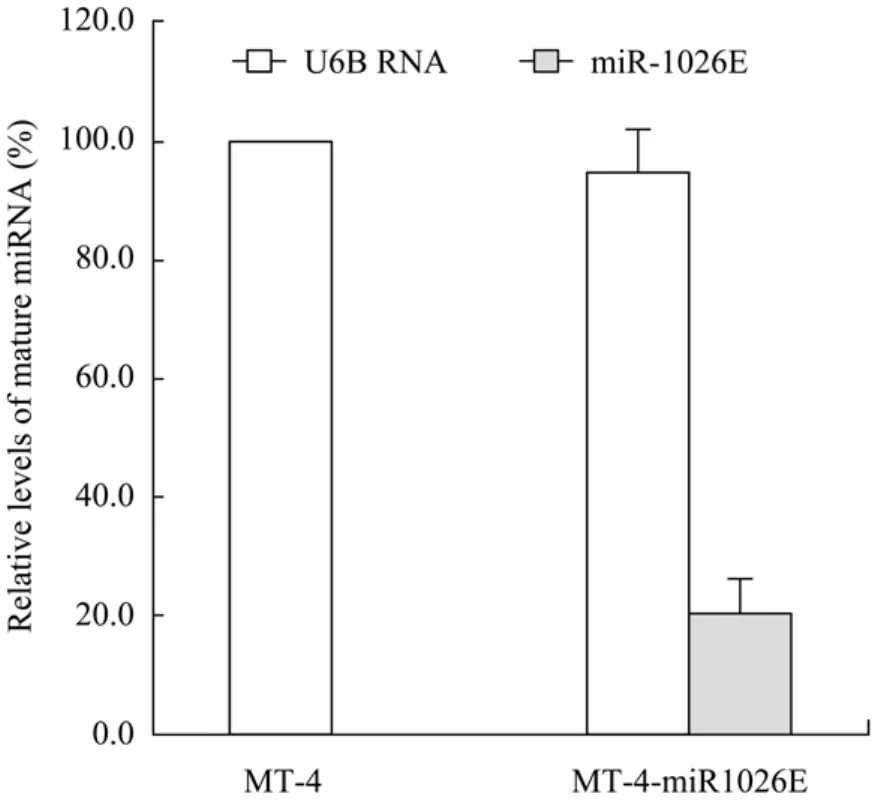
图6 miR-1026E在MT-4-miR1026E细胞中的表达水平Fig.6 Expession level of miR-1026E in MT-4-miR1026E cells.
本研究进一步检测了miR-1026E对细胞内源性代表 miRNA水平和干扰素应答途径的影响。miR-181和miR-16是目前可用于评价脱靶效应的内源性miRNA[15]。通过实时RT-PCR方法检测MT-4-miR1026E细胞和对照 MT-4细胞中 miR-181和miR-16的表达水平,结果显示miR-1026E在细胞中未对内源性 miRNA的表达造成明显影响(图7A)。同时应用实时RT-PCR方法检测了干扰素效应相关基因stat1的mRNA水平,检测结果如图7B所示,与MT-4细胞对照相比MT-4-miR1026E细胞的stat1 mRNA水平未发生明显变化,说明miR-1026E在细胞中不会影响细胞的干扰素应答途径,进一步证明miR-1026E具有良好的抑制特异性。
2.6 稳定表达miR-1026E的MT-4细胞对HIV-1的抑制效果
本研究通过体外攻毒实验检验稳定表达miR-1026E的MT-4细胞对HIV-1复制的抑制效果。HIV-1NL4-3为B亚型、T细胞嗜性的HIV-1病毒,可有效感染并在 MT-4细胞中复制[14]。HIV-1NL4-3以MOI=0.2的剂量对MT-4-miR1026E进行攻毒实验,在感染后不同时间点收集细胞培养上清检测 HIV-1 p24蛋白的活性,同时也检测各时间节点的活细胞数量,以MT-4细胞和未携带MT-4-miR1026E表达元件的重组慢病毒Lenti-H2K转导后的MT-4细胞为对照。结果如图8所示,稳定表达 miR-1026E的MT-4-miR1026E细胞对HIV-1感染可获得显著的抑制效果,并可有效存活和增殖。
3 讨论
RNAi技术已在基因功能研究和疾病治疗等方面显示出巨大的应用潜力,将 RNAi技术应用于抗HIV-1治疗研究是目前的研究热点。获得对 HIV-1具有高效抑制能力和特异性好的 RNAi元件是进行相关治疗方法研究的重要基础。本研究在HIV-1 pol区筛选获得一个具有较好保守性特征和抑制效率的siRNA靶位,以天然miR-30a为基础骨架构建了可特异靶向该靶点的人工miRNA(miR-1026E),通过脱靶效应检测、抑制特异性分析和体外攻毒实验证明该人工miRNA具有高效的抑制效率和特异性,具有良好的应用潜力。
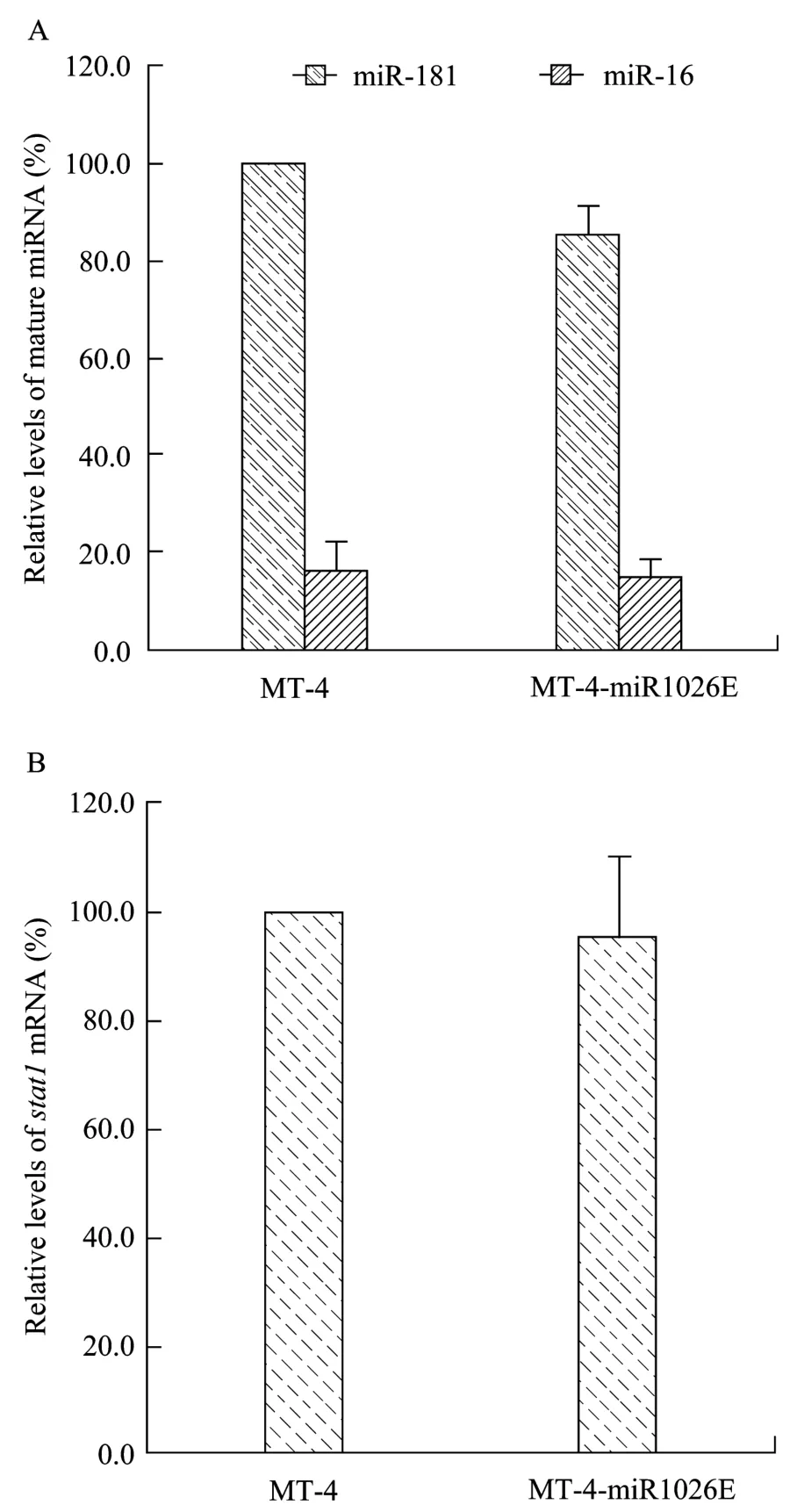
图7 miR-1026E的脱靶效应分析Fig.7 Analysis of off-target effects of miR-1026E.
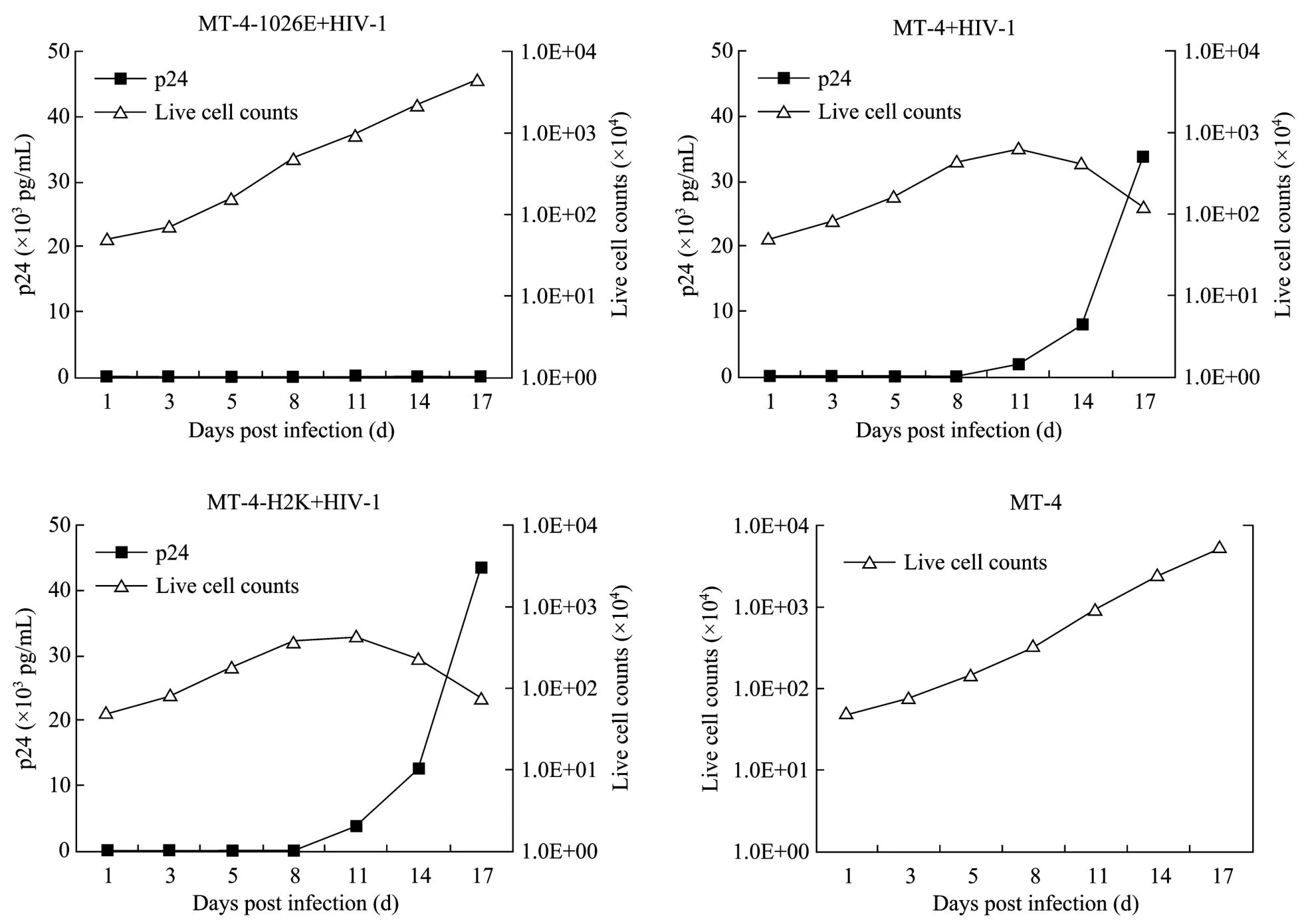
图8 稳定表达miR-1026E的MT-4细胞对HIV-1复制的抑制效果Fig.8 Inhibition of HIV-1NL4-3replication in MT-4-miR1026E cells.
siRNA和miRNA是RNA干扰作用的主要作用成分,其加工和作用机制存在一些差别。siRNA主要由病毒感染或基因转移表达产生的dsRNA或shRNA在细胞中被 Dicer酶所识别并剪切形成siRNA,与RNA诱导的沉默复合物(RISC)结合形成 siRISC,通过序列特异性识别并切割降解靶mRNA[16]。内源miRNA来自于细胞内非编码蛋白基因区转录产生的具有茎环结构的pri-miRNA,经过2个RNase III家族的Drosha和Dicer酶加工后形成成熟的miRNA,与siRNA作用途径相似,miRNA可与 RISC结合形成 miRNA蛋白复合物(miRISC)。miRISC对靶mRNA的抑制方式和miRNA与靶序列的匹配程度有关,天然 miRNA多数是与靶 mRNA序列不完全匹配,通过非降解的翻译抑制方式调控靶基因的表达[4],近来研究显示当 miRNA与靶mRNA序列高度匹配时也可通过类似siRNA的作用机制以剪切降解方式抑制靶基因的表达[17]。目前以miRNA作为特异性的基因表达抑制工具已成为RNAi研究的一个重要方面。
同时,值得注意的是通过miRNA抑制靶基因表达相比siRNA具有一些独特的优势。miRNA抑制靶基因表达方式的多样性使其可能在应对病毒的靶序列突变导致的逃避抑制上具有一定优势。同时,miRNA相比 siRNA在表达载体启动子选择上具有更大的范围和灵活性,miRNA基因可使用RNA聚合酶 II类启动子进行转录,而不限制于 RNA聚合酶III类启动子,可通过使用组织特异性或诱导型启动子以实现 RNAi分子表达的可控性,也有利于避免表达过量而可能引发的对细胞正常功能的影响,利于脱靶效应的控制。本研究构建获得的靶向HIV-1 pol区的人工 miRNA(miR-1026E)即使用了属于RNA聚合酶II类启动子的H2K启动子进行表达,在与 HIV-1感染性克隆的共转染实验和体外攻毒实验中均显示出良好的抑制效果,同时脱靶效应分析显示不会影响细胞正常 miRNA的表达和干扰素应答途径。
目前,构建靶向特定靶点的人工miRNA的策略是将靶序列替换入天然miRNA的靶序列区。有研究显示,在 miRNA的加工成熟过程中,影响 Drosha剪切pri-miRNA的关键因素是其二级结构,如环的大小、发卡长度、自由末端,与miRNA一级序列无关[18],这为在天然 miRNA基础上进行靶序列替换构建人工miRNA提供理论支持。本研究将筛选获得的靶向 HIV-1 pol区的具有高保守性特征和抑制能力的siRNA的靶点序列替换入目前剪切位点较为清楚的 miR-30a的靶序列区,分别构建了两种其靶序列仅位移1 nt的人工miRNA。共转染抑制检测实验结果显示,二者的抑制效率存在很大区别,应用mFOLD软件进行的二级结构预测未显示出具有明显差别,这说明靶位区序列的微小差别即可能会对miRNA的抑制效率产生较大影响,其具体机制将有待后续研究阐明。Brake等在siRNA上的研究也显示出类似现象,靶点序列仅移位1 nt的siRNA即可在抑制效率上存在显著差别[10]。目前对于 miRNA的加工成熟与作用机制仍需要进行更全面和深入地研究,本研究结果可为进一步研究其作用机制提供一定的启示。
慢病毒载体可有效转导非分裂期细胞特别是造血系统来源细胞,是目前包括对 HIV-1感染在内的基因治疗研究中常用的基因转移载体。然而目前常用的慢病毒载体的包装质粒的 gag-pol区来源于HIV-1,因此在构建表达靶向HIV-1 gag-pol区域的RNAi表达元件时会存在抑制自身慢病毒载体构建效率的现象。本研究应用常规方法构建携带miR-1026E表达元件的重组慢病毒的效率与对照病毒相比很低(结果未显示)。因此,本研究在制备重组慢病毒Lenti-miR1026E时在构建体系中增加了靶向Drosha的siRNA表达质粒,可较好的恢复慢病毒的构建效率。该方法可无需对慢病毒包装序列进行同义突变,可为应用于 HIV-1基因治疗研究的慢病毒载体构建提供一个适用的选择。通过慢病毒载体转导筛选获得的 MT-4细胞株在体外高剂量的攻毒实验中具有显著的抑制 HIV-1复制的能力,也进一步证明了本研究获得的特异靶向 HIV-1 pol高保守区的人工miRNA具有良好的应用潜力,可为进一步应用于新型艾滋病治疗方法研究提供重要基础。
REFERENCES
[1]Kanzaki LI, Ornelas SS, Argañaraz ER.RNA interference and HIV-1 infection.Rev Med Virol, 2008, 18(1): 5−18.
[2]Berkhout B, ter Brake O.Towards a durable RNAi gene therapy for HIV-AIDS.Expert Opin Biol Ther, 2009, 9(2):161−170.
[3]Guarnieri DJ, DiLeone RJ.MicroRNAs: a new class of gene regulators.Ann Med, 2008, 40(3): 197−208.
[4]Bartel DP.MicroRNAs: genomics, biogenesis, mechanism,and function.Cell, 2004, 116(2): 281−297.
[5]Lo HL, Chang T, Yam P, et al.Inhibition of HIV-1 replication with designed miRNAs expressed from RNA polymerase II promoters.Gene Ther, 2007, 14(21):1503−1512.
[6]Rubinson DA, Dillon CP, Kwiatkowski AV, et al.A lentivirus-based system to functionally silence genes in primary mammalian cells, stem cells and transgenic mice by RNA interference.Nat Genet, 2003, 33(3): 401−406.
[7]Miyagishi M, Taira K.U6 promoter-driven siRNAs with four uridine 3' overhangs efficiently suppress targeted gene expression in mammalian cells.Nat Biotechnol, 2002,20(5): 497−500.
[8]Chang LJ, Liu X, He J.Lentiviral siRNAs targeting multiple highly conserved RNA sequences of human immunodeficiency virus type 1.Gene Ther, 2005, 12(14):1133−1144.
[9]Kuehbacher A, Urbich C, Zeiher AM, et al.Role of Dicer and Drosha for endothelial microRNA expression and angiogenesis.Circ Res, 2007, 101(1): 59−68.
[10]Brake OT, Konstantinova P, Ceylan M, et al.Silencing of HIV-1 with RNA interference: a multiple shRNA approach.Mol Ther, 2006, 14(6): 883−892.
[11]Liu YP, Haasnoot J, ter Brake O, et al.Inhibition of HIV-1 by multiple siRNAs expressed from a single microRNA polycistron.Nucleic Acids Res, 2008, 36(9): 2811−2824.
[12]Strayer DS, Akkina R, Bunnell BA, et al.Current status of gene therapy strategies to treat HIV/AIDS.Mol Ther, 2005,11(6): 823−842.
[13]Rubinson DA, Dillon CP, Kwiatkowski AV, et al.A lentivirus-based system to functionally silence genes in primary mammalian cells, stem cells and transgenic mice by RNA interference.Nat Genet, 2003, 33(3): 401−406.
[14]Schols D, Este JA, Cabrera C, et al.T-cell-line-tropic human immunodeficiency virus type 1 that is made resistant to stromal cell-derived factor 1alpha contains mutations in the envelope gp120 but does not show a switch in coreceptor use.J Virol, 1998, 72(5): 4032−4037.
[15]Kumar P, Ban HS, Kim SS, et al.T cell-specific siRNA delivery suppresses HIV-1 infection in humanized mice.Cell, 2008, 134(4): 577−586.
[16]Carthew RW, Sontheimer EJ.Origins and mechanisms of miRNAs and siRNAs.Cell, 2009, 136(4): 642−655.
[17]Steiner FA, Hoogstrate SW, Okihara KL et al.Structural features of small RNA precursors determine Argonaute loading in Caenorhabditis elegans.Nat Struct Mol Biol,2007, 14(10): 927−933.
[18]Zeng Y, Yi R, Cullen BR.Recognition and cleavage of primary microRNA precursors by the nuclear processing enzyme Drosha.Embo J, 2005, 24(1): 138−148.
Construction and in vitro antiviral avcitvity of highly effective artifical miRNA targeting to HIV-1 pol
Tong Cheng, Tao Zhang, Yali Zhang, Lihua Wei, Dezhen Xia, Yingbin Wang, Jun Zhang, and Ningshao Xia
National Institute of Diagnostics and Vaccine Development in Infectious Diseases, School of Life Sciences, Xiamen University, Xiamen 361005, China
Received:July 22, 2009;Accepted:November 4, 2009
Supported by:Natural Science Foundation of Fujian Province(No.C0710041), Key Knowledgment Innovation Project of MOE(No.705031), Key Science and Technology Projects of Fujian Province(No.2004YZ01).
Corresponding author:Ningshao Xia.Tel: +86-592-2184113; Fax: +86-592-2181258; E-mail: nsxia@xmu.edu.cn福建省自然科学基金计划(No.C0710041),教育部科学技术研究重大项目培育基金(No.705031),福建省科技重大专项(No.2004YZ01)资助。
