产纤溶酶微生物的筛选鉴定及生长模型的建立*
2010-09-12刘彩平崔堂兵
刘彩平,崔堂兵
(华南理工大学生物科学与工程学院,广东广州,510006)
产纤溶酶微生物的筛选鉴定及生长模型的建立*
刘彩平,崔堂兵
(华南理工大学生物科学与工程学院,广东广州,510006)
使用酪蛋白平板初筛、摇瓶复筛的方法,从9种豆豉中分离到纤溶酶产生菌59株,其中DC-YJ11菌株的酶活较高。通过对其形态特征、生理生化特征结合16S rDNA序列分析,鉴定为枯草芽孢杆菌(Bacillus subtilis)。并对其构建了系统发育树和基于BP神经网络的生长模型BP-YJ11。
枯草芽孢杆菌,纤溶酶,鉴定,BP神经网络
BP神经网络(back propagation neural network)是人工神经网络(artificial neural network,ANN)的一种,具有很强的非线性映射能力,故可以反映十分复杂的非线性关系,在发酵工业中被广泛应用,如培养基的优化[1]、发酵建模过程和控制优化[2],以及发酵分批补料控制[3]等,已成为目前研究的热点之一[4]。
豆豉纤溶酶是一种具有良好溶血栓作用的丝氨酸蛋白酶,与纳豆激酶具有类似的生化性质和生理学功能[5],与目前临床使用的纤溶酶原活化因子类(plasminogen activators)溶栓剂如尿激酶、链激酶、组织型纤溶酶原激活物(tPA)等相比,具有纤溶活性,分子量小,可通过消化道直接吸收,半衰期长[6],不溶解血细胞,安全无毒[7]等优点,是治疗血栓病最有效的药物之一[8]。国内外已有关于筛选出产豆豉纤溶酶的枯草芽胞杆菌的报道,对此类菌的研究主要集中于分子水平改良[9]、发酵条件优化[10]和酶的分离纯化[11]方面,而此类菌生长模型的详细研究未见报道。
本文采用酪蛋白平板初筛-摇瓶复筛的策略,从豆豉中筛选豆豉纤溶酶产生菌并对菌株进行菌种鉴定,建立了基于BP神经网络的生长模型和产酶动力学模型。
1 材料与仪器
1.1 筛选材料
生产于广东、四川、北京、重庆4地的9种豆豉。
1.2 试剂与仪器
牛纤维蛋白原,Sigma公司;凝血酶,中国药品生物制品检定所;标准尿激酶,天普制药厂;W izard Genomic DNA Purification Kit promega公司;普通DNA产物纯化试剂盒,天根公司;其它试剂均为国产分析纯。BPG-9240A紫外可见分光光度计,尤尼科(上海)仪器有限公司;SARTORIUS AG标准型pH计,SARTARIUS公司;SHP-450D型生化培养箱,上海森信实验仪器有限公司;SKYB211B双层特大容量全温恒温培养摇床,广州科桥实验技术设备有限公司。
1.3 培养基
斜面培养基(NA):蛋白胨10 g,牛肉膏3 g,NaCl5g,琼脂15 g,蒸馏水1000 mL,pH7.2。
种子培养基:蛋白胨10 g,牛肉膏3 g,NaCl5g,蒸馏水1000 mL,pH7.4。
初筛培养基:麦芽糖15 g,酪蛋白10 g,琼脂20 g,NaCl 15 g,蒸馏水1000 mL,调节pH至7.2~7.4。
发酵培养基:麦芽糖15 g,大豆蛋白胨20 g,牛肉浸膏1.5 g,KH2PO41 g,K2HPO42.5 g,MgSO4·7H2O 2.5 g,蒸馏水1000 mL,调节pH值至7.0。
2 试验方法
2.1 高产纤溶酶的菌株筛选
2.1.1 初筛
取适量豆豉(6~8粒),加入到盛有灭菌的玻璃珠和95 mL生理盐水的锥形瓶中,振荡20 min,选择3个适宜的连续稀释度涂布于初筛培养基上,平板数不少于10个,倒置37℃培养,18 h后取出。
2.1.2 复筛
参照齐海萍[12]的方法,略有改进。挑取筛选平板上透明圈大的菌株作为复筛菌株。接种于NA培养基上,37℃培养12~16 h,保存于斜面。将上述菌株对应接于种子液,12 h后以2%接种量接入发酵液(50/250 mL三角瓶)中。37℃,往复式摇床180 r/min培养72 h后,将发酵液离心,取上清液,稀释不同倍数,测定其纤溶酶酶活,挑取高产菌株。
2.1.3 纤溶酶酶活的测定方法
根据Astrup等[13]的方法稍加修改。8.1 mL 1%的琼脂糖于55℃时加入8.1 mL 0.3%的纤维蛋白原溶液及0.65 mL凝血酶(1BP/mL),混匀倒入平板中,静置冷却。在平板上用直径3 mm的打孔器打孔,每孔点样10μL样品,于37℃保温18 h,测定水解圈的大小,计算其面积,以尿激酶作标准曲线。
2.2 菌株鉴定
2.2.1 菌株形态观察及生理生化特性测定
菌株的形态特征:在NA培养基上30℃、培养48 h,革兰氏染色观察细菌形态。
生理生化特征:参照东秀珠等[14]的方法对DCYJ11菌株进行生理生化特征鉴定。
2.2.216S rDNA序列测定与比对
利用W izard GenomicDNA Purification Kit提取细菌基因组DNA,以芽孢杆菌16SrDNA保守序列设计特异性引物P16 S L:5′-GA GA G T T TGA T C C T G G C T C A G-3′、P16 S R:5′-G C CCCCGTCAATTCCTTTGAG-3′,进行16S r DNA的PCR扩增。PCR产物经分离纯化后,送至上海博尚生物技术有限公司测序,然后将所测得16S rDNA序列与Gen-Bank数据库中的16S rDNA序列进行比对。
2.3 建立基于BP神经网络的菌株的生长模型
2.3.1 生物量测定方法
采用比浊法,以发酵液在660 nm处,测定细胞悬浮液的吸光度OD值,以未接种的培养基等量稀释作对照。
2.3.2 pH测定方法
用pH计测定。
2.3.3 还原糖测定方法
利用DNS法测定方法。
2.3.4 纤溶酶酶活的测定方法
参照2.1.3方法。
2.3.5 生长曲线的测定
按照2.3.1~2.3.4的方法,每2~10h取1次样。
2.3.6 BP神经网络编程
使用matlab 2006中文版实现神经网络工具。具体设计:采用3层神经网络结构,输入层、输出层各一个神经元。训练函数是LM,网络训练以均方误差作为目标误差,训练次数为epoch=500次[15]。以取样时间和取样时还原糖含量构成的矩阵为输入,菌体生物量(OD660)和酶活的1/10(E/10)组成的数组为输出建立生长模型。使用20组实验数据训练神经网络BP-YJ11,同时使用5组非训练样本的实验数据,验证网络的合理性。
3 结果与分析
3.1 纤溶酶产生菌株的筛选
经初筛和复筛,共获得产纤溶酶的59株菌,其中DC-YJ11的酶活为89.67U/mL(发酵液),酶活力最高。
3.2 菌株鉴定
3.2.1 DC-YJ11菌株的形态特征
DC-YJ11革兰氏染色阳性,细胞形态呈短杆状,芽胞中生或偏端生,椭圆或柱形,菌体多成对或链状。
3.2.2 菌落的形态特征
在NA培养基上,30℃培养48 h。单菌落为圆形或近圆形,直径为4~5 mm,浅黄色,不透明,菌落表面略粗糙,无光泽,边缘稍不整齐。
3.2.3 生理生化特征
DC-YJ11的生理生化特征实验结果见表1。

表1 DC-YJ11菌株生理生化特征
综合以上DC-YJ11的形态特征和生理生化特征,参照《常见细菌系统鉴定手册》,DC-YJ11与枯草芽孢杆菌(B acillus subtilis)非常相近。
3.2.4 DC-YJ1116S rDNA序列分析
测出的DC-YJ11菌株的16S rDNA序列全长为1579 bp,将测得的序列与数据库中已注册的16S rDNA序列用BLAST程序进行比较分析,在前100个比对结果中相似性都在99%以上,其中绝大多数为枯草芽孢杆菌。将Genbank中与DC-YJ11菌株相似性较高的8个菌株,以蜡样芽胞杆菌为外群,构建系统进化树,结果如图1所示。
形态特征、生理生化特征结合16S rDNA序列比对结果,鉴定DC-YJ11菌株为枯草芽孢杆菌(Bacillus subtilis)。
3.3 基于BP神经网络的菌株的生长模型的建立
3.3.1 DC-YJ11的生长曲线
DC-YJ11生物量、产物以及培养基pH和还原糖的变化趋势如图2所示,细菌在接种到发酵培养基以后,几乎没有延滞期,细菌迅速生长,0~16 h为对数生长期,此后细胞进入生长稳定期。发酵后期,生物量值略有下降;并且纤溶酶活曲线和还原糖的曲线有明显的相关关系。
3.3.2 BP神经网络BP-YJ11的训练结果
使用训练样本(见表2),对神经网络BP-YJ11进行训练,仿真后模型的拟和值见表2,图3为均方误差曲线。由图3可知,神经网络BP-YJ11在训练约100次后,均方误差达到1.096×10-2。
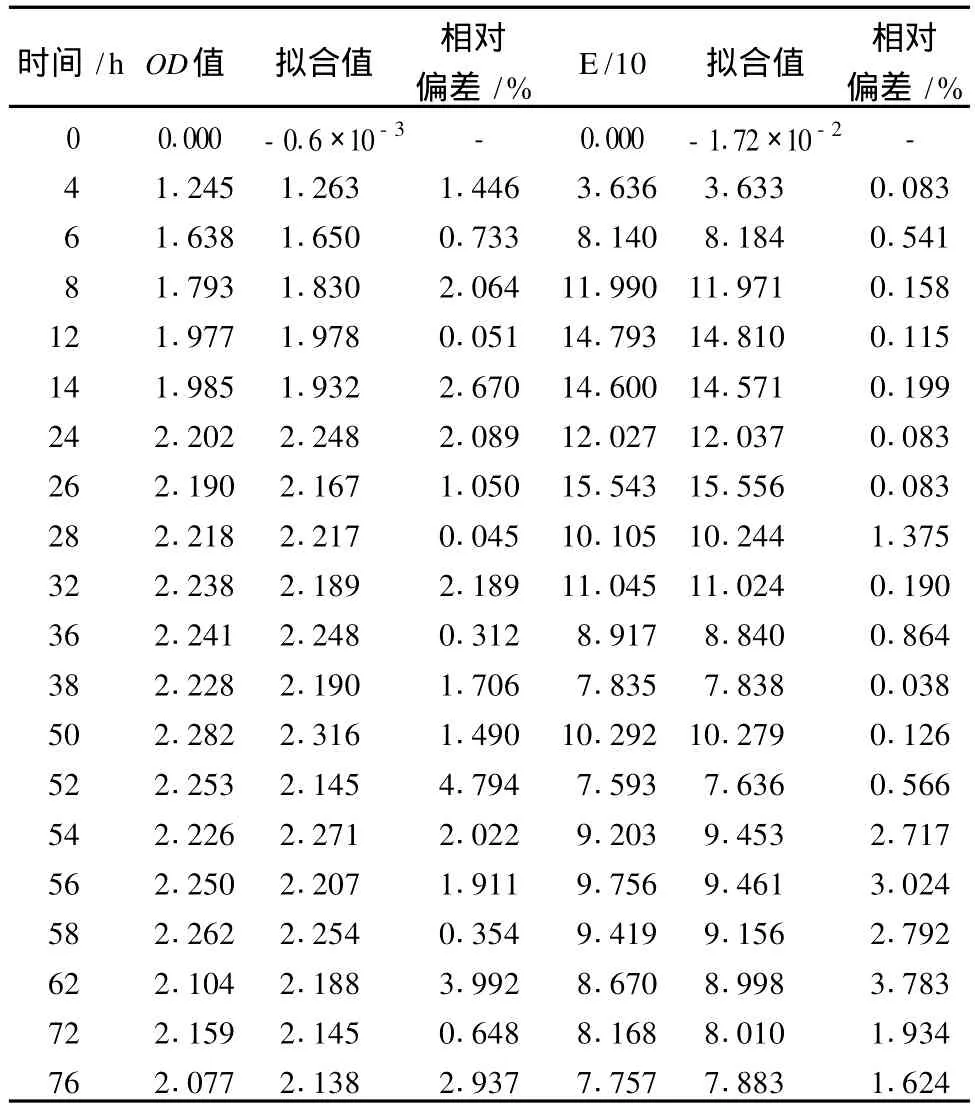
表2 训练样本
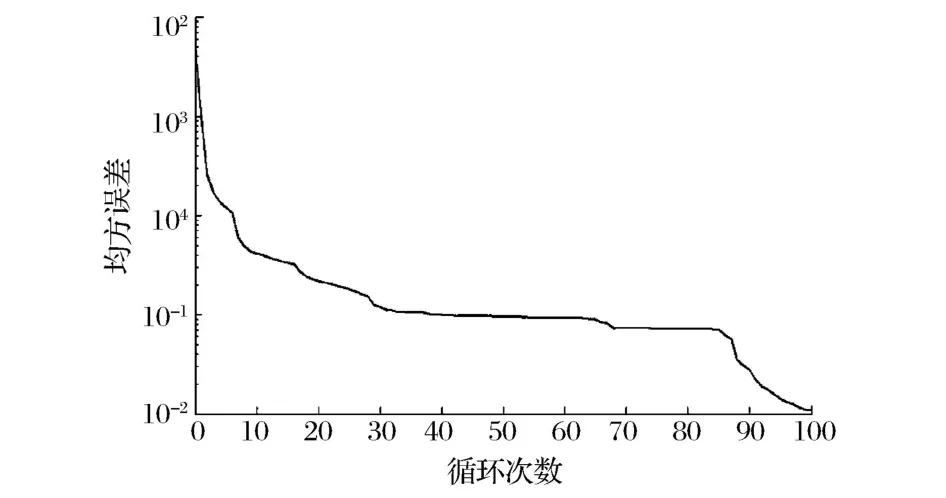
图3 均方误差曲线
3.3.3 BP神经网络的验证及模型可信度检验
将验证点输入训练好的神经网络BP-YJ11,仿真,通过实测值与模型拟和值对比分析BP-YJ11的准确性,结果见表3。实验值与拟和值相比较,均方误差0.336,相对误差在5.90%以内,优于贾翠英等6.25%的结果[16]。进一步利用零点平均偏差(zero mean deviation)分析[17]检验模型的可信度。每个变量平均偏差计算如下:
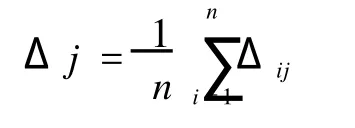
式中,n是实验数据点的个数,Δij是实验值与模型计算值的差值。
每个变量的方差Sj计算如下:
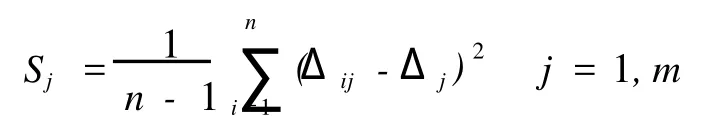
式中m为变量个数,定义统计量λ为:
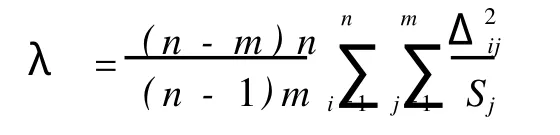
λ服从Fm,n-m分布,在本文中,λ服从F2,3分布,经计算λ的值为29.688,小于查表得到30.82(置信度99%),这说明,本文所建立的模型具有一定的可信度,所建立的模型的拟合度是可行的[18],能够满足试验要求,可以用该网络模型进行枯草芽孢杆菌液态发酵产纤溶酶产酶进程曲线和菌体生长曲线的绘制。
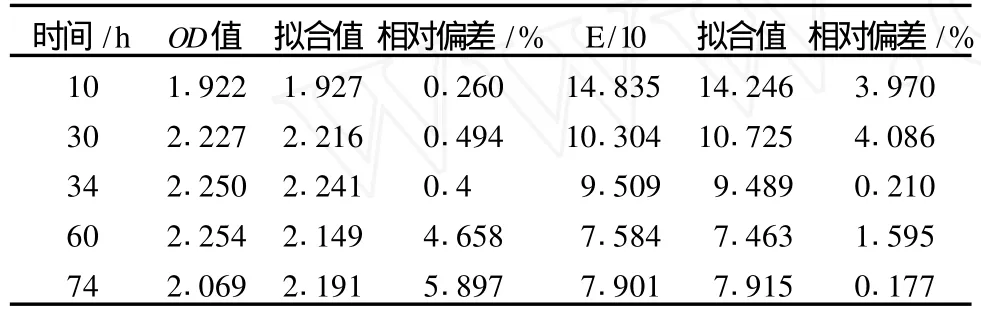
表3 验证样本及检验
4 小结与讨论
近年来不断有相关文献报道从豆豉中分离到具有纤溶活性的菌株,一般都是采用直接点纤维蛋白平板法来进行菌株的筛选。本实验利用酪蛋白平板作为初筛从中国传统发酵食品豆豉中筛选到1株迅速产纤溶酶的菌株DC-YJ11,经生理生化试验及16S rDNA分析,鉴定为枯草芽孢杆菌(B acillus subtilis),证明了使用酪蛋白平板法筛选产豆豉纤溶酶微生物的可行性,为今后筛选产纤溶酶微生物节约成本;同时对该菌建立了基于BP神经网络的高仿真生长模型,为通过人工控制发酵条件来快速获得大量的纤溶酶提供了参考,为这类溶栓药物早日在临床上大量推广奠定了基础。
[1]潘丽军,张婵婵,姜绍通,等.菜籽饼粕水解液为氮源细菌发酵产L-乳酸的发酵培养基优化[J].食品科学,2007,34(10):313-316.
[2]葛爱冬,隋青美,王斌鹏.混合神经网络建模方法在青霉素发酵过程中的应用[J].山东轻工业学院学报,2006(3):30-33.
[3]杨梁,熊伟丽,徐保国.基于粒子群算法的基因工程菌发酵分批补料控制[J].计算机测量与控制,2005(3):255-257.
[4]彭锋,李安明.BP神经网络和遗传算法在红发夫酵母培养基优化中的应用[J].应用与环境生物学报,2008,14(6):834-837.
[5]张仁怀,王海燕,谈宁馨,等.豆豉溶栓酶基因在毕赤酵母中的表达及其产物的纯化[J].应用与环境生物学报,2005,11(5):623-626.
[6]FAN Xiao-dan,XIE Xiu-zhen,GUO Yong.Breeding of fibrinolytic enzyme-producing strains isolated from Douchi[J].Life Science Research,2006,10(1):45-49.
[7]彭勇,张义正.豆豉溶栓酶产生菌的筛选及其酶学性质的初步研究[J],高技术通讯,2002,12(2):30-34.
[8]Wang CT,JiBP,LiB.Purification and characterization of a brinolytic enzyme of Bacillus subtilis DC33,isolated from Chinese traditional Douchi[J].J Ind Microbiol Biotechnol,2006,33:750-758.
[9]Kho CW,Park S G,Cho S,et al.Confir mation ofVpr as a fibrinolytic enzyme present in extracellularproteins of Bacillus subtilis[J].Protein Expression and Pur iWcation 2005,39:1-7.
[10]孙月娥,钱和.豆豉纤溶酶生产菌液体发酵条件的优化[J].河南工业大学学报:自然科学版,2005,26(6):43-46.
[11]Lu Feng-xia,Sun Li-jun,Lu Zhao-xin,et al.Isolation and Identification of an Endophytic Strain EJS-3 ProducingNovel Fibrinolytic Enzymes[J].CurrentMicrobiology,2007,54:435-439.
[12]齐海萍,钱和,王璋,等.高活性豆豉纤溶酶产生菌出发菌株的筛选[J].中国调味品,2004(2):12-15.
[13]Astrup T,Mullertz S.The fibrin plate method for est imating fibrinolytic activity[J].Arch Biochem Biophys,1952,40:346-351.
[14]东秀珠,蔡妙英,等.常见细菌系统鉴定手册[M].北京:科学出版社,2001:43-65.
[15]刘彩平,崔堂兵.基于BP神经网络的产β-葡萄糖苷酶的真菌生长模型研究[J].安徽农业大学,2009,37(34):16913-16915.
[16]贾翠英,张玉辉,常景玲.酵母菌生长与产人参皂苷β-葡萄糖苷酶的动力学模型[J].郑州大学学报:理学版,2009,41(2):108-111.
[17]Vandana Goswami,Srivastava A K.Fed-batch propionic acid production by Propioni-bacterium acidipropionici[J].Biochemical Engineering Journal,2000(4):121-125.
[18]李春喜.生物统计学(第二版)[M].北京:科学出版社,2001.
ABSTRACTIn this research,fifty-nine strains producing fibrinolytic enzyme were successfully isolated from nine kinds ofDouchi by using casein in plate at first and secondly by both fibrin plates and plas ma plates,and the oneDCYJ11 was the highest fibrinolytic enzyme producing strain.Itwas identified asBacillus subtilisbymorphological,biochemical,physiological characters analysis,and 16S rDNA sequence analysis.This study also constructed a phylogenetic tree and established its growth model based on BP NeuralNetwork.
Key wordsBacillus subtilis,fibrinolytic enzyme,identification,BP NeuralNetwork
Isolation and Identification of the Fibrinolytic Enzyme Producing Stra in and Its Growth Model
Liu Cai-ping,Cui Tang-bing
(College of Biology Science and Engineering,South China University of Technology,Guangzhou 510006,China)
硕士研究生(崔堂兵副教授为通讯作者)。
*国家自然科学基金项目(30970044)
2010-02-10,改回日期:2010-03-30
