利用ERIC-PCR和PCR-DGGE技术分析喂服枯草芽孢杆菌肉鸡肠道菌群的多样性
2010-04-17潘康成陈正礼崔恒敏
潘康成 陈正礼 崔恒敏 冯 兴 盛 琴 赵 爽
(四川农业大学动物医学院,动物疫病与人类健康四川省重点实验室,雅安 625014)
动物肠道中寄生着种类繁多和数量巨大的微生物,正常情况下,它们之间以稳定的比例关系与宿主肠道相互依存又相互影响,形成动态的微生态平衡。肠道正常菌群与肠道免疫系统的形成和功能、肠上皮细胞的生长有密切关系,并可以阻挡病原微生物的入侵[1-3]。过去对肠道菌群结构的认识常采用传统的微生物培养技术,但由于肠道中约占总数90%的微生物不能在体外培养[4],因此不能客观评价肠道中微生物群落的变化规律。肠道细菌基因间保守重复序列(ERIC)-聚合酶链反应(PCR)是近年来被广泛用于菌群结构及种群变化的一种分子生物学方法,该方法快速、简洁、不依赖于纯培养,可直接根据样品ERIC条带的分布、数量和亮度等信息去分析,灵敏度、可重复性和可靠性较好[5-6]。基于16S rDNA为基础的分子生物学技术发展,不依赖微生物培养的分子生物技术为研究肠道微生态提供了简便而快捷的方法[7],其中变性梯度凝胶电泳(DGGE)技术近年来逐渐被应用于检测猪、牛、羊和鸡等肠道主要细菌类群的多态性[8-12]。目前,关于用传统的分离培养方法检测枯草芽孢杆菌对肉鸡消化道菌群的影响已有报道。闫凤兰等[13]报道肉仔鸡饲喂枯草芽孢杆菌D1制剂后可显著增加肠道中乳杆菌的数量和极显著减少沙门氏菌的数量,但对大肠杆菌没有影响。Teo等[14]报道肉鸡饲喂每t含有109个枯草芽孢杆菌的饲粮,对肠道乳杆菌和双歧杆菌的数量没有影响,但可使有害菌如大肠杆菌和梭菌的数量减少1~2 lg(CFU/g)。但利用ERIC-PCR和PCR-DGGE分析动物饲喂芽孢杆菌之后肠道菌群结构及多样性未见报道,因此本试验拟通过对肉鸡喂服枯草芽孢杆菌Pab02菌悬液后,采用ERIC-PCR和PCR-DGGE方法研究肠道菌群结构的多样性,为该菌的应用提供理论基础。
1 材料与方法
1.1 芽孢杆菌菌悬液的制备
枯草芽孢杆菌Pab02菌种(本实验室保存)经活化后,接种于2 000m L营养肉汤中,37℃、160 r/m in振荡培养18 h。5 000 r/m in、4℃离心20m in,收集菌体,无菌PBS(0.05m ol/L、pH 6.5)调整活菌浓度至1×109CFU/m L,4℃保存备用。
1.2 动物分组及饲养
1日龄的艾维茵肉鸡30只常规饲养至28日龄,体重(1 050.5±56.1)g,随机分成2组,即试验组和对照组。试验组按2 m L/kg BW喂服菌悬液,每日08:00和20:00各1次,连续3 d。对照组喂服等量的灭菌PBS液。2组鸡分别饲养于不同鸡舍内,自由采食不含任何抗生素和微生态制剂的饲料,自由饮水。舍内温度控制在20~25℃。
1.3 肠道菌群总DNA的提取
上述鸡只,在停服芽孢杆菌3 d后即34日龄时,每组取5只,颈动脉放血处死,各取十二指肠、空肠、回肠和盲肠内容物约1 g,称重并置于50m L灭菌离心管内,按照张保卫等[15]的方法抽提细菌基因组,基因组定容于50μL TE缓冲液中溶解,-70℃保存备用。
1.4 ERIC-PCR和PCR-DGGE分析
1.4.1 ERIC-PCR反应
引物ERIC-1:5′-ATGTAAGCTCCTGGGGATTCAC-3′和ERIC-2:5′-AAGTAAGTGACTGGGGTGAGCG-3′。PCR反应体系(25μL):2.5μL 10×Taq Buffer(500mmol/L KCl、100mm ol/L Tris-HCl、15 mmol/L MgCl2),4μL dNTP mixture(各2.5 mmol/L),0.5μL Taq酶(5 U/μL),20 pm ol/L上下游引物各1.5μL,2μL DNA模板,补ddH2O至25μL。反应条件:95℃7min,30个循环(95℃30 s、52℃1m in、65℃8 min),65℃16min。
1.4.2 16S rRNA V3区PCR扩增
根据大肠杆菌16S rRNA基因V 3片段(339~539)设计一对引物:HDA 1-GC(5′-CGCCCGGGGCGCGCCCCGGGCGGGGCGGGGGCACGGGGGGACTCCTACGGGAGGCAGCAGT-3′)和HDA 2(5′-GTATTACCGCGGCTGCTGGCAC-3′)。PCR反应体系(25μL):2×Long Taq M aster M ix 10μL,引物(10 pm ol/μL)各1μL,模板DNA 1μL,ddH 2O补足25μL。PCR扩增条件:94℃4m in,30个循环(94℃30 s、58℃30 s、72℃2m in),最后72℃延伸10 m in。
1.4.3 肠道菌群DNA的ERIC-PCR/PCRDGGE图谱分析
以提取的各肠段细菌DNA作为模板,按上述反应体系和反应条件进行扩增,ERIC-PCR产物经2%琼脂糖凝胶电泳并拍照。16S rRNA V3区PCR扩增产物进行DGGE分离,反应条件为:10%聚丙烯酰胺凝胶,变性剂浓度梯度为35%~65% (7 mo l/L尿素和40%去离子甲酰胺为100%变性剂),电泳采用D-code DGGE系统(Bio-Rad),电泳缓冲液为1×TAE,200 V预电泳5m in,85 V电泳16 h,电泳结束后进行硝酸银染色,并将DGGE条带用凝胶成像系统拍照。使用分析软件NTSYS 2.1对ERIC-PCR/PCR-DGGE指纹图谱进行条带计数,用D ice方法计算相似性指数Cs,用UPGM A (unweighted pair group mean average)进行聚类。
1.5 割胶回收条带并克隆测序
将DGGE图谱上出现的特征性条带割胶回收,用无菌去离子水冲洗3次,放入1.5 m L离心管中,加入100μL TE缓冲液4℃过夜,取1μL为模板,按1.4.2方法再次扩增16S rDNA V3区,扩增产物做DGGE电泳确认正确性,重复1~3次,直至在图谱上得到单一条带。然后,取1μL回收的纯化DNA为模板,采用不带GC发夹的引物(HDA 1:5′-ACTCCTACGGGAGGCAGCAGT-3′,HDA 2:5′-GTATTACCGCGGCTG CTGGCAC-3′)按1.4.2方法扩增V3区。PCR产物经pMD18-T载体连接,转化E.coliDH 5α感受态细胞,在含有氨苄青霉素的LB固体培养基上选择具有氨苄青霉素抗性的白色转化子,采用T载体通用引物进行菌落PCR检测,送菌液至上海生工生物技术公司进行测序,将序列在GenBank数据库中进行同源性分析。
2 结 果
2.1 肉鸡肠道菌群ERIC-PCR/PCR-DGGE指纹图谱及条带数
ERIC-PCR扩增结果显示,对照组中扩增条带以盲肠数量最多,其次是空肠和回肠,十二指肠最少;肉鸡喂服芽孢杆菌Pab02菌悬液后肠道的条带数都有一定程度增加,而且条带的亮度普遍提高,说明肠道菌群多样性丰富和种群密度有所增加,条带数量以空肠最多,其次是十二指肠,最后是盲肠和回肠。16 S rRNA V 3区的PCR-DGGE扩增结果,对照组中扩增条带以盲肠数量最多,其次是空肠和回肠,十二指肠最少;肉鸡喂服芽孢杆菌Pab02菌悬液后,各肠段的条带数及特征性条带均明显多于对照组,说明肉鸡口服芽孢杆菌Pab02后肠道菌群多样性丰富和种群密度有所增加,与ERIC-PCR结果相似,不同的是PCR-DGGE的条带数量比ERICPCR更多,肉鸡口服芽孢杆菌Pab02后,肠道中的条带数量顺序分别为盲肠、空肠、十二指肠、回肠(图1和表1)。
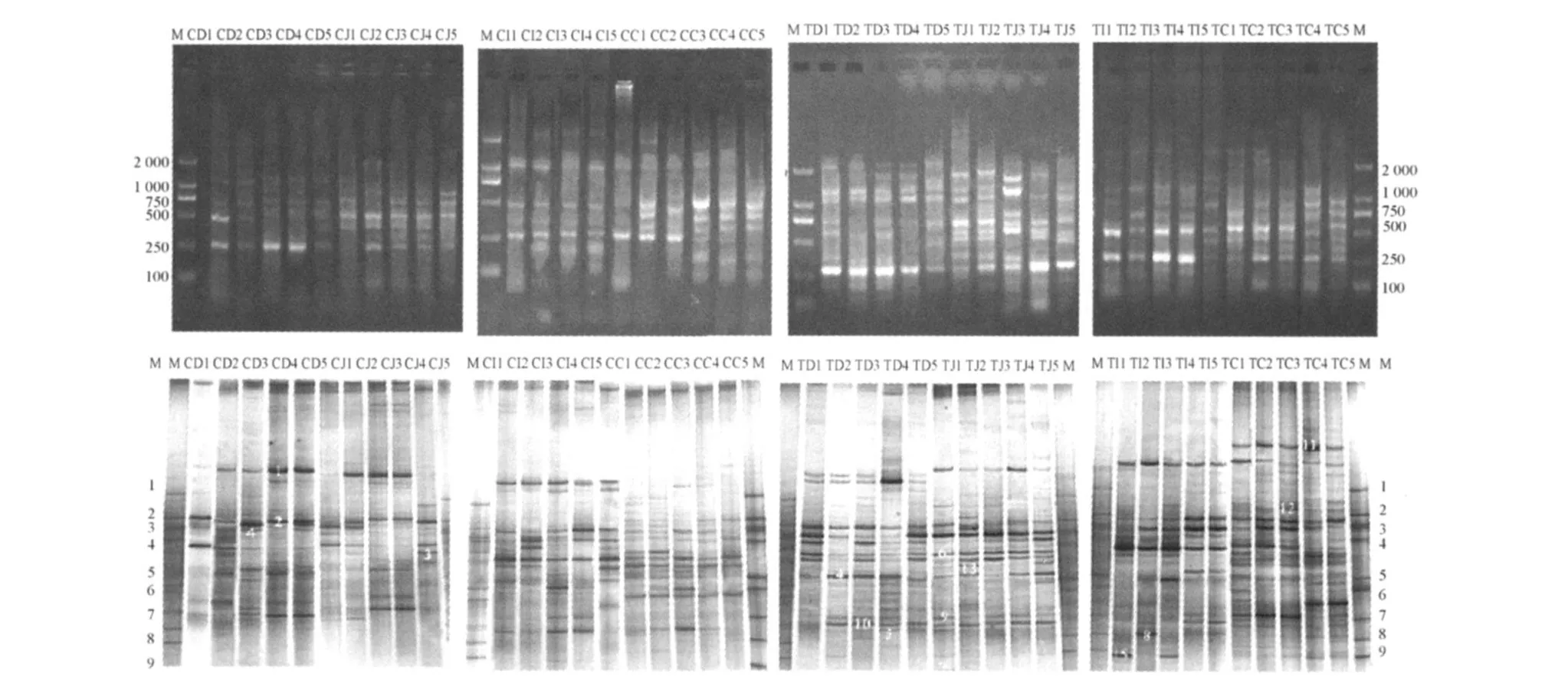
图1 肉鸡肠道菌群ERIC-PCR(上图)和PCR-DGGE(下图)指纹图谱Fig.1 ERIC-PCR(above)and PCR-DGGE(dow n)fingerprint of intestinalm icroflora of broilers

表1 肉鸡肠道菌群ERIC-PCR和PCR-DGGE条带数Table 1 The number of ERIC-PCR and PCR-DGGE bands of intestinalm icroflora of broilers(x—±SD)
2.2 ERIC-PCR/PCR-DGGE检测肉鸡肠道菌群多样性
ERIC-PCR条带聚类相似性分析结果,对照组中的十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为87.5%、70.8%、75.0%和72.9%,各肠段总相似性为52.5%;肉鸡连续喂服芽孢杆菌Pab02菌悬液3 d,并停服3 d后,各肠段肠道菌群组成相似性分别为88.9%、69.4%、81.5%和72.2%,各肠段总相似性为62.7%;2组之间肠道菌群的相似性为53.2%。PCR-DGGE条带聚类相似性分析结果,对照组中的十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为63.9%、57.2%、71.0%和65.8%,各肠段总相似性为46.9%;肉鸡连续喂服芽孢杆菌Pab02菌悬液3 d,并停服3 d后,各肠道菌群组成相似性分别为66.7%、60.0%、67.2%和82.2%,各肠段总相似性为59.4%。2组之间肠道菌群的相似性为53.2%(图2)。
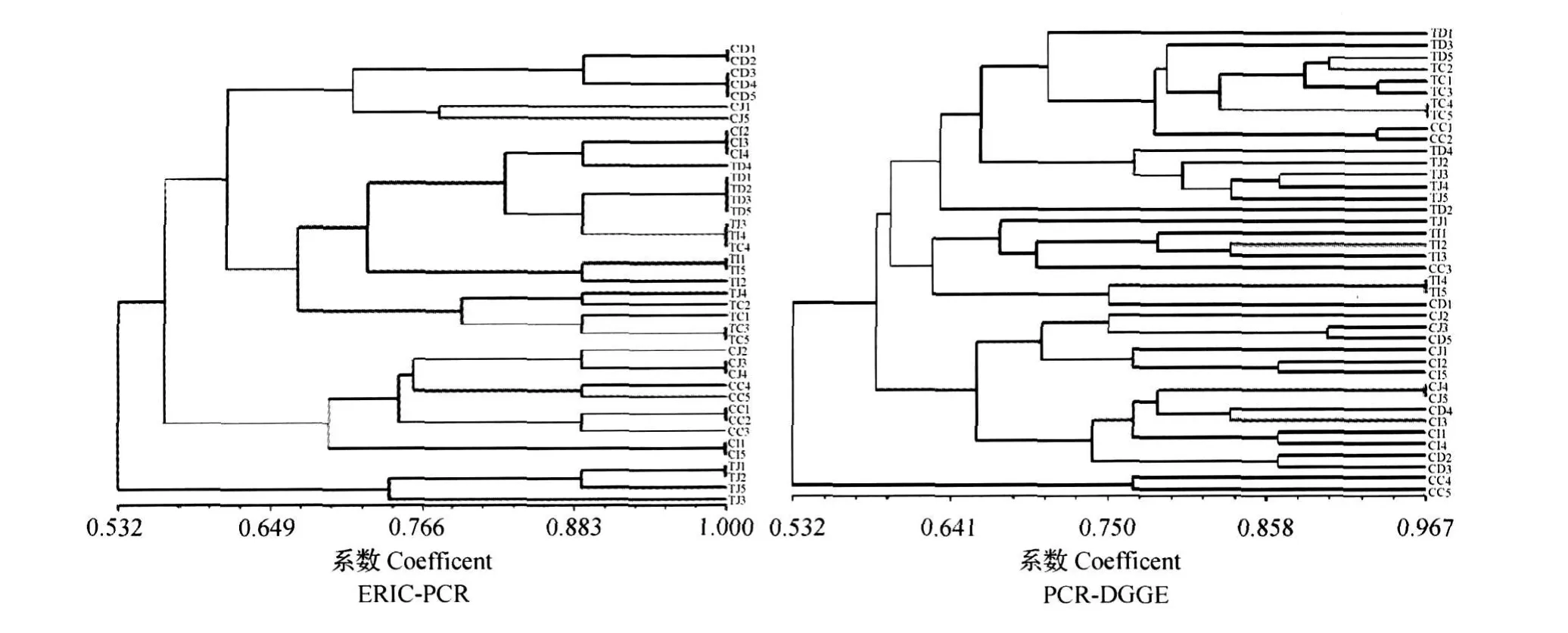
图2 肉鸡肠道菌群ERIC-PCR和PCR-DGGE条带相似性分析Fig.2 Sim ilarity analysis of intestinalm icro flora of broilers by ERIC-PCR and PCR-DGGE
2.3 DGGE图谱对应的肠道优势条带DNA序列分析
分别选取对照组和试验组的DGGE图谱上出现的7个共同条带和8个试验组的特异性条带,共15条特征性条带割胶回收,进行测序,在NCBI中进行BLAST对比,结果见表2。这些回收测序条带中,第7号、第11号、第12号和第15号条带序列为未培养的细菌(均为试验组条带),其余条带都是已知的基因库中收录种属,而鉴定出来的细菌以乳杆菌居多,表明乳杆菌在肉鸡肠道中属于优势菌群。

表2 DGGE图谱对应的单个条带DNA序列Tab le 2 Sequences analysis of DNA recovered from single band in DGGE fingerp rints
3 讨 论
3.1 肉鸡喂服芽孢杆菌Pab02后对肠道菌群结构多样性影响
由于肠道正常菌群的功能越来越受到重视,肠道菌群结构多样性和种群的动态变化也逐渐成为人们研究的热点。过去,对肠道菌群结构多样性和种群变化的认识,主要通过传统的选择性培养细菌和分型鉴定。由于动物肠道内的菌群种类繁多,数量巨大,人们对许多细菌的生存状况及其生理特性缺乏了解,绝大部分的细菌种群无法通过纯培养的方法获得,从而导致低估菌群结构的多样性[4,16]。
ERIC-PCR和PCR-DGGE是近年来被广泛用于菌群结构及种群变化的分子生物学方法,与其他方法相比,其灵敏度、可重复性和可靠性更好,省时省力,克服了传统培养方法费时、费力等缺点。本试验对正常对照组肉鸡各肠段的菌群DNA进行ERIC-PCR检测,各肠段内的菌群DNA扩增条带以盲肠数量最多,其次是空肠和回肠,十二指肠最少。用PCR-DGGE检测时其肠道菌群DNA扩增条带数量、排序顺序与ERIC-PCR方法检测的结果相似。与倪学勤等[17]、Gong等[18]采用PCRDGGE检测蛋鸡的肠道菌群DNA扩增结果相似。肉鸡喂服芽孢杆菌Pab02菌悬液3 d,并在停服3 d后分别采集各肠段内容物提取基因组,分别进行ERIC-PCR和PCR-DGGE分析,结果2种方法证明肉鸡口服枯草芽孢杆菌Pab02后各肠段的条带数及特征性条带均明显多于对照组,差异显著,说明枯草芽孢杆菌Pab02具有增加肉鸡肠道菌群多样性和种群密度的功能。与Pieper等[19]报道仔猪断奶前3天和断奶后3天口服植物乳杆菌菌悬液后,采用PCR-DGGE检测仔猪第25天(断奶时)、第28天、第33天和第39天的肠道菌群多样性的变化一致。
在对电泳条带的聚类分析中,ERIC-PCR检测对照组中的十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为87.5%、70.8%、75.0%和72.9%,各肠道总菌群的相似性为52.5%;而肉鸡连续喂服枯草芽孢杆菌Pab02菌悬液3 d后,十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为88.9%、69.4%、81.5%和72.2%,各肠道总菌群的相似性为62.7%。PCR-DGGE检测对照组中的十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为63.9%、57.2%、71.0%和65.8%,各肠道总菌群的相似性为46.9%;而肉鸡连续喂服枯草芽孢杆菌Pab02菌悬液3 d后,十二指肠、空肠、回肠和盲肠的肠道菌群组成相似性分别为66.7%、60.0%、67.2%和82.2%,各段肠道总菌群的相似性为59.4%。结果说明2组肠道细菌之间存在着一定种群的差异性,但2组之间肠道总菌群的相似性ERIC-PCR和PCR-DGGE检测均为53.2%,仍然存在着比较高的相似性。在正常情况下,虽然肠道内细菌种类和数量会随着肠道内环境的改变和年龄的增长等生理因素而有所改变,但在一定的年龄阶段,肠道正常菌群是机体内一种相对稳定的微生态系统,与宿主相互依赖又相互作用,保持着动态的微生态平衡[1-2]。进一步对DGGE分离的7条对照组和试验组的共性条带,以及8条试验组的特异性条带进行克隆测序,结果表明,在34日龄的肉鸡肠道中主要以乳杆菌为主要菌群,饲喂芽孢杆菌Pab02后能提高肉鸡肠道乳杆菌种群的丰度和密度。
3.2 ERIC-PCR和PCR-DGGE检测肉鸡肠道菌群结构的比较
在复杂的样品如肠道内容物或粪便,电泳分析ERIC-PCR和PCR-DGGE扩增得到的不同生态系统微生物群落指纹图谱,不同系统中有很多条带具有相同的电泳特性,但其序列组成信息不同。对于ERIC-PCR,每个种或菌株因染色体上带有的ERIC重复序列个数和片段大小不同,加上琼脂糖凝胶电泳的分辨率小,可能导致指纹图谱上多种细菌的特异性条带在同一位置上相互叠加。基于细菌16S rRNA/rDNA基因序列中可变区(V3区或V6~V8区)配对的引物,扩增出细菌16S rRNA/rDNA基因的部分序列,并通过DGGE得到分离,电泳条带的数目、位置和强度可反映样品中微生物的种类及细菌的相对构成比例,反映样本的微生物结构多样性,它的分辨精度高,可以检测到1个核苷酸水平的差异,因此该技术在揭示自然界微生物区系的遗传多样性和种群差异方面具有独特的优越性。Y ang等[20]通过ERIC-PCR扩增产物后,采用琼脂糖凝胶电泳(AGE)和DGGE对比分析鹅肠道菌群结构多样性,研究结果表明,肠道菌群DNA电泳条带数量差异性与采用的PCR技术关联小,而与电泳的凝胶类型有关,因此凝胶电泳的选择比PCR过程更重要。通过本试验2种检测方法均可证明肉鸡口服枯草芽孢杆菌Pab02后可以提高肠道菌群的丰度和种群密度,对照组和试验组各肠段总菌群的相似性均为53.2%,但从电泳指纹图谱和统计条带数量分析PCR-DGGE明显优于ERIC-PCR。
4 结 论
①采用ERIC-PCR和PCR-DGGE对比检测肉鸡口服芽孢杆菌Pab02后肠道菌群结构的多样性,2种检测方法结果相同,肉鸡口服枯草芽孢杆菌Pab02后可以提高肠道菌群的丰度和种群密度,对照组和试验组各肠段总菌群的相似性均为53.2%。
②从电泳指纹图谱和统计条带数量分析PCRDGGE明显优于ERIC-PCR。
[1] 杨汝德,李武明,许燕滨.动物和人类的肠道菌群的形成及意义[J].微生物学杂志,1998,18(1):52-55.
[2] Steinhof f U.W ho controls the crow d?New findings and old questions about the intestinalm icroflora[J]. Immunology Letters,2005,99(1):12-16.
[3] Gophna U,Sommer feld K,Gophna S,et al.Differences betw een tissue-associated intestinal m icrof loras of patients w ith Crohn's disease and ulcerative colitis [J].Journal Clinical M icrobiology,2006,44(11): 4 136-4 141.
[4] Am ann R I,Ludw ig W,Sch leifer K H.Phy logenetic identification and in situ detection of individual m icrobial cells without cultivation[J].M icrobiological Reviews,1995,59(1):143-169.
[5] Peng Y,Jin J,W u C,et al.Orthogonal array design in optim izing ERIC-PCR system for fingerprinting rat's intestinal m icroflora[J].Journal of App lied M icrobiology,2007,103(6):2 095-2 101.
[6] Cao S Y,W ang M S,Cheng A C,et al.Com parative analysis of intestinalm icrobial community diversity between healthy and orally infected duck lings w ithSalmonellaenteritidis by ERIC-PCR[J]. W orld Journal o f Gastroenterology,2008,14(7): 1 120-1 125.
[7] Zoetendal E G,Collier C T,Koike S,etal.Molecu lar eco logical analysis o f the gastrointestinalm icrobiota:a review[J].The Journalo f Nutrition,2004, 134(2):465-472.
[8] Sim pson JM,M cCracken V J,Gaskins H R,et al. Denaturing gradient gel electrophoresis analysis of 16S ribosom al DNA amp licons tomonitor changes in fecal bacterial populations of w eaning pigs after introduction ofLactobacillus reu teristrain MM 53[J]. App lied and Environm ental M icrobiology,2000,66 (11):4 705-4 714.
[9] Kocherginskaya S,Am inov R I,W hite B A.Analysis of the rumen bacterial diversity under two different diet conditions using denaturing gradient gel electrophoresis,random sequencing,and statistical ecology approaches[J].Anaerobe,2001,7(3):119-134.
[10] Zhu X Y,Zhong T,Pandya Y,et al.16S rRNA based analysis o f m icrobiota from the cecum of broiler chickens[J].App lied and EnvironmentalM icrobio logy,2002,68(1):124-137.
[11] Metzler BU,Vahjen W T,Baumgärtel T,et al.Changes in bac terial populations in the ileum of pigs fed low-phosphorus diets supp lemented with different sources of fermentab le carbohyd rates[J].Animal Feed Science and Technology,2009,148(1):68-89.
[12] 淡瑞芳,龙瑞军,张海涛,等.藏系绵羊瘤胃细菌区系的季节动态分析[J].动物营养学报,2009,21 (5):798-802.
[13] 闫凤兰,卢 峥,朱玉琴.肉仔鸡饲喂枯草芽孢杆菌(Bacillus subtilis)效果的研究[J].动物营养学报, 1996,8(4):34-38.
[14] Teo A Y,Tan H M.Evaluation o f the perform ance and intestinal gutm icroflora of broilers fed on cornsoy diets supp lemented w ithBacillus subtilisPB6 (CloSTAT)[J].The Journal o f Applied Pou ltry Research,2007,16(3):296-303.
[15] 张保卫,魏辅文,李 明,等.大熊猫和小熊猫粪便DNA提取的简易方法[J].动物学报,2004,50(3): 452-458.
[16] Amann R I,Strom ley J,Devereux R,et al.M olecular and m icroscopic identification of sulfate-reducing bacteria in multispecies biofilms[J].App lied and Environmental M icrobio logy,1992,58(2):614-623.
[17] 倪学勤,Joshua G,HaiY,等.采用PCR-DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J].畜牧兽医学报,2008,39(7):955-961.
[18] Gong J,SiW,Forster R J,et al.16S rRNA genebased analysis of m ucosa-associated bacterial community and phylogeny in the chicken gastrointestinal tracts:from crops to ceca[J].FEMS M icrobio logy Ecology,2007,59(26):147-157.
[19] Pieper R,Janczyk P,U rubschurov V,et al.Effect of a single oraladm inistration ofLactobacillus p lantarumDSMZ 8862/8866 before and at the time point of w eaning on intestinalm icrobial communities in piglets[J].International Journal of Food M icrobiology,2009,130(3):227-232.
[20] Yang J L,Cheng A C,W ang M S,et al.New strategies for electrophoresis analysis of enterobacterial repetitive intergenic consensus PCR in animal intestinal m icroflora[J].Journal of M icrobiological Methods,2009,77(1):63-66.
*Correspond ing au thor,p rofessor,E-m ail:cuihengm in2008@sina.com
(编辑 何丽霞)
