大珠母贝微卫星DNA标记的分离与筛选
2010-03-15喻达辉黄桂菊
柳 明,喻达辉黄桂菊
(1.农业部海水养殖生态与质量控制重点开放实验室,中国水产科学研究院 南海水产研究所,广东 广州510300; 2.上海海洋大学 水产与生命学院,上海 201306)
大珠母贝(Pinctada maxima),俗称白蝶贝,属于软体动物门双壳纲异柱目珍珠贝科珠母贝属(Pinctada)。主要分布于热带海域,如澳大利亚、菲律宾、马来西亚、印度尼西亚等地[1]。大珠母贝经济价值很高,其肉(闭壳肌)质味道鲜美、营养丰富,堪称为宴席佳品。它的壳形独特,珍珠层厚而有美丽光泽,是名贵的工艺原料。用大珠母贝养殖出来的珍珠颗粒大,色泽好,价格高,既是贵重的装饰品,又是名贵药材。我国的大珠母贝资源本来就很少,属于国家二级保护动物,过度利用使其更处于濒危状态,因此不能直接用野生大珠母贝进行育珠生产而必须使用人工繁殖个体。人工养殖贝育成的珍珠一致性和质量都较高。然而大珠母贝的海区养殖一直难以成功,幼贝出现大规模死亡现象,导致其珍珠养殖不能实现产业化开发。其原因除了养殖环境恶化外,种质资源的衰退也是重要原因之一。因此开展遗传选育,改良种质是有效开发大珠母贝资源的重要途径。在开发过程中,对选育群体进行遗传多样性分析和监测是十分必要的。微卫星DNA是遗传多样性分析和分子标记辅助育种最适遗传标记之一。但大珠母贝的微卫星标记数量不多。Evans等[2]报道了6个多态标记和Smith等[3]报道了8个微卫星标记,远不能满足应用的需要,因此必须开发大量的微卫星标记。
本实验采用生物素-磁珠富集法和探针杂交相结合的方法,构建了大珠母贝的微卫星富集文库,筛选大珠母贝的微卫星标记,为大珠母贝的遗传育种、保护生物学和种群遗传多样性等研究奠定基础。
1 材料与方法
1.1 材料来源
用于微卫星分离的大珠母贝样品于2008年8月采集于海南三亚,用于微卫星多态引物筛选的大珠母贝30个样品个体于2007年7月采集于海南三亚养殖群体,取闭壳肌保存于95%酒精中。
1.2 实验方法
1.2.1 基因组DNA提取
按照传统的酚-氯仿法[4]提取基因组DNA,溶于100 µL灭菌双蒸水中,-20℃保存待用。
1.2.2 基因组DNA酶切、接头连接与PCR扩增
基因组DNA用内切酶MseI酶切3 h,20 µL反应体系包括:10×NEB Buffer2 2 µL,100×BSA 0.2 µL,MseI (10 U/µL) 0.5 µL,20 mg/LDNA 模板 3 µL,ddH2O 14.3 µL。将酶切产物与双链接头(接头A序列为:5′-TAC TCA GGA CTC AT-3′,接头 B 序列为:5′-GAC GAT GAG TCC TGA G-3′) 于 16 ℃连接过夜,反应体系如下:10 mmol/L T4 DNA ligase缓冲液2 µL,50 mmol/L 接头 1.8 µL,酶切产物 11 µL,400 U/µL 的T4 DNA ligase 0.2 µL。连接产物用简并引物MseI-N[5′-GAT GAG TCC TGA GTA A(N)-3′]进行 PCR 扩增,PCR 反应总体积 20 µL,包括 10×PCR buffer 2.0 µL,10 mmol/L dNTPs 0.4 µL,50 mmol/LMseI-N 0.2 µL,1UTaq酶,10×连接产物 2 µL。反应程序如下:95 ℃预变性5 min,然后进行PCR循环。循环参数为:95℃变性30 s,53 ℃退火1 min,72 ℃延伸1 min,循环数进行梯度筛选,分别做 14、17、20、23、26个循环,以确定最佳循环数,最后72 ℃延伸5 min。
1.2.3 探针杂交与磁珠富集
将杂交缓冲液70 µL、PCR产物25 µL和生物素标记的(CA)15探针(10 µmol/L)5 µL 混合,于 65 ℃杂交1 h。结束后,将磁珠加入到100 mL杂交液中,室温温浴 30 min,移去杂交液并保存。用 400 µL的TEN1000[5]于室温下洗涤磁珠 3次,每次 5 min,保留第 3次的洗脱液。用 400 µL 的 0.2×SSC(含0.1%SDS)洗3次,每次5 min,保留第3次的洗脱液。用 400 µL TEN1000 再洗 5 min,保存洗脱液。加 100 µL TE重悬磁珠,100 ℃水浴10 min,将DNA从磁珠-探针-DNA混合物中分离,水浴结束后将离心管置于磁架上,吸出含有 DNA片段的洗脱液,然后立刻置于冰上。用100 µL TE再次重悬磁珠,保存洗脱液。以上述6管洗脱液为模板,用引物MseI-N进行PCR扩增,将 PCR扩增产物于 1.5%的琼脂糖凝胶上电泳,检测富集效果。富集效果好的样品用 DNA Gel Extraction Kit(AXYGEN)回收纯化PCR产物,纯化产物按常规方法与pMD20-T载体16 ℃连接过夜,将连接产物转化到大肠杆菌感受态细胞中进行培养,构建微卫星富集文库。
1.2.4 富集文库的筛选、测序与引物设计
挑选单克隆,接种到含有氨卞青霉素的LB培养基中,37 ℃振荡培养2 h,菌液用载体通用引物和探针序列引物进行PCR筛选鉴定,经1.5%琼脂糖凝胶中电泳检测,选取符合条件的候选克隆送上海生工生物技术有限公司进行DNA测序。利用Clustal X[6]对序列特征进行分析,并用引物设计软件primer 5.0对所得的微卫星序列设计引物。
1.2.5 微卫星多态引物的筛选
将引物稀释成10 µmol/L,取海南三亚养殖群体共 30个个体,进行 PCR 扩增,体系如下:10×PCR buffer 2.0 µL,10 mmol/L dNTPs 0.4 µL,10 µmol/L正、反向引物各 0.5 µL,5 g/LTaq酶 0.2 µL,20 mg/L DNA模板2µL,ddH2O 14.4 µL。通过调整退火温度,来获得最佳PCR条件,将PCR产物用1.5%琼脂糖凝胶进行电泳,EB染色,将有目的条带出现的PCR产物再进行5%(w/v)聚丙烯酰胺凝胶电泳,筛选具有多态性的引物。
1.3 数据统计与分析
统计每个位点的等位基因数量(A),用 Pop-Gen1.32软件[7]来计算群体的有效等位基因数(Ae)、观测杂合度(Ho)和期望杂合度(He)、Hardy-Weinberg平衡偏离指数(D)以及卡方检验(PHW)。根据Botstein等[8]的公式计算每个微卫星位点的多态性信息含量(CPI),计算公式如下:
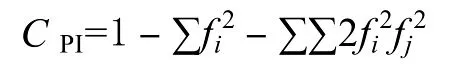
式中fi和fj分别表示某一位点第i个和第j个等位基因在群体中的频率。
2 结果与分析
2.1 微卫星DNA的分离筛选
用内切酶MseI对基因组 DNA进行酶切后用1.5%琼脂糖凝胶电泳检测,发现酶切片段主要集中在200~1 000 bp,符合构建基因组文库的要求。通过探针杂交,磁珠吸附,清洗和洗脱,PCR扩增,载体连接和转化等步骤,构建了大珠母贝基因组微卫星富集文库,共获得近 2 000个阳性克隆,随机挑选1 200个克隆进行PCR检测。选取具有稳定扩增带的阳性克隆298个进行测序,分析测序结果获得了211个含有重复次数大于或等于6的微卫星序列。211个序列共含有 251个微卫星位点,一般每个克隆含有1~3个位点,其中1个克隆含有6个位点,微卫星序列长度大小范围为 101~787 bp。对所得微卫星位点进行分类,发现(CA/GT)n微卫星重复序列204个,占了绝大多数,此外还发现了AG、AT、GC、TC、CAA、AGG、GATA、GACA、GTGC、GACG等重复类型。根据Weber[9]提出的微卫星序列分类标准,在所获得的微卫星位点中,完美型159个,占63%; 非完美型80个,占32%; 复合型12个,占5%。
2.2 微卫星引物的筛选
根据包含微卫星位点的序列,用引物设计软件primer 5.0设计引物,共设计引物90对,随机挑选了其中30对送去上海生物工程有限公司合成。通过优化PCR反应条件,筛选出21对能够稳定扩增的引物。将有目的条带出现的PCR产物再进行5%(w/v)聚丙烯酰胺凝胶电泳,筛选出10对多态性引物(表1)。
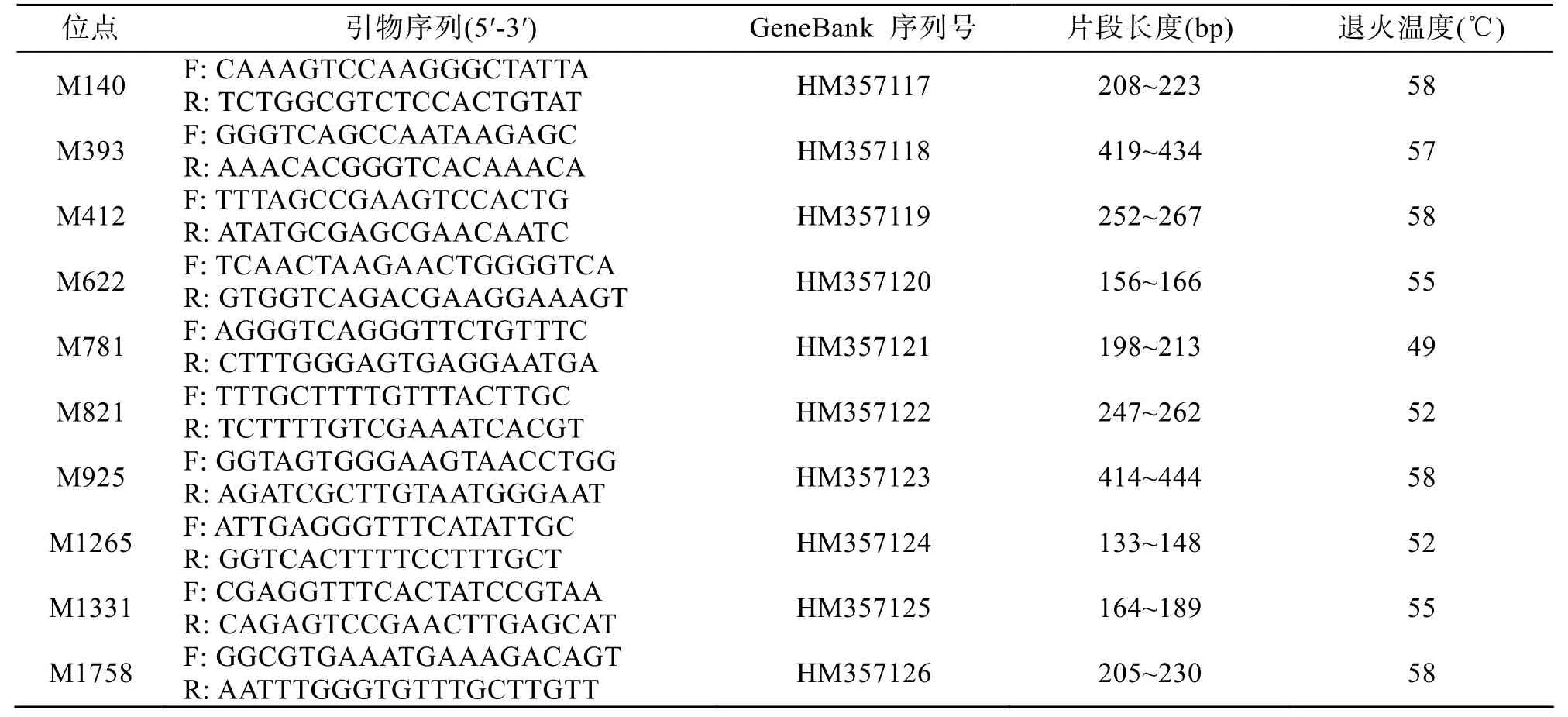
表1 10对多态性微卫星引物序列Tab.1 Sequences of ten pairs of polymorphic microsatellite primers
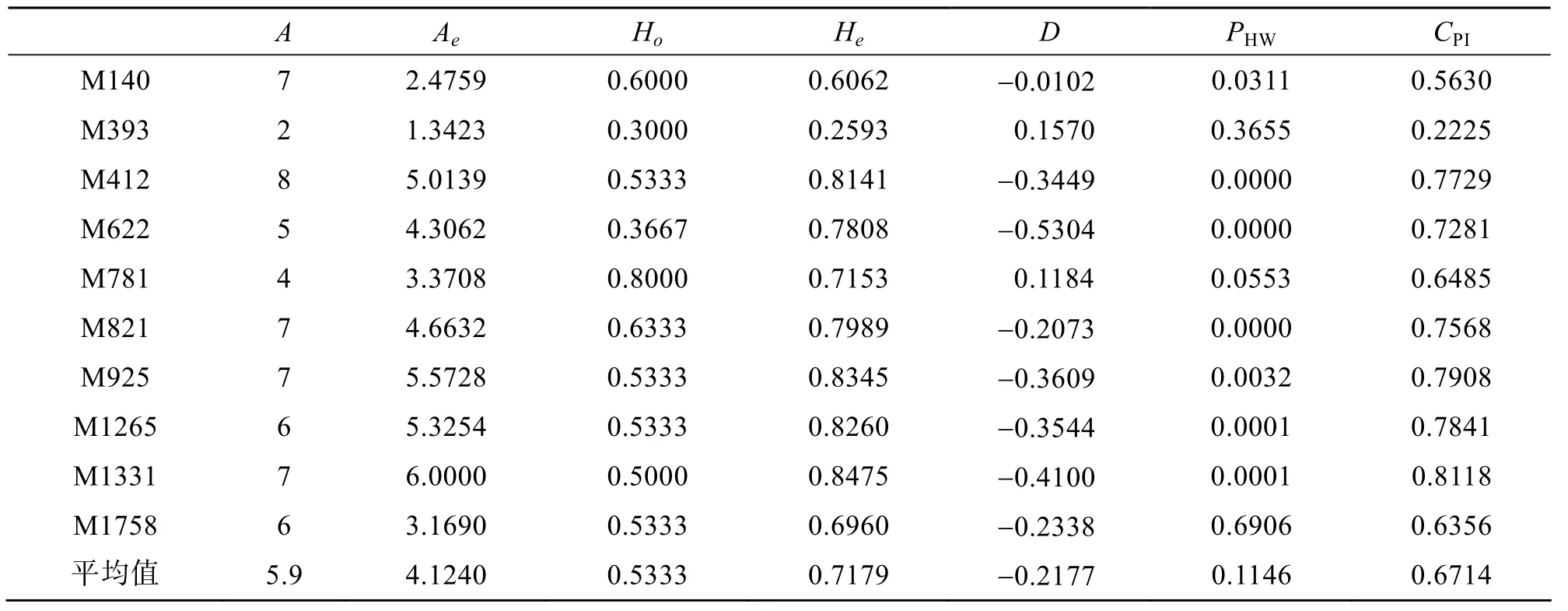
表2 10个微卫星多态性位点的数据统计Tab.2 The statistics of ten microsatellite loci
2.3 微卫星位点多态性分析
使用PopGen1.32软件对所得的微卫星多态性位点进行数据分析,由表2可知,10个位点共获得59个等位基因,其中M412位点检测出的等位基因最多(8个),M393位点的等位基因最少(2个),大部分位点为5~7个,大小在133~444 bp之间,表现出高度多态性。有效等位基因数(Ae)为1.342 3~6.000 0,平均为4.124 0; 观测杂合度(Ho)的范围为 0.300 0~0.800 0,平均为 0.533 3; 期望杂合度(He)范围为 0.259 3~0.847 5,平均为 0.717 9; 多态性信息含量(CPI)在0.222 5~0.811 8之间,平均为0.671 4。根据杂合度计算的遗传偏离指数(D),发现除了M393和M781位点为正值外,其余各位点均表现为不同程度的杂合子缺失。根据 Hardy-Weinberg平衡的卡方检验(PHW),只有 M393、M781和 M1758位点处于平衡状态(PHW>0.05),其他位点都不同程度地偏离了平衡。
3 讨论
磁珠富集法是一种简单快速的筛选微卫星的方法,通过带有链霉亲和素的磁珠亲和捕捉生物素标记的微卫星探针结合的DNA基因组片段,从而获得高度富集微卫星小插入片段的基因组文库,且获得的微卫星重复序列的比例比小克隆的比例高[10],目前国内外较多采用这种方法[11,12]。本实验在所测298个克隆中有 211个含有微卫星序列,阳性克隆率高达70.81%。姬长虹等[13]的报道为95.3%,李小宁等[14]为 88.5%,说明此法是一种高效快速的微卫星标记分离方法。但贝类多态位点的筛选效率相对较低,需要设计大量的引物才能获得为数不多的多态标记[15],本实验合成的 30对引物中仅有10对扩增出具有多态性的目的条带,曲妮妮等[16]在筛选合浦珠母贝微卫星标记的研究中49对引物只有9对具有多态性。出现这类情况的原因可能与贝类个体之间的遗传变异程度较大有关。Arias 等[17]对扇贝的 SNPs检测,发现大约 100 个碱基就出现 1 次 SNP。Sauvage等[18]对太平洋牡蛎(Crassostrea gigas) 的研究,发现在编码区平均每60 bp 出现1 次SNPs,在非编码区每40 bp 就出现1 次SNPs。这些高频率的点突变为微卫星引物的扩增筛选带来了很大困难。
在动物基因组中微卫星大约 6~10 kb就出现一次[19],其中二碱基重复类型(CA/GT,AG/TC,AT/TA)的微卫星最为常见[20,21]。本实验采用了其中的(CA)15探针,在所得的 251个微卫星位点中,含(CA/GT)n的有 204个,约占总数的 81%,其次是(AG/TC)n和(AT)n,各有24和6个,分别占总数的10%和2%。结果表明在大珠母贝中除了有大量的双碱基序列外,还有其他三碱基、四碱基和四碱基以上的微卫星序列,因此可以考虑采用其他类型的探针研究开发大珠母贝基因组微卫星,以便获得更多的微卫星分子标记。
微卫星核心序列突变率相对较高,造成了微卫星核心序列重复次数的变化,这是微卫星多态性的基础[22]。Ellegren[23]认为真核生物中微卫星重复碱基重复序列长度大多在30次重复以下,本实验所得序列重复次数在8~25的有58%,与上述分析微卫星的结论相似。关于微卫星重复数与多态性的关系,多数学者认为微卫星重复次数与多态性之间存在正相关,Valdes[24]认为重复次数低于 5的微卫星几乎检测不出多态性,一般微卫星的核心序列重复次数越高,其等位基因数也就越多,即多态性也就越高[25]。但较长的(AT)n容易形成二级结构而造成PCR效率下降,以及在电泳分离中造成较大误差,并会给测序工作带来一些麻烦[26]。本研究所得的大珠母贝微卫星序列中,除去(AT)n重复单元以外,重复次数在 5次以上的约占94%。因此理论上讲,设计的引物可以进行群体的遗传多样性研究
杂合度是度量群体变异的一个重要参数[27],包括观测杂合度和期望杂合度。本实验所获得的10个微卫星位点有 8个位点的观测杂合度低于期望杂合度,其范围相应地低于Smith等[3]报道的Ho(0.479~0.891)和He(0.872~0.972),与 Evans 等[2]报 道 的Ho(0.172~0.813)和He(0.163~0.878)比较接近,与谷龙春等[28]报道的Ho(0.167~0.833)相近,而略低于其He(0.658~0.915)。CPI值是等位基因频率和数目变化的函数,是衡量片段多态性的较好指标,能反映出某个遗传标记所含的遗传信息容量。根据 Bostein等[8]提出的衡量基因变异程度高低的多态信息含量指标,当CPI>0.5 时,该基因座为高度多态基因座;当 0.25 <CPI< 0.5 时,为中度多态基因座; 当CPI<0.25 时,则为低度多态基因座。试验中开发的10个微卫星基因座除M393为低度多态性外,其他9个均为高度多态性,表明这些微卫星位点适合大珠母贝的遗传多样性研究。结果为进一步开展大珠母贝的遗传多样性、遗传连锁图谱构建和遗传选育等研究工作奠定了一定基础。
[1]姜因萍,何毛贤.大珠母贝的研究概况[J].海洋科学,2009,33(2):92-96.
[2]Evans B S,Knauer J,Taylor J U,et al.Development and characterization of six new microsatellite markers for the silver-or gold-lipped pear oyster,Pinctadamaxima(Pteriidae) [J].Molecular Ecology Notes,2006,6(3):835-837.
[3]Smith C,Benzie J A H,Wilson K J.Isolation and characterization of eight microsatellite loci from silver-lipped pearl oysterPinctada maxima[J].Molecular Ecology Notes,2003,3 (1):125-127.
[4]Sambrook J,Fritsch E F,Maniatis T.Molecular cloning:A Labortory Manual[M].NewYork:Cold Spring Harbour Press,1989.463-468.
[5]Zane L,Bargelloni L,Patarnello T.Strategies for microsatellite isolation:a review[J].Molecular Ecology,2002,11:1-16.
[6]Jeffs P.Multiple sequence alignment with Clustal X[J].Computer Corner,1998,23(1):78-80.
[7]Yeh F C,Boyle T J B.Population genetic analysis of Co-dominant and dominant markers and quantitative traits [J].Belgian Journal of Botany,1997,129:157.
[8]Botstein D,White R L,Skolnick M,et al.Constructio of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[9]Weber J L.Informativeness of human (dC-dA)n(dG-dT)npolymorphisms[J].Genomics,1990,7(3):524-530.
[10]孙效文,贾智英,魏东旺,等.磁珠富集法与小片段克隆法筛选鲤微卫星的比较研究[J].中国水产科学,2005,12(2):126-132.
[11]Li Q,Kijima A.Identification of novel microsatellite loci in the Pacific oyster (Crassastrea gigas) by magnetic bead hybridization selection[J].Tohoku Journal of Aqricultural Research,2002,53 (1-2):25-32
[12]孙效文,鲁翠云,梁利群.磁珠富集法分离草鱼微卫星分子标记[J].水产学报,2005,29(4):482-486.
[13]姬长虹,孙效文.用磁珠富集法快速制备银鲫微卫星标记[J].大连水产学报,2007,22(6):460-464.
[14]李小宁,张殿昌,江世贵,等.合浦珠母贝微卫星DNA 标记分离与分析[J].福建水产,2009,13(5):48-54.
[15]Herbinger C M,Smith C A,Langy S.Development and characterization of novel tetra- and dinucleotide microsatellite markers for the French Polynesia black-lipped pearl oyster,Pinctada margaritifera[J].Molecular Ecology Notes,2006,6(1):107-109.
[16]曲妮妮,龚世圆,黄桂菊,等.基于 FIASCO 技术的合浦珠母贝微卫星标记分离与筛选研究[J].热带海洋学报,2010,29(3):1-8.
[17]Arias A,Freire R,Boudry P,et al.Single nucleotide polymorphism for population studies in the scallopsAequipecten opercularisandMimachlamys varia[J].Conserv Genet,2008,56:1000-1007.
[18]Sauvage C,Bieme N,Lapegue S,et al.Single Nucleotide polymorphisms and their relationship to codon usage bias in the Pacific oysterCrassostrea gigas[J].Gene,2007,406(1-2):13-22.
[19]Wang Y,Guo X.Chromosomal rearrangement in Pectinidae revealed by rRNA loci and implications for bivalve evolution[J].Biological Bulletin,2004,207(3):247-256.
[20]Chistiako D A,Helleman B,Volckaert F A M.Microsatellites and their genomic distribution,evolution,function and applications:A review with special reference to fish genetics[J].Aquaculture,2006,255(1-4):1-29.
[21]Brenner S,Elgar G,Sandford R,et al.Characterization of the pufferfish (Fugu) genome as a compact model vertebrate genome [J].Nature,1993,366:265-268.
[22]刘志毅,相建海.微卫星 DNA分子标记在海洋动物遗传分子中的应用[J].海洋科学,2001,25(6):11-13.
[23]Ellegren H.Microsatellite evolution:a battle between replication slippage and point mutation[J].Trends in Genetics,2002,18:70.
[24]Valdes A M.Allele frequencies at microsatellite loci:the stepwise mutation model revisited[J].Genetics,1993,133(3):737-749.
[25]Ma Z Q,Roder M,Sorrells.Frequencies and sequence characteristics of di,tri-,and tetra-nucleotide microsatellite in wheat[J].Genome,1996,39(1):123-130.
[26]林能锋,苏永全,丁少雄,等.赤点石斑鱼微卫星DNA筛选[J].福建农业学报,2007,22(2):115-119.
[27]陈微,张全启,于海洋,等.牙鲆微卫星标记的筛选及群体多态性分析[J].中国水产科学,2005,12(6):682-687.
[28]谷龙春,黄桂菊,喻达辉,等.大珠母贝两个野生群体遗传多样性的微卫星分析[J].渔业科学进展,2009,30(4):98-101.
