组织中全基因组DNA甲基化的液相色谱-串联质谱分析
2010-02-02张俊杰张立坚刘春安张良滔
张俊杰,张立坚,刘春安,张良滔,蔡 春
(广东医学院,广东 湛江 524023)
组织中全基因组DNA甲基化的液相色谱-串联质谱分析
张俊杰,张立坚,刘春安,张良滔,蔡 春
(广东医学院,广东 湛江 524023)
建立液相色谱-串联质谱测定组织中全基因组DNA甲基化水平的方法。采用苯酚氯仿提取组织DNA,提取的DNA用88%甲酸在140℃下裂解,DNA裂解液加入同位素胞嘧啶作内标,经N2吹干后,用甲醇溶解,以液相色谱-串联质谱检测胞嘧啶和5-甲基胞嘧啶的含量,并计算全基因组中DNA甲基化的水平。结果表明,胞嘧啶的线性范围为1~100μg·L-1,相关系数为0.997 4,相对标准偏差为0.70%~4.09%;5-甲基胞嘧啶的线性范围为1~50μg·L-1,相关系数为0.994 8,相对标准偏差为0.60%~4.81%。胞嘧啶和5-甲基胞嘧啶的检出限为1 pg,日内相对标准偏差为1.86%~4.67%,日间相对标准偏差为3.72%~4.68%,胞嘧啶和5-甲基胞嘧啶的加样回收率为86.52%~105.14%。本研究所建立的方法检测组织中DNA甲基化程度,具有专一性强、操作简便的优点,能较好的满足全基因组DNA甲基化检测的要求。
DNA甲基化;液相色谱-串联质谱(LC-MS/MS);胞嘧啶;5-甲基胞嘧啶
DNA甲基化是指在DNA甲基化转移酶介导,在碱基上结合一个甲基的化学修饰过程。高等生物中的甲基化以胞嘧啶5位甲基化为主。DNA甲基化是表观遗传学的重要组成部分,它是有核细胞特有的复制和转录后修饰,能够在不改变基因组中碱基排序的情况下影响基因转录和表达,它涉及到个体发育,细胞增殖、分化、基因印迹、X染色体失活,基因表达调控和碱基突变等多方面的生物学功能[1-4],特别对肿瘤的发生和发展具有重要的生物学意义。肿瘤中DNA甲基化模式发生改变,表现为全基因组低甲基化和某些基因启动子Cp G岛区的高甲基化。这种甲基化模式的改变与肿瘤变化的关系已成为肿瘤研究的一个热点。
DNA甲基化的准确检测对于研究DNA甲基化变化极为重要。有文献[5-7]报道,DNA经酶解后,运用LC-M S/M S技术检测5′-甲基胞嘧啶核苷和胞嘧啶核苷的含量,从而计算DNA甲基化程度。此外,也有文献[8]报道,DNA通过化学方法裂解后,采用 GC/MS方法进行5′-甲基胞嘧啶和胞嘧啶含量的检测。本工作使用化学方法将DNA裂解后,运用LC-M S/M S法测定5′-甲基胞嘧啶和胞嘧啶的含量,评价DNA的甲基化程度,并用所建立的方法分析6例结肠癌病人肿瘤组织和正常结肠组织中全基因组DNA甲基化的变化。
1 实验部分
1.1 仪器与试剂
3K15台式高速冷冻离心机:德国Sigma公司产品;UV-2100型紫外可见分光光度计:上海元析仪器有限公司产品;Aglient 1200-6430A液相色谱-串联质谱仪:美国安捷伦公司产品。
胞嘧啶(cytosine,Cyt)和5-甲基胞嘧啶(5-methylcytosine,5m Cyt):购于 Sigma公司;同位素胞嘧啶(cytosine-13C15N2):购于 Toronto research chemicals inc.。
氯仿,异戊醇,十二烷基磺酸钠(SDS),无水乙醇,甲酸,乙二胺四乙酸二钠(EDTA)和盐酸(HCl)均为分析纯;乙腈 (色谱纯)/甲酸胺和甲酸(色谱纯);实验用水为M illi-Q超纯水;三羟甲基胺基甲烷(Tris)和 Tris饱和酚为生化试剂。
1.2 储备液和工作液的配制
1.2.1 储备液的配制 准确称取10 mg Cyt和5mCyt,用甲醇溶解定容至250 m L,得40 mg·L-1Cyt和 5m Cyt标准储备液;取 10 mg Cyt13C15N2内标物,用甲醇溶解后,置于250 mL容量瓶中,定容至刻度,得 40 mg·L-1Cyt13C15N2标准储备液。所有储备液在-20℃冰箱保存,待用。
1.2.2 工作液的配制 临用时取Cyt储备液,加入甲醇 ,稀释成浓度为 100、75、50、40、25、10、7.5、5、2.5和 1μg·L-1工作液;取 5mCyt储备液 ,加入甲醇 ,稀释成浓度为 50、40、25、10、7.5、5、4、2.5、1和 0.5μg·L-1工作液;临用时取 40 mg·L-1的Cyt13C15N2标准储备液,加入甲醇,稀释成浓度为40μg·L-1的Cyt13C15N2内标工作液。
1.3 样品处理
取结肠癌病人手术后的少量肿瘤组织和肿瘤旁正常结肠粘膜组织,置于-70℃冰箱保存。
1.3.1 DNA提取 取 0.05 mg组织,加入0.12 mL蛋白酶 K(20 g·L-1)、4 mL Tris-HCl-EDTA(TE)缓冲液和0.48 m L 10%SDS,45℃水浴过夜,加入V(苯酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1进行提取,摇匀6 000×g离心 5 min,取上清,重复提取 1次;再加入V(氯仿)∶V(异戊醇)=24∶1,摇匀 6 000×g离心5 min;取上清加入1/10倍体积的3 mol·L-1醋酸钠和2.5倍的无水乙醇,12 000×g离心5 m in,倒去溶液,加入1 m L TE缓冲液溶解。用紫外可见分光光度计检测 DNA纯度,当A260nm/A280nm=1.7~1.9时,视为纯DNA。若有RNA污染加入RNase A,重复以上步骤。
1.3.2 DNA裂解 取约2.5μg DNA放入反应瓶中,用 N2吹干,然后加入0.2 mL 88%甲酸,140℃反应90 min,待其冷却至室温后,用N2吹干,加入400μL甲醇溶解,15 000×g离心4 min,取上清液,LC/M S分析。
1.4 仪器条件
1.4.1 液相色谱条件 BEH H IL IC色谱柱(1.7μm×2.1 m×100 mm,Waters公司产品);流动相:A为0.1%甲酸铵,B为乙腈;流速:0.4 m L·min-1;柱温为室温,进样量5μL;洗脱程序:0~1 min A为10%,1~2 min A从10%变为 50%,2~2.5 min A为 50%,2.5~8 m in A从50%变为10%。
1.4.2 质谱条件 电喷雾正离子电离模式,离子源温度300℃;喷雾电压3.5 kV,喷雾气流量9 L·min-1;入口电压:Cyt和5m Cyt为114 V,Cyt13C15N为115 V。采用多反应监测模式检测;碰撞能量:Cyt和5mCyt为20 V,Cyt13C15N为16 V。Cyt定量离子m/z112/95,辅助定量离子m/z69;5m Cyt定量离子m/z126/109,辅助定量离子m/z83;Cyt13C15N定量离子m/z115/97,辅助定量离子m/z115/70。
1.5 数据处理
采用内标法定量,以w(5mCyt)/[w(5mCyt)+w(Cyt)]×100%的方式表示DNA甲基化程度,Excell进行统计分析。
2 结果
2.1 5mCyt和Cyt质谱图
分别加入5μL 1 mg·L-1Cyt、40μg·L-1Cyt13C15N2和 1 mg·L-15mCyt标准溶液,经LC-M S/M S分析,其质谱图示于图1。图1中,m/z112([M+H]+)、m/z115([M+H]+)和m/z126([M+H]+)分别为 Cyt、Cyt13C15N2和5m Cyt分子离子峰;m/z112/95和m/z126/109分别为Cyt和5mCyt的主要碎片离子,因此选用m/z112/95和126/109作为Cyt和5m Cyt的定量检测。
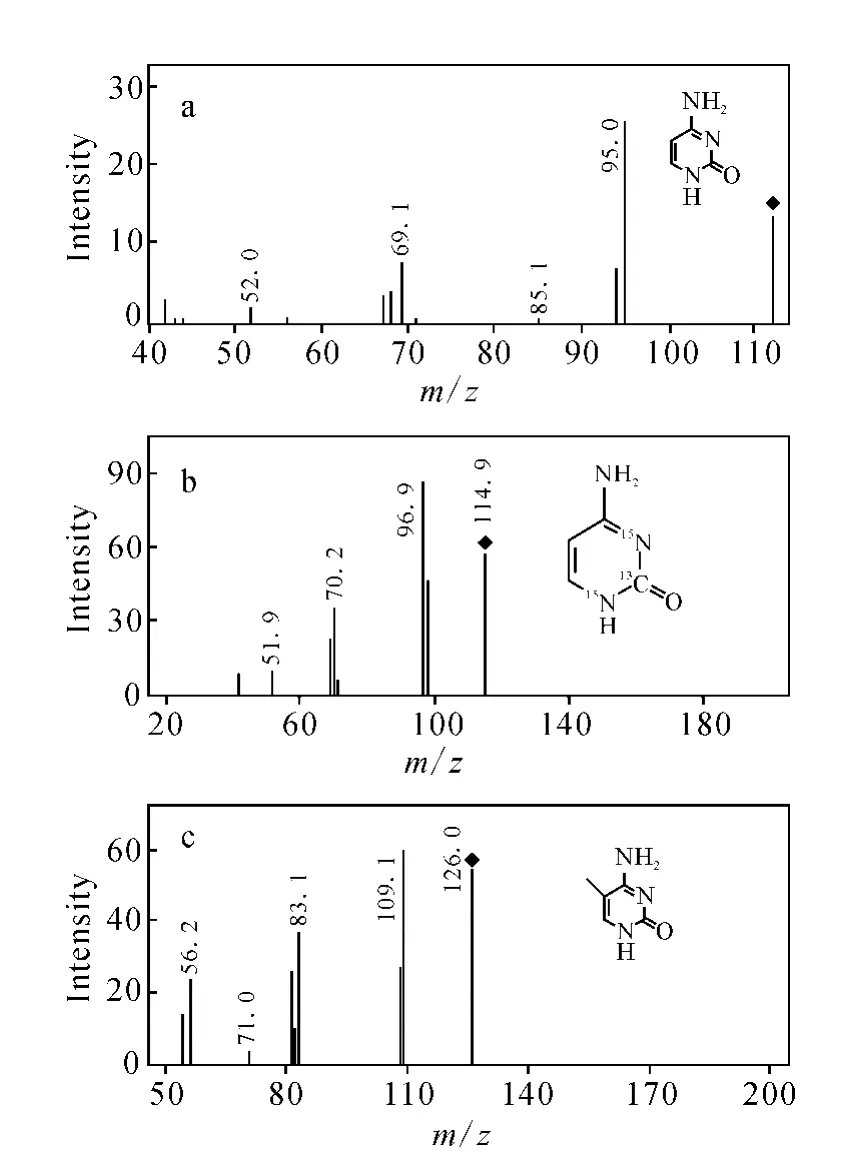
图1 Cyt(a)、Cyt13 C15 N2(b)和5mCyt(c)的子离子图Fig.1 Product ion scan of Cyt(a),Cyt13 C15 N(b)and 5mCyt(c)
2.2 Cyt和5mCyt色谱条件
实验中所需分离的嘧啶为碱性物质,因此采用适合于低p H值流动相的 H IL IC柱,对甲酸铵-乙腈、乙腈-甲酸溶液等多种流动相进行实验。结果表明,以乙腈-0.1%甲酸铵溶液为流动相,流速0.4 m L·min-1;洗脱条件:0~1 min A为10%,1~2 m in A从 10%变为 50%,2~2.5 min A为50%,2.5~8 min A从50%变为10%时得到的峰形最好,示于图2。

图2 标准品Cyt(a),Cyt13 C15 N2(b)和5mCyt(c)的M RM图Fig.2 M RM of Cyt(a),Cyt13 C15 N2(b)and 5mCyt(c)in standard solution
2.3 线性范围及精密度实验
混合标准液包括5mCyt和Cyt,其中质量浓度系列为 0.5、1、2.5、4、5、7.5、10、25、40 和 50 μg·L-1,Cyt质量浓度系列为 1、2.5、4、5、7.5、10、25、40、50、75 和 100 μg·L-1,加 入40μg·L-1Cyt13C15N2作内标,进样 5次。分别以Cyt和5m Cyt质量浓度为横坐标,色谱峰面积为纵坐标绘制标准曲线,并以信噪比为3确定样品的最低检测限。结果表明,Cyt和5m Cyt的检出限为 1 pg,Cyt的线性范围为 1~100 μg·L-1,标准曲线为y=1.258 0x-0.625 5,相关系数R2为0.997 4,相对标准偏差 RSD为0.70%~4.09%;5mCyt的线性范围为1~50 μg·L-1,标准曲线为y=1 216.621 2x-593.699 8,相关系数R2为0.994 8,相对标准偏差RSD为0.60%~4.81%。
2.4 重现性实验
取同一样品,按 1.3方法制备 3份,测定Cyt和5mCyt两个色谱峰相对丰度的RSD列于表1。
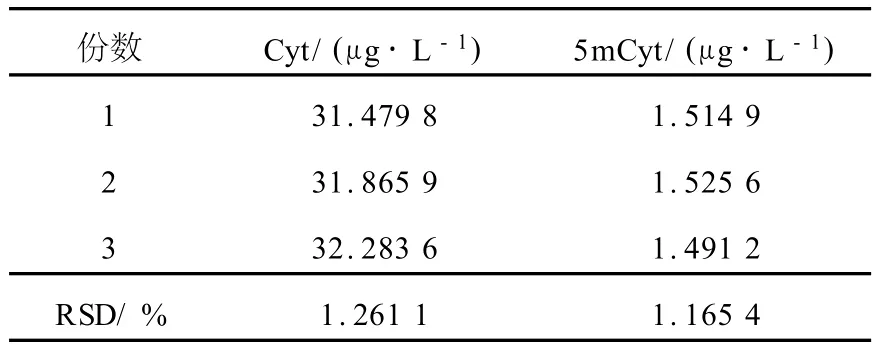
表1 定量测定结肠癌组织中Cyt和5m Cyt的重现性Table 1 Reproducibility of Cyt and 5mCyt in colon cancer
2.5 稳定性实验
取同一DNA样品试液,在-20℃分别保存2、4、6、8、10 h 后 ,测定 Cyt和 5mCyt两个定量质谱峰相对丰度的 RSD分别为1.86%和4.67%。
取同一DNA样品试液,在-20℃分别保存1、2、3、4、5 d 后 ,测定 Cyt和 5mCyt两个定量质谱峰相对丰度的RSD分别为3.72%和4.68%。
2.6 回收率实验
取同一结肠癌病人的肿瘤DNA溶液,经88%甲酸在140℃下裂解后,分别加入400μL 50、10、1和 0μg·L-1的 Cyt和 5m Cyt,N2吹干,加入甲醇溶解,15 000 r·min-1离心5 min,取上清液,用于LC/M S分析。测定Cyt回收率分别为87.38%、105.14%和96.18%;5m Cyt回收率分别为89.58%、86.52%和94.52%,其结果列于表2。
2.7 结肠癌组织样品分析
利用上述方法检测了6例结肠癌组织中DNA甲基化水平,结肠癌患者正常组织和肿瘤组织甲基化分析结果列于表3。实验结果表明,癌组织的甲基化水平低于相应的正常组织,这与文献[9]报道一致,说明本方法可以很好地用于全基因组DNA甲基化水平的检测。

表2 样品添加回收率实验(n=3)Table 2 Recoveries of Cyt and 5m Cyt from spiked sam ples(n=3)
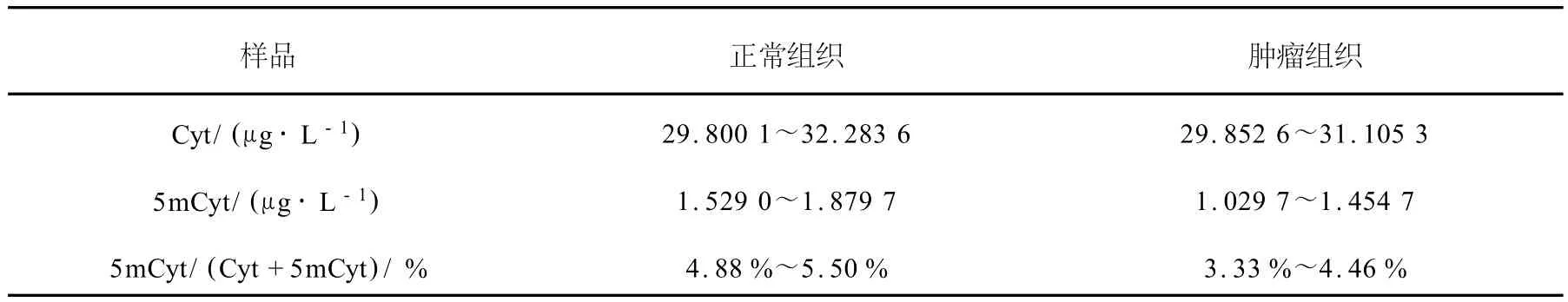
表3 结肠癌和正常组织样品甲基化分析结果Table 3 Results of contents in colon cancer tissuesand normal tissues
3 讨论
目前检测基因组DNA甲基化主要有SssI甲基转移酶法和测序法,其中SssI甲基转移酶法由于SAM和SssI甲基转移酶性质不稳定,易造成实验结果误差比较大,必须设立自身对照来标化实验数据;测序法需要大量的克隆测序,较为繁琐[10]。最近报道[11-13],LC-M S/M S测定嘧啶核苷,此方法需要核酸酶、磷酸二酯酶和碱性磷酸酶等酶水解DNA,由于酶活性的影响可能存在由于酶解不完全带来的误差。另有报道[8]将DNA用化学方法裂解为嘧啶后,通过衍生用GC/M S测定甲基化胞嘧啶和胞嘧啶的含量,该方法中嘧啶的衍生增加了操作的难度。本方法通过化学裂解DNA,既排除酶解法可能带来的误差,又不需要衍生,大大简化了操作。
胞嘧啶和5-甲基胞嘧啶在C 18柱上没有保留,改变洗脱溶剂也无法解决,改用适合碱性化合物分析的 H IL IC柱后,胞嘧啶和5-甲基嘧啶实现了分离,并发现 H IL IC柱比C 18柱的灵敏度高,这可能与 H IL IC柱使用高比例有机相有关。
用乙腈和0.1%甲酸作为流动相时,胞嘧啶和5-甲基胞嘧啶在柱上的保留时间基本相同;当用p H 4.5~6的乙酸-乙酸铵缓冲液作流动相时,色谱峰发生分岔的现象;而用p H 3.6的乙酸-乙酸铵缓冲液作为流动相,色谱峰没有分岔,但是重现性不好,提示p H值和缓冲体系对胞嘧啶和5-甲基-胞嘧啶的分析影响比较大;当改用乙腈和0.1%甲酸铵水溶液作流动相时,胞嘧啶和5-甲基胞嘧啶基本分离,且重现性较好。
[1]BOUM IL R M,LEEJ T.Forty yearsof decoding the silence in X-chromosome inactivation[J].Hum Mol Genet,2001,10(20):2 225-2 232.
[2]L IM H N,VAN O A.A multistep epigenetic switch enables the stable inheritance of DNA methylation states[J]. Nat Genet,2007,39(2):269-275.
[3]FLANAGAN J M,POPEND IKYTE V,POZDNIA KOVA ITE N,et al.Intra-and interindividual epigenetic variation in human germ cells[J].Am J Hum Genet,2006,79(1):67-84.
[4]SHA K.A mechanistic view of genomic imp rinting[J].Annu Rev Genomics Hum Genet,2008,9:197-216.
[5]SONG L,JAM ES SR,KAZIM L,et al.Specific method for the determination of genomic DNA methylation by liquid chromatography-electrosp ray ionization tandem mass spectrometry[J].Anal Chem,2005,77(2):504-510.
[6]FR ISO S,CHO ISW,DOLN IKOW SK IG G,et al.A method to assess genomic DNA methylation using high-performance liquid chromatography/electrosp ray ionization mass spectrometry[J].Anal Chem,2002,74(17):4 526-4 531.
[7]KOK RM,SM ITH D E,BARTO R,et al.Global DNA methylation measured by liquid chromatography-tandem mass spectrometry:analytical technique,reference values and determinants in healthy subjects[J].Clin Chem Lab Med,2007,45(7):903-911.
[8]ROSSELLA F,POLL EDRIE,BOLLA T IV,et al.Development and validation of a gas chromatography/mass spectrometry method for the assessment of genomic DNA methylation[J].Rapid Commun Mass Spectrom,2009,23(17):2 637-2 646.
[9]HERNANDEZ-BLAZQUEZ FJ,HABIB M,DUMOLLARD J M,et al.Evaluation of global DNA hypomethylation in human colon cancer tissues by immunohistochemistry and image analysis[J].Gut,2000,47(5):689-693.
[10]李海飞.DNA甲基化的分析与状态检测[J].医学综述,2004,10(3):131-133.
[11]QU INL IVAN EP,3rd GJF.DNA methylation determination by liquid chromatography-tandem mass spectrometry using novel biosynthetic[U-15N]deoxycytidine and[U-15N]methyldeoxycytidine internal standards[J].Nucleic Acids Res,2008,36(18):e119.
[12]L IU Z,L IU S,XIE Z,et al.Characterization of in vitro and in vivo hypomethylating effects of decitabine in acute myeloid leukemia by a rapid,specific and sensitive LC-MS/MS method[J].Nucleic Acids Res,2007,35(5):e31.
[13]MA H,ZHANGW,SONG R,et al.Characterization of methylated and unmethlyated Cp GODNs by electrosp ray ionization tandem mass spectrometry[J].Eur J Mass Spectrom(Chichester,Eng),2005,11(6):581-589.
Analysis of Global DNA Methylation in Tissue by Liquid Chromatography-Tandem Mass Spectrometry
ZHANG Jun-jie,ZHANG Li-jian,L IU Chun-an,ZHANG Liang-tao,CA IChun
(Guangdong M ed ical College,Zhanjiang 524023,China)
Global DNA methylation in tissue was determined by liquid chromatography-tandem mass spectrometry(LC-M S/M S).DNA w as extracted by phenol-chlo roform,hydrolyzed using 88%formic acid at 140 ℃,spiked with cytosine-2,4-13C2,15N2as internal standard,reconstituted in methanol and analyzed by LC-M S/M Swith multip le reaction monitoring mode,to reflect the global DNA methylation level of the tissue.Results show that the limit of quantification is 1μg·L-1for both Cytosine(Cyt)and 5-methylcytosine(5m Cyt),and the linear rangesof calibration curve are 1—50μg·L-1and 1—100μg·L-1for 5mCyt and Cyt,respectively,with correlation coefficient higher than 0.99.The relative standard deviations(RSDs)are 0.70%—4.09%and 0.60%—4.81%for Cyt and 5mCyt,respectively.The intra-day precision exp ressed as RSD ranges from 1.86%to 4.67%,while the inter-day values f rom 3.72%to 4.68%.The recovery ratio of method varies from 86.52%to 105.14%.The method is simple,and can be used for detection of Cyt and 5mCyt,thus enabling the evaluation of global DNA methylation.
DNA methylation;liquid chromatography-tandem mass spectrum(LC-MS/M S);cytosine;5-methylcytosine
O 657.63
A
1004-2997(2010)06-0326-05
2010-06-30;
2010-09-29
张俊杰(1985~),男(汉族),广东东莞人,硕士研究生,药物分析与仪器分析专业。E-mail:zhjunjie357753@163.com
蔡 春(1961~),男(汉族),广东湛江人,教授,从事生物医学分析研究。E-mail:caichun@gdmc.edu.cn
